糜子GRF转录因子全基因组鉴定及在茎分生组织中的表达特征
韦恒,刘天鹏,何继红,董孔军,任瑞玉,张磊,李亚伟,郝子义,杨天育,
研究报告
糜子GRF转录因子全基因组鉴定及在茎分生组织中的表达特征
韦恒1,刘天鹏2,何继红2,董孔军2,任瑞玉2,张磊2,李亚伟2,郝子义1,杨天育1,2
1. 甘肃农业大学生命科学技术学院,兰州 730070 2. 甘肃省农业科学院作物研究所,兰州 730070
为了解糜子(L.) GRF (growth-regulating factor)基因家族成员全基因组信息及其在营养生长阶段分生组织中的表达特征,本研究通过生物信息学和转录组测序相结合的方法,分析了糜子GRF基因家族成员的染色体分布、基因结构、系统进化关系、顺式作用元件及其在营养器官茎分生组织中的表达特征。结果表明:糜子GRF基因家族包含21个成员,家族成员含有1~4个内含子和2~5个外显子,编码蛋白长度为224~618个氨基酸,等电点为4.93~9.69;基因不均等分布于12条染色体上,除定位于细胞核和叶绿体外,其余均定位于细胞核。系统进化分析显示,糜子21个基因分为4个亚族(A、B、C和D)。顺式作用元件分析表明,在糜子基因上游2000 bp序列中,普遍存在数目不等、种类不同的参与光响应、激素响应、干旱诱导、低温和其他环境胁迫响应的顺式作用元件。对糜子高秆品种陇糜12号和矮秆品种张778拔节期节间和节部分生组织分别取样进行转录组测序及qRT-PCR分析,结果发现:、在矮秆品种张778中表达量显著高于高秆品种陇糜12号,而、和的表达特征与之相反;和在张778节间分生组织中的表达量显著高于陇糜12号,其余基因不表达或差异表达不显著,表明、、、、、和等7个基因与糜子株高的形成相关。
糜子;基因;转录组;株高
植物转录因子(transcription factors,TFs)是一种能识别并结合转录起始位点的上游启动子序列,可以特异地与相应DNA顺式作用元件结合并调控相关基因表达。转录因子家族基因结构复杂,既有单个家族基因,也有多个家族基因的参与[1,2],对靶基因的调节作用可发生在植物生长发育的各个阶段。
生长调节因子(growth-regulating factors,GRFs)广泛存在于植物基因组中,是一类重要的转录因子,参与调控植物生长和发育过程,对植物形态建成和适应环境具有重要调控作用。GRF的N-末端含有QLQ(Gln、Leu、Gln)和WRC(Trp、Arg、Cys)两个保守结构域,构成GRF蛋白家族[3],GRF蛋白的C端区域在长度和氨基酸序列上存在差异,但仍具有与转录因子相似的共同特征[4]。QLQ在所有真核生物中均有发现,具有介导蛋白-蛋白互作的功能,与GIF(GRF interacting factors)蛋白中的SNH结构域结合,促使GRF与GIF相互作用形成转录激活因子[5],miR396可调节及基因的表达。WRC是植物特有的一个功能域,包含一个功能性的核定位信号和用于DNA结合的C3H型锌指结构,可与下游基因的启动子区域相互作用从而调节其表达[6]。
GRFs作为植物中特有的转录因子,参与调控植物生长发育过程。GRFs对植物叶、根、茎、花、种子初级生长的影响,主要通过对细胞的增生或膨胀来实现[7,8];此外,GRFs在碳-氮代谢以及激素和非生物逆境应答中也起关键作用[9]。基因最初在水稻()中被鉴定,该基因对水稻茎节伸长起作用,经由赤霉素(gibberellin,GA)调控作用而增强表达[10]。迄今为止,对植物GRF全基因组的鉴定分析已有很多报道,其中在水稻中鉴定出12个成员[11];拟南芥()中发现9个成员[12],大多数基因参与拟南芥的生长发育,例如和的过表达会导致叶片和子叶增大,表明AtGRF蛋白参与调控叶片及子叶的细胞扩增[13]。在玉米(L.)中发现21个成员[14],基因对穗的生长和发育起关键作用[15],其中和的过表达推迟茎秆发育,但加速花序的生长。大豆()中有22个成员,多数基因在其生长发育过程中表达丰度较高,遮光处理后大豆叶片大多表达下调,其中在茎尖分生组织中表达量最高[16]。烟草(L.)中有25个成员,其中大部分主要在萌发种子中转录表达,而在茎组织中的表达量最高[17]。另外,在柳枝稷(L.)[18]、甜瓜(L.)[19]、桃[(L.) Batsch.][20]、野草莓(L.)[21]和地菍(Lour.)[22]等植物生长发育过程中,基因均普遍表达,并且在多个生物过程中起着重要作用。
糜子(L)是禾本科黍属一年生草本作物,起源于我国黄河流域,是种植历史悠久的谷类作物之一[23]。与其他大多数谷物比较,糜子耐盐、耐高温、抗旱、水分利用效率高,是一种拥有广谱抗性及抵抗非生物胁迫较强的作物[24];而且糜子生育期短,种植成本低,因此在我国北方旱作农业中占有重要地位[25]。倒伏是糜子生产中普遍存在的现象,严重影响其产量形成和籽粒品质,因此挖掘其矮秆基因、创制矮化种质、培育矮秆品种十分必要。植物茎杆的生长发育与基因的表达密切相关,但目前在糜子中尚未见报道,仅有SAMS[26]、MYB[27]、YABBY[28]、bZIP[29]和NAC[30]等基因家族被鉴定与分析。本文采用生物信息学方法并结合转录组测序分析,对糜子GRF基因家族进行了系统鉴定和茎分生组织表达特征分析,旨在为深入研究GRF基因家族在糜子株高形态建成中的功能和分子机制提供科学依据。
1 材料与方法
1.1 数据来源
以拟南芥及水稻基因为参考,拟南芥蛋白数据来自tair网站(https://www.arabidopsis.org/),水稻蛋白数据来自RGAP网站(http://rice.plantbiology. msu.edu/),糜子全基因组基因注释数据来自国家基因组科学数据中心(BIG搜索-cncb.ac.cn)。
1.2 糜子GRF基因家族鉴定
首先通过糜子、水稻、拟南芥全基因组序列构建蛋白序列文库,然后以拟南芥9个GRF蛋白(AT2G45480、AT4G24150、AT5G53660、AT2G22840、AT4G37740、AT3G52910、AT2G36400、AT2G06200和AT3G13960)和水稻12个GRF蛋白(OSGRF1、OSGRF2、OSGRF3、OSGRF4、OSGRF5、OSGRF6、OSGRF7、OSGRF8、OSGRF9、OSGRF10、OSGRF11和OSGRF12)为基础序列,在构建的蛋白序列文库中进行本地Blast搜索(-value<1e–10),得到糜子GRF基因家族候选基因的初筛结果;再利用HMM模型对糜子GRF基因家族进行鉴定,利用PFam网站(http://pfam.sanger.ac.uk/)查询并下载GRF蛋白WRC(PF08879)与QLQ(PF08880)两个特征结构域的隐马尔可夫模型,基于HMMER软件在糜子全基因组范围内鉴定GRF基因家族成员[31];最后将两种方法的鉴定结果进行比较并剔除重复项,利用CD-Search工具(https://www.ncbi.nlm.nih.gov)对蛋白结构域进行验证并保留有效值,从而得到糜子GRF基因家族。最后,利用在线软件ExpasyProtParam (https://web.expasy.org/protparam/)对糜子GRF蛋白的疏水性、氨基酸数、分子量等理化性质进行分析,同时通过PlantmPLoc网站(http://www.csbio.sjtu. edu.cn/cgi-bin/PlantmPLoc.cgi)查询糜子GRF基因家族成员的亚细胞定位。
1.3 糜子GRF基因家族系统进化树的构建
利用MAFFT网站(https://www.ebi.ac.uk/Tools/ msa/mafft/)将糜子、拟南芥、水稻蛋白序列进行多序列比对,将得到的数据采用MEGA7.0软件中的NJ法绘制进化树,设Bootstrap值为1000,处理缺失数据,选用Partialdeletion、SiteCoverageCutoff(%)为50,其余参数使用默认值[32],得到GRF基因家族系统进化树。
1.4 糜子GRF基因家族染色体分布
根据鉴定出的基因蛋白名称在NCBI (https://www.ncbi.nlm.nih.gov/gene)中确定其各家族成员所在的染色体位置及染色体长度。根据糜子基因组gff3注释文件提取基因的染色体位置;通过上述数据在MG2C网站(http://mg2c.iask.in/mg2c_ v2.1/)上对糜子GRF基因家族成员的染色体分布进行分析。
1.5 糜子GRF基因家族保守基序及基因结构分析
通过MEME在线服务器(https://meme-suite.org/ meme/tools/meme)对糜子GRF基因家族成员进行基序分析,预测基序数目设置为15,将生成的文件通过TBtools软件进行可视化处理。另外依据鉴定出的糜子基因序列在其GFF3文件中进行处理筛选,将结果通过在线软件GSDS(http://gsds.gao-lab.org/)完成基因结构分析,并利用TBtools软件进行可视化处理。
1.6 糜子GRF基因家族顺式作用元件分析
通过软件Tbtools获取糜子基因CDS序列上游2000 bp的启动子区域序列,将结果导入PlantCARE数据库(http://bioinformatics.psb.ugent.be/ webtools/plantcare/html/),鉴定糜子GRF基因家族顺式作用元件,通过TBtools进行可视化处理。
1.7 糜子GRF基因家族在其株高形态建成中的转录组及qRT-PCR分析
以甘肃省农业科学院作物研究所提供的陇糜12号[33]和张778[34]为材料,于2022年4月在甘肃省白银市会宁县试验地种植,小区面积8 m2,行长5 m,行宽40 cm,株距8 cm,5叶期定苗;2022年7月取拔节期陇糜12号和张778植株,分别取节间和节部分生组织3个生物学重复样,依托北京诺禾致源科技股份有限公司Illumina技术测序平台进行转录组测序,采用FPKM (Fragments Per Kilobase of transcript per Million fragments mapped)作为衡量转录本或基因表达水平的指标,利用TBtools软件分析GRF基因家族在茎分生组织的表达特征,并利用SPSS软件中成对样本检验方法对fpkm值进行显著性分析。
糜子成熟期,取陇糜12号和张778各10株对株高、节间长度、穗长进行室内考种,计算平均值,利用GraphPad Prism绘图,并利用SPSS软件的成对样本检验方法进行显著性分析。
糜子拔节期,对陇糜12号和张778主茎节间和节部分生组织再取3个生物学重复样,参照TRizol RNA试剂盒方法提取RNA,根据PrimerScript RT Reagent Kit (宝生物工程(大连)有限公司)试剂盒提供的方法进行反转录获得cDNA,进行qRT-PCR分析。其中,用Primer3.0设计家族基因qRT-PCR引物,以基因作为内参基因,引物序列见表1。qRT-PCR扩增体系:2×UltraSYBR Mixture 10.0 μL,上、下游引物(10 μmol/L)各1.0 μL,cDNA 1.0 μL,ddH2O 7.0 μL。扩增程序:94℃预变性1 min,94℃变性15 s,56℃退火15 s,65℃延伸10 s,40次循环,重复3次,最后采用2-∆∆CT法进行定量数据分析[35],并用SPSS软件中成对样本检验方法进行显著性分析。
2 结果与分析
2.1 糜子GRF基因家族成员、系统进化关系及染色体分布
采用blastp、hummer和CDD方法进行GRF基因家族成员鉴定,结果表明糜子GRF基因家族共有21个成员,依据基因ID号命名为(表2)。对糜子GRF基因家族成员的氨基酸数量、分子量(MW)和等电点(pI)等理化特性进行分析,发现该家族蛋白含224()~ 618()个氨基酸,分子量为23,148.19 Da()~64,962.13 Da(),等电点为4.93()~9.69(),17个PmGRF蛋白等电点超过7.0,表明糜子大多数GRF蛋白为碱性蛋白,不稳定指数在43.53~66.46之间,脂肪族氨基酸指数阈为47.23~70.34,亲水性平均系数–0.866 ~ –0.228之间(表2)。糜子GRF基因家族成员几乎都定位于细胞核,但的亚细胞定位出现在叶绿体上;~分布于染色体1、2、4、6、7、11、12、13、14、15、16和17,表现出较复杂的理化性质和多样分布特征。

表1 qRT-PCR的引物序列

表2 糜子GRF基因家族理化性质及亚细胞定位信息
利用MEGA7.0软件对鉴定出的21个糜子GRF蛋白序列与通过tair网站得到的9个拟南芥GRF蛋白序列以及通过RGAP网站得到的12个水稻GRF蛋白序列进行系统进化树构建(图1),并根据拟南芥GRF基因家族和水稻GRF基因家族的分类方法,将糜子21个GRF基因家族成员分为A、B、C和D共4个亚族,其中每个亚族分别有4、1、6和10个成员,A亚族包括、、和;B亚族中仅有;C亚族包括、、、、和;D亚族包括、、、、、、、、和。
染色体定位结果显示(图2),糜子21个GRF基因家族成员在12条染色体上不均匀分布,每条染色体上分布1~3个基因,第4、第6和第12染色体上均有3个基因,第1、第7和第16都均有2个基因,其余的6条染色体上只有1个基因。此外,糜子基因在染色体上的分布位置比较固定,大多数基因都分布在染色体的端部。
2.2 糜子GRF基因家族蛋白基序、基因结构及顺式作用元件
基因蛋白基序分析结果显示,糜子GRF基因家族检测出15个保守结构域,其中motif1和motif2分别以WRC和QLQ结构域为特征,存在于所有PmGRF蛋白中(图3)。在4个亚族中,A亚族的4个成员含有3种共有的蛋白基序类型,为motif1、motif2和motif11;B亚族的1个成员含有3种蛋白基序类型,为motif1、motif2和motif10;C亚族的6个成员共含有11种蛋白基序类型,分别为motif1、motif2、motif4、motif6、motif7、motif8、motif9、motif10、motif11、motif12和motif13,每个成员包含5~9种蛋白基序,其中PmGRF4和PmGRF1蛋白基序组成相似,PmGRF5和PmGRF2、PmGRF16和PmGRF21蛋白基序组成相同;D亚族的10个成员共含有10种蛋白基序类型,分别为motif1、motif2、motif3、motif4、motif5、motif8、motif9、motif10、motif14和motif15,每个成员包含5~8种蛋白基序,其中PmGRF15和PmGRF7、PmGRF10和PmGRF19、PmGRF11和PmGRF20以及PmGRF8、PmGRF14、PmGRF17和PmGRF18蛋白基序组成相同。
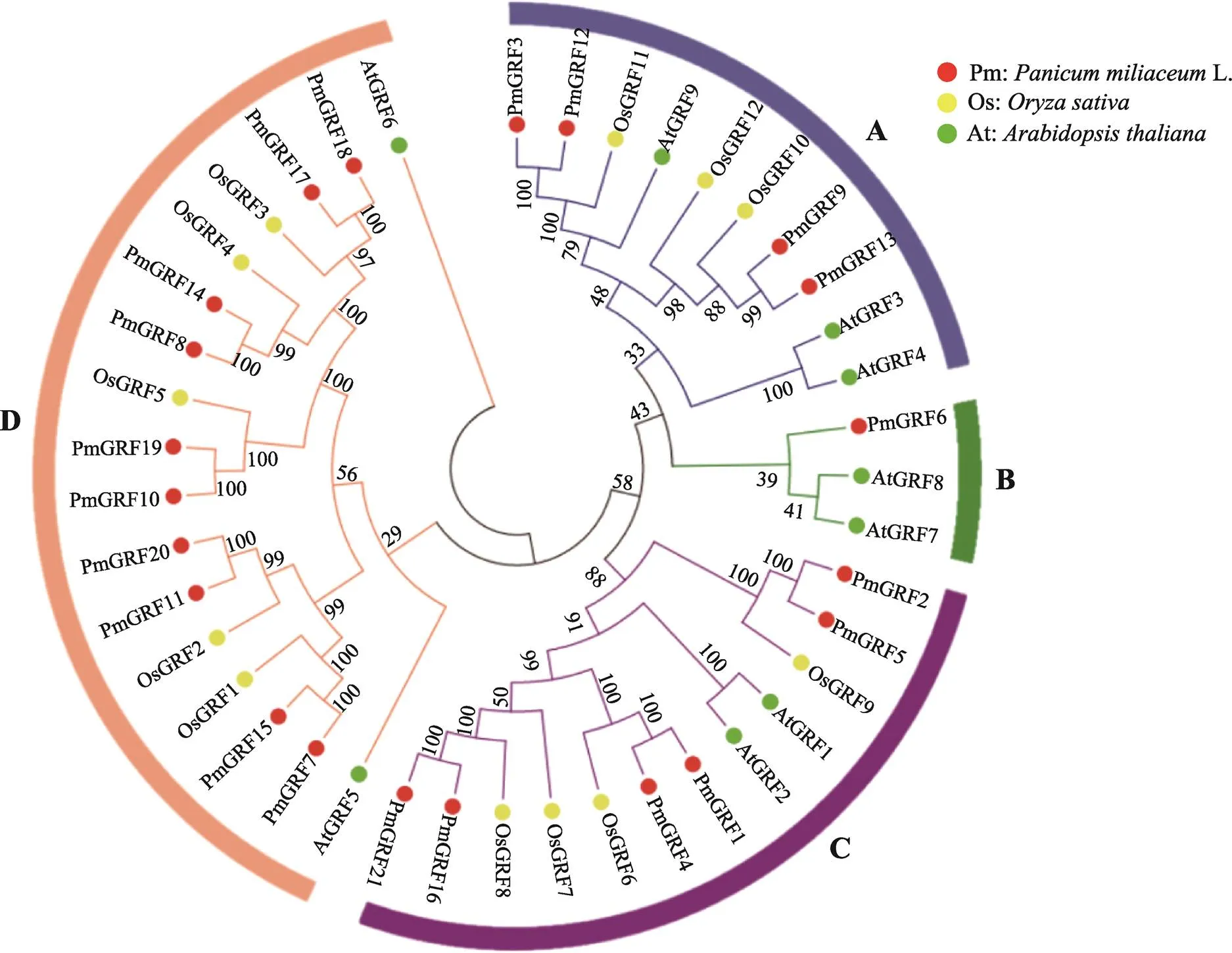
图1 糜子GRF基因家族系统进化树
不同物种的基因用不同颜色的圆点表示,其中红色圆点表示糜子,黄色圆点表示水稻,绿色圆点表示拟南芥;A、B、C和D表示不同亚族,用不同颜色的弧线表示,其中蓝色弧线表示A亚族,绿色弧线表示B亚族,紫色弧线表示C亚族,橙色弧线表示D亚族。
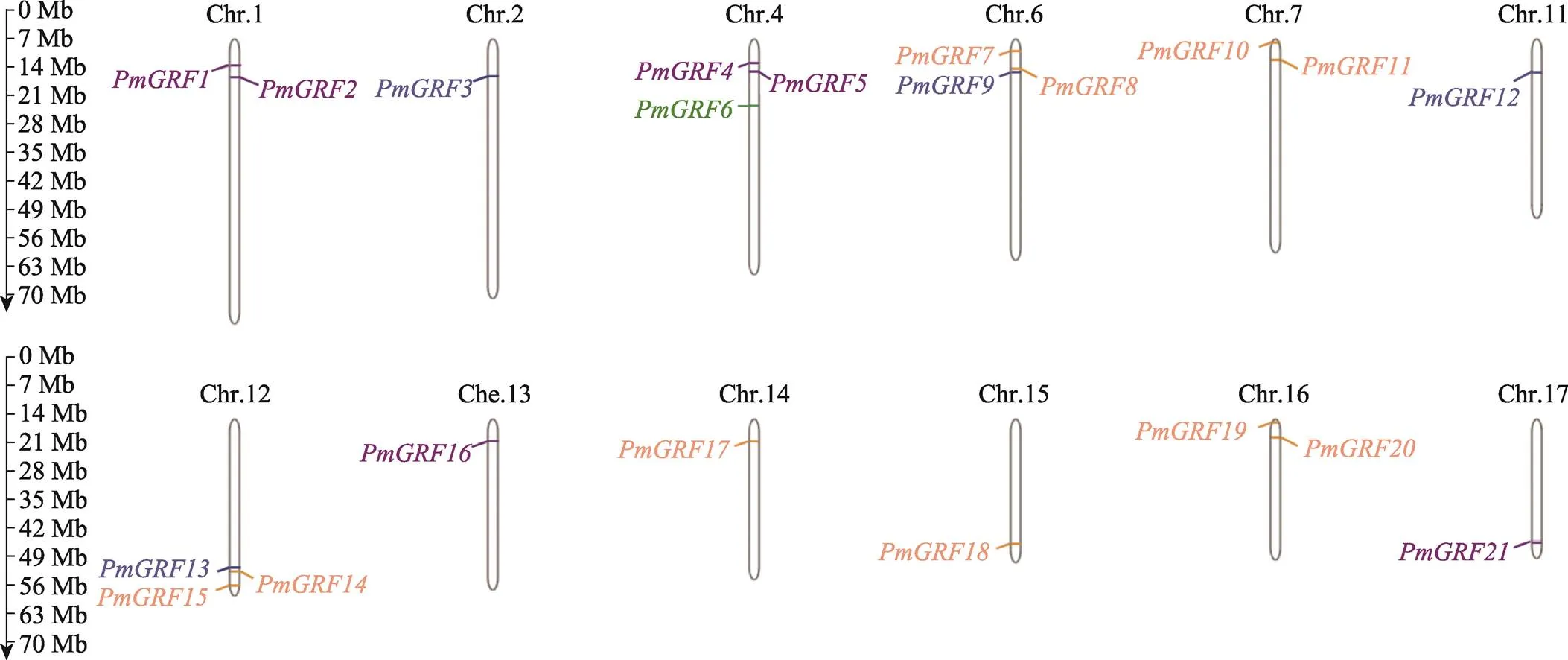
图2 糜子GRF基因染色体分布
不同亚族成员用不同的颜色表示:蓝色表示A亚族,绿色表示B亚族,紫色表示C亚族,橙色表示D亚族。
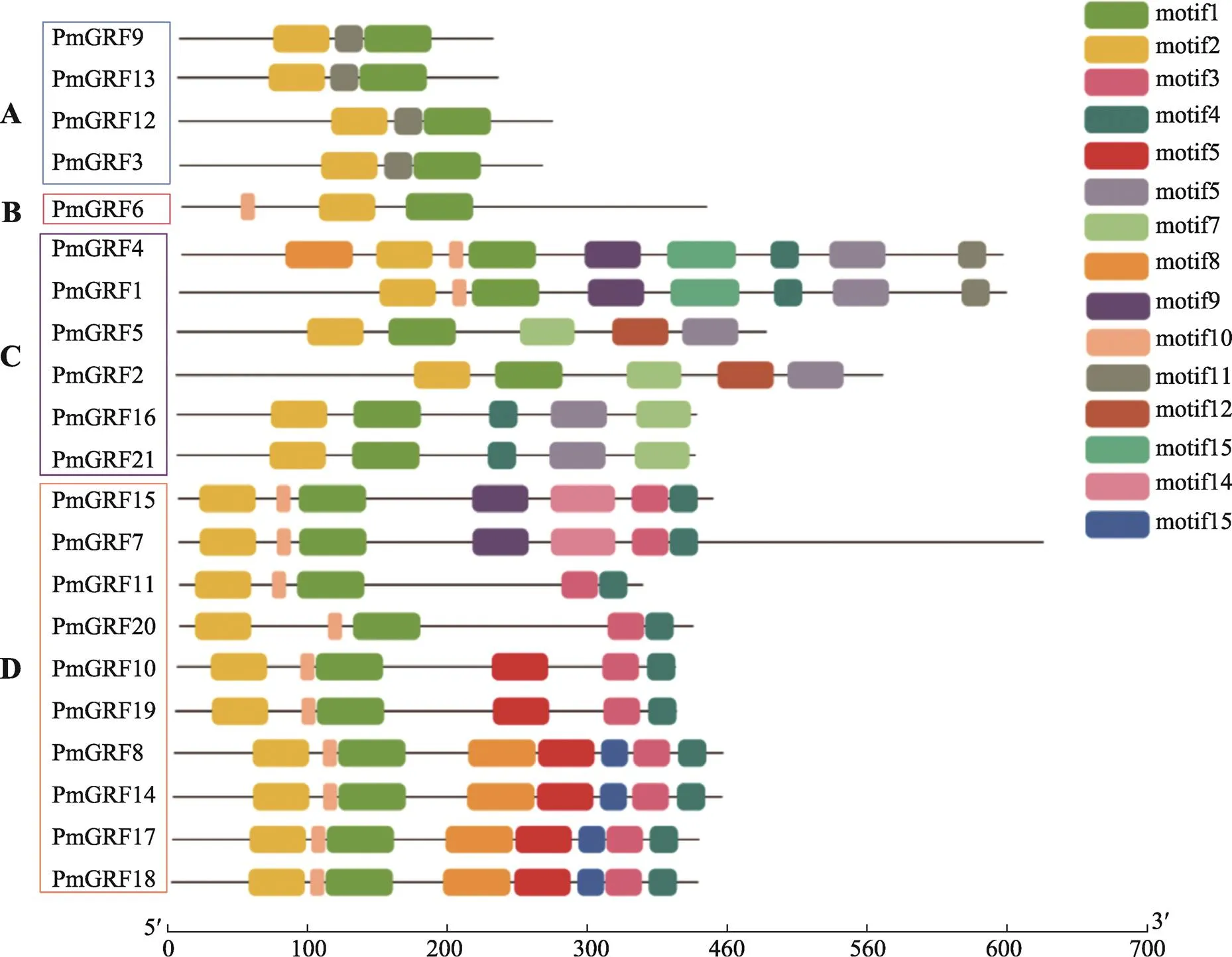
图3 糜子GRF蛋白基序组成
A、B、C和D表示4个亚族。
基因结构分析结果显示,在糜子GRF基因家族中,、、和等4个基因外显子数目最多(5);外显子数目最少(2),且都属于D亚族,同时D亚族包含的糜子GRF基因家族成员数量最多,而B亚族的基因家族成员最少。、、、、、、、和等9个基因外显子数目为4个,、、、、、和等7个基因外显子数目为3个(图4),说明GRF基因家族结构较为保守,含有3个或4个外显子是糜子GRF基因家族结构的主要特征。另外,和等4个基因只含有3′-UTR(非翻译区),和没有非翻译区,其余15个基因同时具有5′-UTR和3′-UTR。
糜子GRF基因家族上游2000 bp序列的启动子作用元件预测结果表明,糜子GRF基因家族共有25种顺式作用元件,按照功能可以划分为植物生长发育、植物激素响应和非生物或生物胁迫三大类(图5)。在植物生长发育类中,CAT-box(19)与分生组织表达相关,Sp1(68)是光响应顺式作用元件占比较大,除在上不存在外,其他家族成员上均有;ACE(3)参与光响应性,只在和上存在;G-box(96)是光响应调控作用元件并且在顺式作用元件中占比最大,每一个糜子GRF家族基因都含有;MRE(12)是参与光响应性的MYB结合位点;GCN4_motif(1)胚乳表达顺式作用元件仅存在于上;AACA_motif(2)胚乳特异性的阴性表达顺式作用元件仅存在于和上;MSA-like(2)细胞周期调控元件只存在于和上;RY-element(3)是种子特异性调控元件,只存在于和上;motif I(1)是根特异性的顺式调控元件,仅在上;circadian(6)是昼夜节律调控的元件。
在植物激素类中,TCA-element(12)为水杨酸响应性元件;ABRE(91)为脱落酸响应性元件,也是植物激素调节中占比最大的一类顺式作用元件;P-box(14)和TATC-box(3)参与赤霉素响应,仅存在于、和上;AuxRR-core(8)是生长素响应调控元件。
在非生物或生物胁迫类中,ARE(29)是对厌氧诱导必不可少的顺式调节元件,出现在76%的GRF基因家族成员;GC-motif(47)是参与缺氧特异性诱导的增强样元件,85%的GRF基因家族成员都含有;CGTCA-motif(82)MeJA是响应性调控元件,是该类顺式作用元件中最大的;TC-rich repeats(8)为防御和应激响应性元件;MBS(23)是参与干旱诱导的MYB结合位点;LTR(7)是低温响应性元件;CCAAT- box(16)是MYBHv1的结合位点。
2.3 糜子GRF基因家族在茎分生组织中的表达特征
通过测量,陇糜12号、张778的平均株高分别为148.4 cm和59.2 cm,检验结果显示陇糜12号株高极显著高于张778(<0.01)(图6,A和B),并且两品种株高的差异主要是各节间长度及穗长不同(图6C);陇糜12号茎部第1节间长至第5节间长都显著高于张778,茎部第6和第7节间长、穗下节间长和穗长都极显著高于张778(图6D)。
利用转录组测序数据,对糜子GRF家族基因茎分生组织表达特征进行了分析。结果显示,基因在陇糜12号和张778节间分生组织、节部分生组织中的表达量存在差异(图7)。基因在陇糜12号和张778两个品种节间和节部的分生组织中都有表达,但表达量有所不同,其中和的表达量最高,而、、和在两个品种的节间和节部都未表达。此外,还发现、、、和在陇糜12号中不表达,但在张778中表达;而、、、、和在两个品种中几乎都不表达。在节间分生组织中,和在张778中的表达量显著高于陇糜12号,和在张778中的表达量极显著高于陇糜12号,、和在陇糜12号中的表达量显著高于张778。在节部分生组织中,在张778中的表达量极显著高于陇糜12号,在张778中的表达量显著高于陇糜12号,在陇糜12号中的表达量极显著高于张778,和在陇糜12号中的表达量显著高于张778。由此推测,糜子GRF基因家族成员参与了糜子茎的生长发育。
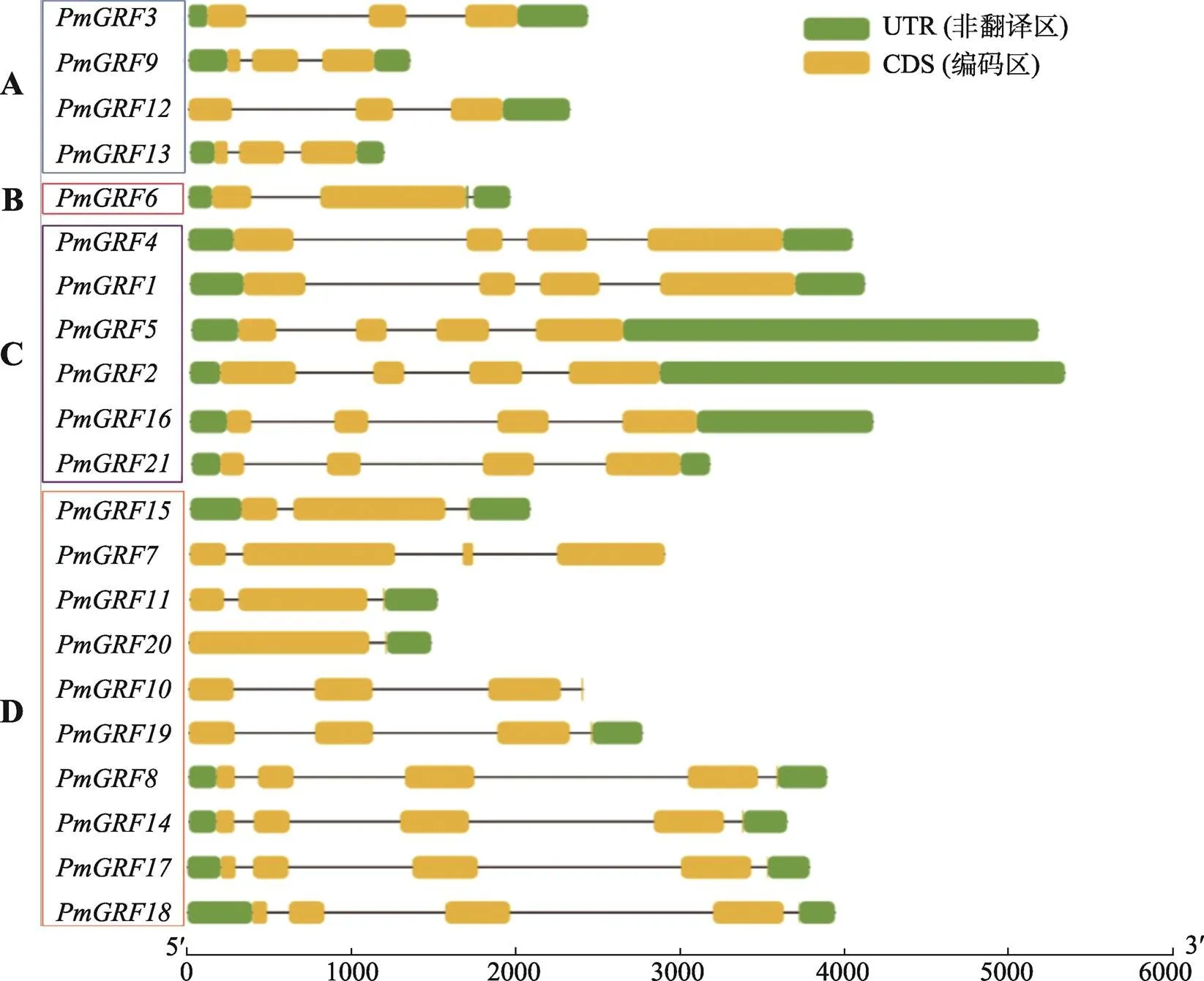
图4 糜子GRF基因家族结构
A、B、C和D表示4个亚族。
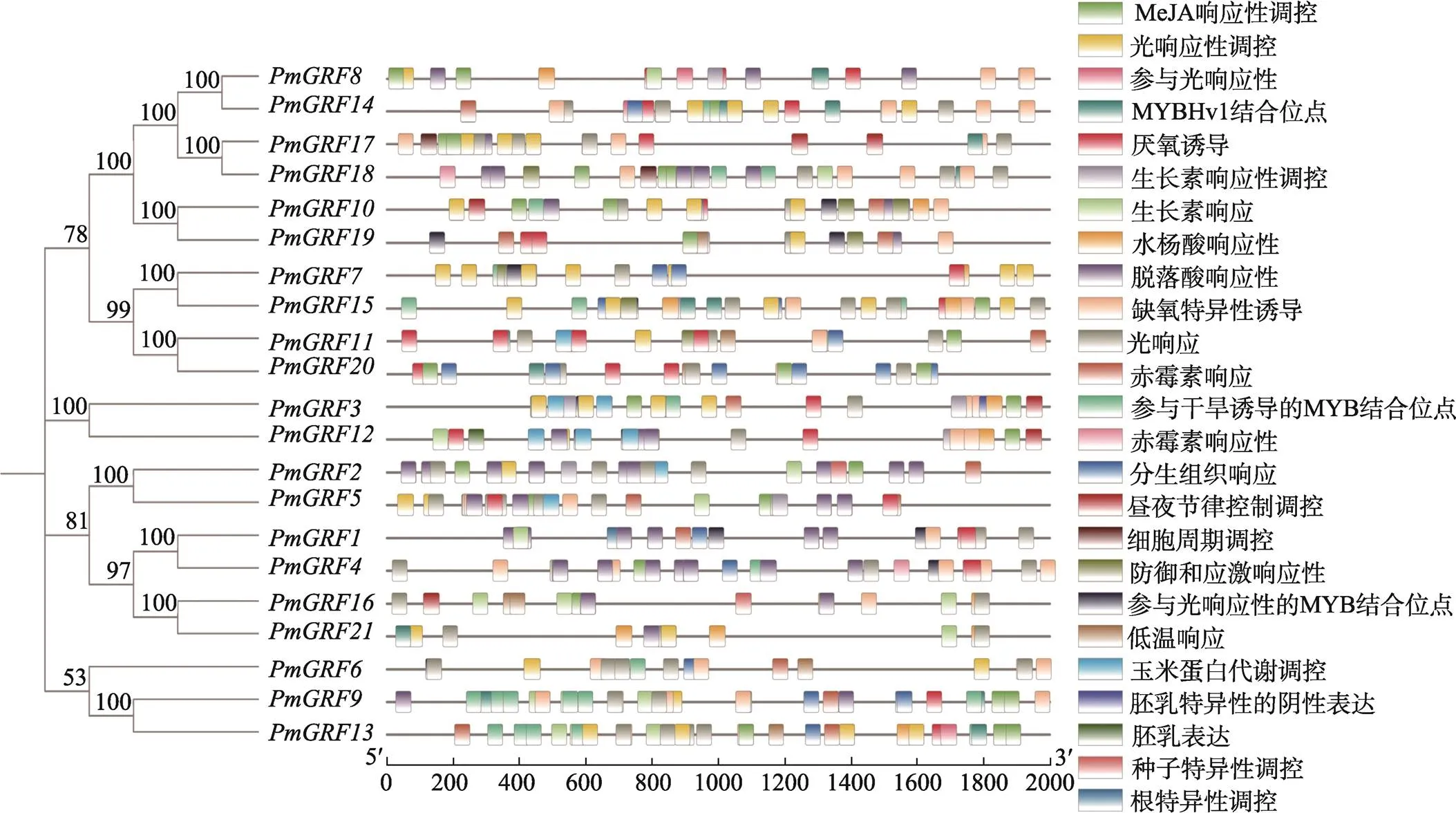
图5 糜子GRF基因家族顺式作用元件
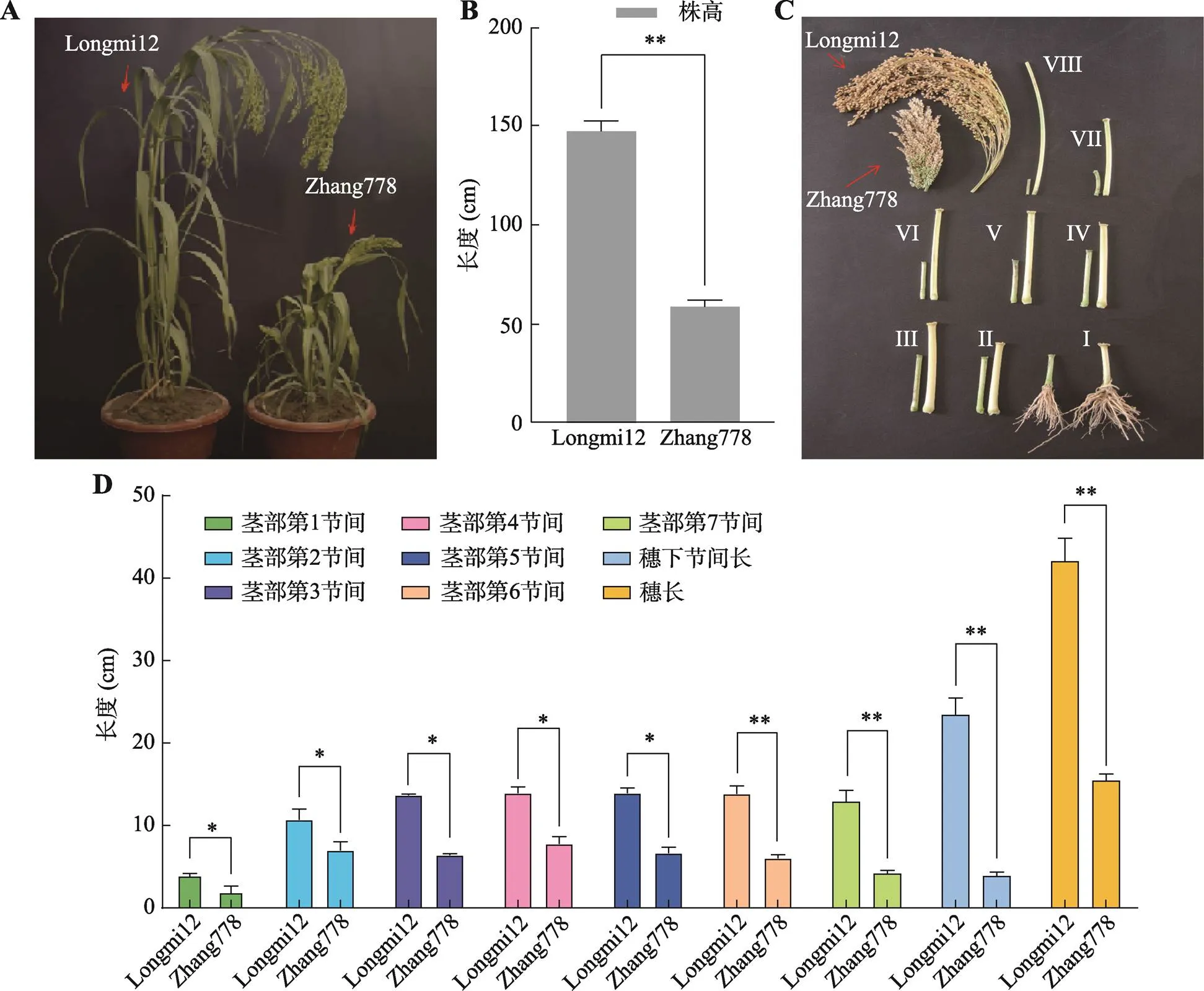
图6 陇糜12号和张778株高特征及各节间、穗长长度比较
A:陇糜12号和张778株型;B:陇糜12号和张778的株高差异比较;C:陇糜12号和张778各节间长度、穗下节间长度和穗长表型特征。I:穗下节长;II:茎部第1节间;III:茎部第2节间;IV:茎部第3节间;V:茎部第4节间;VI:茎部第5节间;VII:茎部第6节间;VIII:茎部第7节间;D:陇糜12号和张778茎部各节间长度、穗下节间长度和穗长差异比较。*:<0.05,**:<0.01。
选取糜子GRF基因家族中转录组测序差异显著的7个基因进行qRT-PCR分析(图8)。结果表明,这7个基因在两个品种节间及茎节分生组织中的表达均存在显著差异,和在张778节间和茎节的表达量都高于陇糜12号,而、和在陇糜12号节间和茎节的表达量均高于张778;和在张778节间的表达量高于陇糜12号,茎节的表达量低于陇糜12号,这与转录组测序数据结果基本相符,进一步说明糜子GRF基因家族成员参与了糜子株高的形态建成。
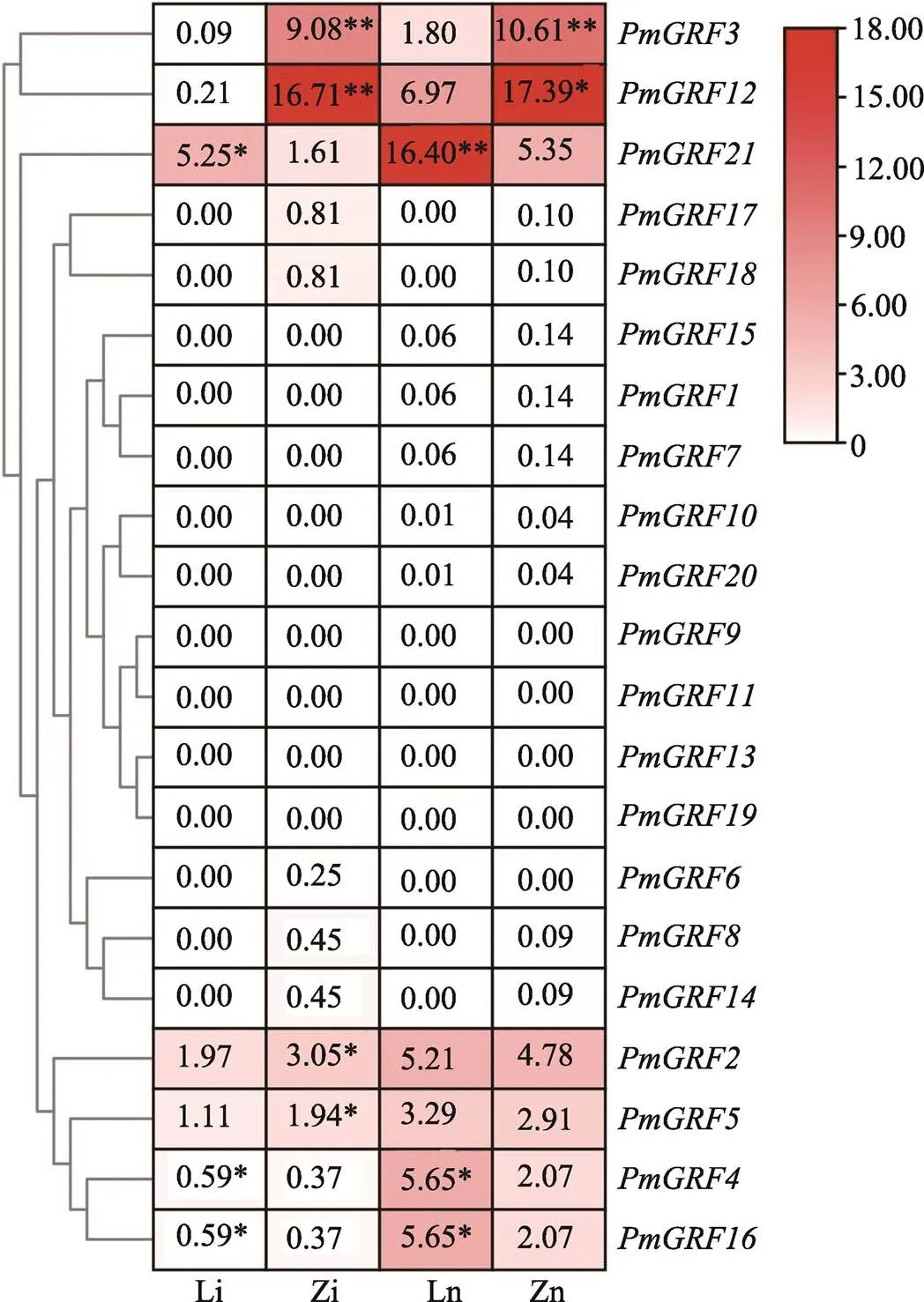
图7 陇糜12号和张778 GRF基因家族转录组表达分析
Li和Zi分别表示陇糜12号和张778节间分生组织;Ln和Zn分别表示陇糜12号和张778节部分生组织;*:<0.05,**:<0.01。
3 讨论
GRF是植物特异性转录因子的一员,在植物生长发育中起着极其重要的作用,其功能在很多植物中得到解析。基因主要在植物分生组织中表达,在多个发育阶段和胁迫下都发挥重要作用[36]。
本研究通过系统进化树发现,糜子GRF基因家族中的21个成员被划分为4个亚族。与的相似性高于,说明水稻和糜子在基因进化中亲缘关系更近;但在B亚族上只有拟南芥和糜子家族,表明糜子可能在进化中发生了某种基因复制和变异,这与棉花(spp.)基因家族的研究结果相似[37]。在D亚族中,拟南芥与糜子GRF基因家族成员进化距离较远,其中的进化距离最远,而且另一个拟南芥成员的自展值仅为29,说明该基因与糜子以及水稻的基因相似度较低,也体现出单子叶植物与双子叶植物在进化方式和行为模式上存在不同[18]。
从基因家族保守基序来看,糜子GRF基因家族21个成员都具有motif1和motif2这两个基序,说明motif1和motif2两个基序在PmGRF中高度保守,且在DNA结合中起关键作用,这与WRC结构域上的基序组成密切相关[38]。糜子GRF基因家族成员中拥有最多外显子数目(5)和最少外显子数目(2)的基因都属于D亚族,表明D亚族中的功能复杂且多样。分析也发现,糜子GRF基因家族在同一个亚族上的基因长度以及内含子、外显子结构相似,说明糜子GRF基因家族亚族间的功能差异较大。

图8 GRF基因家族在陇糜12号和张778茎节间和节部分生组织中相对表达量
A~G:分别表示、、、、、和在陇糜12号和张778茎节间和节部分生组织中相对表达量。LiZi表示在茎节间分生组织中,陇糜12号较张778的相对表达量,LnZn表示在茎节分生组织中,陇糜12号较张778的相对表达量;*:<0.05,**:<0.01。
分析发现,糜子GRF基因家族共有25种顺式作用元件,与分生组织表达、光响应、胚乳表达、细胞周期调控、植物激素响应调节、厌氧诱导、防御应激、干旱诱导和低温响应等相关。在众多顺式作用元件中,占比最大的是对光响应起调控作用的G-box,其次是对脱落酸响应起作用的ABRE,而后是诱导茉莉酸甲酯响应植物化学防御的CGTCA- motif,最后是对光响应起作用的Sp1,这表明糜子GRF家族大多数成员参与光响应过程以及植物激素调节。对大白菜(L. ssp.)的研究也发现,基因在植物激素调节中起作用,例如用赤霉素处理的大白菜对12个基因表达量有明显影响,在叶芽或幼叶中有较高表达量[39]。此外,大豆基因也在植物光合作用过程中参与表达调控,遮荫胁迫下大豆叶片大小、叶面积和干鲜重显著下降,且几乎所有基因在遮荫条件下表达量都下调[16]。对蓖麻() GRF基因家族的研究发现,与调控蓖麻株高的基因可能存在直接或间接关联,利用外源GA对高秆和矮秆蓖麻嫩茎的处理发现,响应外源GA应答,在矮秆蓖麻表达量增加、高秆蓖麻表达量下降[7]。研究发现,小麦(L.)GRF家族成员在茎尖分生组织发育中起关键作用[8]。本研究对糜子GRF基因家族成员在其节间和节部表达特征分析也发现,各成员在茎节间和茎节部表达。转录组分析发现,糜子GRF基因家族成员在陇糜12号的茎节部表达量比节间高,而在张778中节部的表达量大多比节间高。研究也显示,、和在两个部位的分生组织上表达量最为明显,说明糜子GRF基因家族成员参与茎器官的形态建成。糜子GRF基因家族成员在茎上部位的表达量总体表现为张788比陇糜12号高,但不同成员在不同部位的表达量差异较大。例如,和在张778节间和节部的表达量都显著高于陇糜12号,而和在张778节间的表达量则显著高于陇糜12号,但在节部的表达量差异不大。和在陇糜12号节间和节部的表达量均显著高于张778,在陇糜12号节间和节部的表达量都显著高于张778,这可能与糜子品种遗传特性相关。陇糜12号株高介于163.1~188.5 cm之间[34],为高杆品种;张778株高介于58.3~72.1 cm之间[35],是典型的矮秆品种。GRF基因家族成员的上调表达可能对高秆糜子品种茎伸长生长起到了一定的抑制作用。
本研究鉴定出糜子GRF基因家族包含21个成员,分为4个亚族;GRF基因家族成员的理化性质复杂、结构多样,在糜子高矮秆品种茎部分生组织中差异表达,其中、、、、、和可能参与了糜子茎器官的形态建成,研究结果为探究糜子基因在其株高形态建成中的功能和机理提供了科学依据。
[1] Liebsch D, Palatnik JF. MicroRNA miR396, GRF transcription factors and GIF co-regulators: a conserved plant growth regulatory module with potential for breeding and biotechnology., 2020, 53: 31–42.
[2] Yang XR, He SE, Chen SX. Research progress of GRF transcription factors in plants.2022, 39(3): 57–66.杨雪芮, 何沙娥, 陈少雄. GRF转录因子在植物中的研究进展. 桉树科技, 2022, 39(3): 57–66.
[3] Aida M, Beis D, Heidstra R, Willemsen V, Blilou I, Galinha C, Nussaume L, Noh YS, Amasino R, Scheres B. Thegenes mediate patterning of theroot stem cell niche., 2004, 119(1): 109–120.
[4] Shi PB, He B, Fei YY, Wang J, Wang WY, Wei FY, Lv YD, Gu MF. Identification and expression analysis of GRF transcription factor family of., 2019, 45(12): 1841–1850.时丕彪, 何冰, 费月跃, 王军, 王伟义, 魏福友, 呂远大, 顾闽峰. 藜麦GRF转录因子家族的鉴定及表达分析. 作物学报, 2019, 45(12): 1841–1850.
[5] Kim JH, Kende H. A transcriptional coactivator, AtGIF1, is involved in regulating leaf growth and morphology in., 2004, 101(36): 13374–13379.
[6] Rodriguez RE, Ercoli MF, Debernardi JM, Breakfield NW, Mecchia MA, Sabatini M, Cools T, de Veyldeer L, Benfey PN, Palatnik JF. MicroRNA miR396 regulates the switch between stem cells and transit-amplifying cells inroots., 2015, 27(12): 3354–3366.
[7] Dai MY, Gao M, Li WC. Bioinformatics identification and expression analysis of GRF transcription factor family of., 2019, 19(22): 7383–7390.代梦媛, 高梅, 李文昌. 蓖麻GRF转录因子家族生物信息学鉴定及表达分析. 分子植物育种, 2021, 19(22): 7383–7390.
[8] Huang WD, He YQ, Yang L, Lu C, Zhu YX, Sun C, Ma DF, Yin JL. Genome-wide analysis of growth-regulating factors (GRFs) in., 2021, 9: e10701.
[9] Yuan Q, Zhang CL, Zhao TT, Xu XY. Research advances of GRF transcription factor in plant., 2017, 36(8): 3145–3151.袁岐, 张春利, 赵婷婷, 许向阳. 植物中GRF转录因子的研究进展. 基因组学与应用生物学, 2017, 36(8): 3145–3151.
[10] van der Knaap E, Kim JH, Kende H. A novel gibberellin- induced gene from rice and its potential regulatory role in stem growth., 2000, 122(3): 695–704.
[11] Choi D, Kim JH, Kende H. Whole genome analysis of thegene family encoding plant-specific putative transcription activators in rice (L)., 2004, 45(7): 897–904.
[12] Kim JH, Choi D, Kende H. The AtGRF family of putative transcription factors is involved in leaf and cotyledon growth in., 2003, 36(1): 94–104.
[13] Vroemen CW, Mordhorst AP, Albrecht C, Kwaaitaal MACJ, de Vries SC. Thegene is required for boundary and shoot meristem formation in., 2003, 15(7): 1563–1577.
[14] Chen HL, Ge WN. Identification, molecular characteristics, and evolution of GRF gene family in foxtail millet (L)., 2022, 12: 727674.
[15] Zhang DF, Li B, Jia GQ, Zhang TF, Dai JR, Li JS, Wang SC. Isolation and characterization of genes encoding GRF transcription factors and GIF transcriptional coactivators in maize (L.)., 2008, 175(6): 809–817.
[16] Chen F, Yang YZ, Luo XF, Zhou WG, Dai YJ, Zheng C, Liu WG, Yang WY, Shu K. Genome-wide identification of GRF transcription factors in soybean and expression analysis offamily under shade stress., 2019, 19(1): 269.
[17] Zhang JF, Li ZF, Jin JJ, Xie XD, Zhang H, Chen QS, Luo ZP, Yang J. Genome-wide identification and analysis of the growth-regulating factor family in tobacco ()., 2018, 639: 117–127.
[18] Wang Y, Zhang LN, Tang FY, Zhao XX, Xi YJ, Wang WW. Genome-wide identification and analysis of GRF transcription factors family in switchgrass., 2019, 30(3): 575–586.王燕, 张礼宁, 唐方毅, 赵晓晓, 奚亚军, 王伟伟. 柳枝稷GRF转录因子家族全基因组鉴定与分析. 草地学报, 2022, 30(3): 575–586.
[19] Jin L, Hass Agula, Gao F. Genome-wide identification and analysis of growth regulating factor genes(GRF) in cucumis melo L., 2020, 39(8): 3554– 3560.金兰, 哈斯阿古拉, 高峰. 甜瓜GRF转录因子的全基因组鉴定和分析. 基因组学与应用生物学, 2020, 39(8): 3554–3560.
[20] Liu L, Li XJ, Li B, Sun MY, Li SX. Genome-wide analysis of thegene family and their expression profiling in peach ()., 2022, 17(1): 437–449.
[21] Li ZQ, Xie Q, Yan JH, Chen JQ, Chen QX. Genome-wide identification and characterization of the abiotic-stress- responsivegene family in diploid woodland strawberry ()., 2021, 10(9): 1916.
[22] Huang J, Chen GZ, Ahmad S, Hao Y, Chen JL, Zhou YZ, Lan SR, Liu ZJ, Peng DH. Genome-wide identification and characterization of thegene family in., 2023, 24(2): 1261.
[23] Dong KJ, Liu TP, He JH, Ren RY, Zhang L, Yang TY. Evaluation and identification indexes selection on the drought resistance of broomcorn millet bred cultivars at seeding stage., 2015, 16(5): 968–975.董孔军, 刘天鹏, 何继红, 任瑞玉, 张磊, 杨天育. 糜子育成品种苗期抗旱性评价与鉴定指标筛选. 植物遗传资源学报, 2015, 16(5): 968–975.
[24] Yuan YH, Liu CJ, Gao YB, Ma Q, Yang QH, Feng BL. Proso millet (L): a potential crop to meet demand scenario for sustainable saline agriculture., 2021, 296: 113216.
[25] Yang P, Panhwar RB, Li J, Gao JF, Gao XL, Wang PK, Feng BL. Changes of yield and traits of broomcorn millet cultivars in China based on the data from national cultivars regional adaptation test., 2017, 50(23):4517–4529. 杨璞, Rabia Begum Panhwar, 李境, 高金锋, 高小丽, 王鹏科, 冯佰利. 基于国家品种区域试验数据的中国糜子品种产量和性状变化. 中国农业科学, 2017, 50(23): 4517–4529.
[26] Lin FY, Wang SQ, Hu YG, He BR. Cloning of A S-adenosylmethionine synthetase gene from broomcorn millet (L.) and its expression during drought and re-watering., 2008, 34(5): 777–782.林凡云, 王士强, 胡银岗, 何蓓如. 糜子SAMS基因的克隆及其在干旱复水中的表达模式分析. 作物学报, 2008, 34(5): 777–782.
[27] Lin FY, Wang SQ, Hu YG, He BR. Cloning and expression analysis of drought-tolerant and water saving genein broomcorn millet., 2008, 30(3): 373–379.林凡云, 王士强, 胡银岗, 何蓓如. 糜子抗旱节水相关基因的克隆及表达分析. 遗传, 2008, 30(3): 373–379.
[28] Pan WX, Liu TP, He JH, Dong KJ, Ren RY, Zhang L, Yang TY. Genome-wide identification and expression characteristics of the YABBY gene family under hypertonic solution stress in broomcorn millet (L)., 2022, 41(5): 1067–1078.盘婉向, 刘天鹏, 何继红, 董孔军, 任瑞玉, 张磊, 杨天育. 糜子L)全基因组YABBY基因家族鉴定与高渗溶液胁迫下表达特征. 基因组学与应用生物学, 2022, 41(5): 1067–1078.
[29] Wang M, Liu TP, He JH, Dong KJ, Ren RY, Zhang L, Yang TY. Genome-wide identification of bZIP gene family in broomcorn millet and analysis of its expression characteristics under polyethylene glycol treatment in seedling stage., 2022, 28(4): 920–930.王媚, 刘天鹏, 何继红, 董孔军, 任瑞玉, 张磊, 杨天育. 糜子bZIP基因家族鉴定及幼苗期聚乙二醇6000处理下的表达特征. 应用与环境生物学报, 2022, 28(4): 920–930.
[30] Xin XX, Zheng XR, Wang HG, Chen L, Dipak KS, Wang RY, Qiao ZJ. Cloning and bioinformatics analysis ofin broomcorn millet., 2023, 51(10):1162–1169.辛旭霞, 郑香然, 王海岗, 陈凌, Santra Dipak K, 王瑞云, 乔治军. 糜子的克隆及生物信息学分析. 山西农业科学, 2023, 51(10): 1162–1169.
[31] Finn RD, Coggill P, Eberhardt RY, Eddy SR, Mistry J, Mitchell AL, Potter SC, Punta M, Qureshi M, Sangrador- Vegas A, Salazar GA, Tate J, Bateman A. The Pfam protein families database: towards a more sustainable future., 2016, 44(D1): D279–D285.
[32] Kumar S, Stecher G, Tamura K. MEGA7: molecular evolutionary genetics analysis version 7.0 for bigger datasets., 2016, 33(7): 1870–1874.
[33] Ren RY, He JH, Dong KJ, Zhang L, Liu TP, Yang TY. Report on new-bred broomcorn millet cultivar Longmi 12., 2017, (3): 14–16.任瑞玉, 何继红, 董孔军, 张磊, 刘天鹏, 杨天育. 糜子新品种陇糜12号选育报告. 甘肃农业科技, 2017,(3): 14–16.
[34] Zhang B, Jia XP, Yang DZ, Zhao Y, Dai LF, Kou SJ, Zhang XM, Hou DY, Zhu XH. Investigation on agronomic characters of dwarf mutant inand analysis of its sensitivity to GA., 2019, 31(5): 688–694.张博, 贾小平, 杨德智, 赵渊, 戴凌峰, 寇淑君, 张小梅, 侯典云, 朱学海. 糜子矮秆突变体778农艺性状调查及其对GA的敏感性分析. 浙江农业学报, 2019, 31(5): 688–694.
[35] Livak KJ, Schmittgen TD. Analysis of relative gene expression data using real-time quantitative PCR and the 2(-Delta Delta C(T)) method., 2001, 25: 402–408.
[36] Cao JF, Huang JQ, Liu X, Huang CC, Zheng ZS, Zhang XF, Shangguan XX, Wang LJ, Zhang YG, Wendel JF, Grover CE, Chen ZW. Genome-wide characterization of the GRF family and their roles in response to salt stress in., 2020, 21(1): 1–16.
[37] Wang PJ, Zheng YC, Lin Y, Zhou Z, Yang JF, Ye NX. Genome-wide identification and expression analysis of GRF gene family in., 2019, 39(3): 413–421.王鹏杰, 郑玉成林浥, 周珍, 杨江帆, 叶乃兴. 茶树GRF基因家族的全基因组鉴定及表达分析. 西北植物学报, 2019, 39(3): 413–421.
[38] Zafar I, Rubab A, Aslam M, Ahmad SU, Liyaqat I, Malik A, Alam M, Wani TA, Khan AA. Genome-wide identification and analysis of GRF (growth-regulating factor) gene family inthrough in silico approaches., 2022, 34(4): 102038.
[39] Wang FD, Qiu NW, Ding Q, Li JJ, Zhang YH, Li HY, Gao JW. Genome-wide identification and analysis of the growth-regulating factor family in Chinese cabbage (L. ssp.)., 2014, 15(1): 1–12.
Genome-wide identification of GRF transcription factors and their expression profile in stem meristem of broomcorn millet (L.)
Heng Wei1, Tianpeng Liu2, Jihong He2, Kongjun Dong2, Ruiyu Ren2, Lei Zhang2, Yawei Li2, Ziyi Hao1, Tianyu Yang1,2
To understand the genome-wide information of the GRF family genes in broomcorn millet and their expression profile in the vegetative meristems, bioinformatic methods and transcriptome sequencing were used to analyze the characteristics, physical and chemical properties, phylogenetic relationship, chromosome distribution, gene structure,-acting elements and expression profile in stem meristem for the GRF family members. The results showed that the GRF gene family of millet contains 21 members, and thegene is unevenly distributed on 12 chromosomes. The lengths of PmGRF proteins vary from 224 to 618 amino acids, and the isoelectric points are between 4.93-9.69. Each member of the family has 1-4 introns and 2-5 exons. The proteinis localized in both the nucleus and chloroplast, and the rest PmGRF proteins are located in the nucleus. Phylogenetic analysis showed that the 21genes were divided into 4 subfamilies (A,B,C and D) in broomcorn millet. The analysis of-acting elements showed that there were many-acting elements involved in light response, hormone response, drought induction, low temperature response and other environmental stress responses in the 2000 bp sequence upstream of thegenes. Transcriptome sequencing and qRT-PCR analyses showed that the expression levels ofandin the dwarf variety Zhang778 were significantly higher than those of the tall variety Longmi12 in the internode and node meristems at the jointing stage, while the expression patterns of,andwere reverse. In addition, the expression levels ofandin the internode of Zhang778 were significantly higher than Longmi12. The othergenes were not or insignificantly expressed. These results indicated that seven genes,,,,,,and, were related to the formation of plant height in broomcorn millet.
L.;genes; transcriptome; plant height
2023-08-01;
2024-01-10;
2024-01-26
第二次青藏高原综合科学考察研究项目(编号:2019QZKK0303),甘肃省科技计划项目(编号:22CX8NA028)和国家现代农业产业技术体系项目(编号:CARS-06-14.5-A8)资助[Supported by the Second Tibetan Plateau Scientific Expedition and Research Program (No.2019QZKK0303), the Science and Technology Planning Project of Gansu Province (No. 22CX8NA028) and the China Modern Agricultural Industrial Technology System Project (No.CARS-06-14.5-A8)]
韦恒,硕士研究生,专业方向:小杂粮遗传育种。E-mail: 1715414764@qq.com
杨天育,硕士,研究员,研究方向:小杂粮遗传育种与栽培。E-mail: 13519638111@163.com
10.16288/j.yczz.23-210
(责任编委: 宿振起)