光合细菌对活性污泥微生物群落结构及功能的影响
杨文焕,邓子威,徐 岩,王志超,李卫平*(1.内蒙古科技大学能源与环境学院,内蒙古 包头 014010;2.黄河流域内蒙古段生态保护与综合利用自治区协同创新中心,内蒙古 包头 014010)
光合细菌因光合作用机制的特殊性和营养代谢模式的多样性,在污水生物处理领域具有较大潜力[1-2].已有研究[3-4]将光合细菌应用于生物处理工艺,发现能提高系统对目标污染物的去除性能.目前有关光合细菌的研究主要聚焦于处理各种类型的有机废水,现有研究阐述了光合细菌作用下活性污泥系统处理性能与微生物群落结构的关系,但对微生物群落功能和代谢途径的深入认识较为有限.
废水脱氮是全球性环境问题[5].氨氮、硝态氮和亚硝态氮之间的相互转化是传统生物脱氮机理的基础[6],这一过程需要大量氧气和碳源供应[7].光合细菌的多种能量和物质代谢途径使其具有良好的脱氮性能,光合细菌污水处理技术在污染物去除方面前景广阔[8].Yang 等[9]发现光合细菌可能具有新的氮转化途径,即在光合细菌氮代谢过程中NH4+-N 可直接氧化为N2/N2O,突破了现有技术的限制.这种对光合细菌中氮代谢的新认识可能为进一步了解自然界的氮循环提供帮助.从代谢通路和基因方向着手对系统中氮循环功能微生物进行更加深入地研究,对水生态环境的治理与保护意义重大.
本文以光合细菌为生物强化菌种,构建光序批式活性污泥反应器,在反应器稳定运行条件下,分析光合细菌投加量、pH 值、光照强度和溶解氧(DO)对污染物去除效能的影响.采用高通量测序技术,探究活性污泥微生物群落结构和多样性的变化特征,基于PICRUSt2 功能预测和KEGG 数据库,探讨微生物功能通路、氮代谢功能基因及酶的种类和丰度变化,从分子生物层面揭示光合细菌作用机制,以期为光合细菌污水处理实际应用提供参考.
1 材料与方法
1.1 实验用水及接种污泥
实验进水采用模拟生活污水,分别以葡萄糖(C6H12O6)为碳源,氯化铵(NH4Cl)为氮源,磷酸二氢钾(KH2PO4)作为磷源.保持进水的CODCr、NH4+-N和TP 分别为500,40 和6mg/L 左右.添加碳酸氢钠(NaHCO3)提供碱度以保证反应器内pH值为7.0~7.5.并加入适量的微量元素营养液为微生物的生长提供必需的微量元素[10],微量元素营养液与进水体积比为1mL/L.
本实验所用活性污泥取自包头市某污水处理厂曝气池污泥,污泥沉淀性能较好,MLSS 约为6500mg/L.采用人工配置模拟生活污水对活性污泥进行培养,使污泥保持较好的理化性质,当污泥对CODcr和NH4+-N 的去除率稳定后,认为驯化完成,即可用于接种.实验接种的光合细菌菌液(纯种菌液)采购于广东省科学院微生物研究所,GDMCC 编号及菌种名称:GDMCC1.167,沼泽红假单胞菌(Rhodopseudomonas palustris).经实验室培养至稳定生长期时进行接种,此时菌浓度(OD600)为1.881,满足接种需求.
1.2 实验装置及运行条件
实验一共设置一组序批式反应器(SBR)和4 组光序批式反应器(PSBR)(图1),编号分别为R1~R5.R1 反应器(对照组)无需进行光照,R2~R5 反应器为了保持同样的光照强度,需要在反应器外围缠上相同数量的LED 光带(光照强度为4000lux[11]).反应器高30cm、内径10cm,有效容积2L,主要构件为:反应池、微孔曝气盘、曝气泵、搅拌器、转子流量计和LED 灯条.所有反应器周围环境条件保持相同.反应器侧壁设有取样口,DO 浓度由转子流量计进行调整,反应器最底端取样口为排泥口.

图1 实验装置示意Fig.1 Experimental setup diagram
5 组反应器以12h 为一个反应周期:进水0.2h,曝气7h,沉淀4h,排水0.2h,静置0.6h.沉淀时光照,黑暗时曝气搅拌(光暗比为4:7).待反应器中混合液完全沉降后,手动排水,每次排水的体积为混合液的50%,当液位下降到1000mL 处时,停止排水,每天手动排泥100mL.各组反应器的pH 保持在7.0~7.5,DO浓度维持在2.5~3.5mg/L. R1~R5 反应器中光合细菌投加量分别为0%、1%、5%、10%和15%(菌液体积与反应器有效容积比),其中R1 反应器为空白对照组不加菌.
1.3 单因素实验设计
在光照强度为4000lux,DO 为3mg/L 时,设pH值为5,6,7,8 和9.在pH 7 和DO 为3mg/L 时,设光照强度为2000,3000,4000,5000,6000lux.在pH 7和光照强度4000lux 时,设DO 为1,2,3,4,5mg/L.DO 和pH值变化时,光照强度为固定值(强度大小通过缠绕反应器LED 灯带数量调整);光照强度变化时,DO 由转子流量计进行调整稳定,通过添加NaHCO3提供碱度以保证反应器内pH 值稳定.光合细菌投加量为10%,其他实验条件与运行参数同1.2,单因素试验运行天数为16d.
1.4 分析项目及检测方法
1.4.1 常规理化指标测定 采集水样经过0.45μm水相滤膜过滤后用于理化指标测定.理化指标测定方法参照《水和废水监测分析方法(第四版)》[12],对CODCr、NH4+-N、NO2--N、NO3--N、TP、TN 进行检测.采用便携式水质分析仪(WTW-Multi3420)对DO 进行测定,采用pH 计(PHS-25)对pH 值监测.
1.4.2 DNA 提取、PCR 扩增和高通量测序 将样品加干冰运输至上海美吉生物医药科技有限公司完成DNA 提取、PCR 扩增和16S rRNA 基因测序.细菌DNA的提取利用E.Z.N.A.® soil试剂盒完成[13];利用 338F(5'-ACTCCTACGGGAGGCAGCAG-3')和806R(5'GGAC-TACHVGGGTWTCTAAT -3')引物以V3~V4 可变区为对象,在此基础之上展开相应的PCR 扩增[14],程序如下:95°C 的温度条件下预变性,此环节所对应的时间为3min,循环的次数则设定为27 个,72°C 的温度条件下延伸,此环节所对应的时间为10min.扩增体系20µL,4µL 5×FastPfu(5 倍浓度)缓冲液,2µL 2.5mmol/L dNTPs,引物(5µmol/L)在该体系中所需添加量具体为0.8µL,FastPfu 聚合酶在该体系中所需添加量具体为0.4µL;DNA 模板在该体系中所需添加量具体为10ng.利用2%琼脂糖凝胶电泳,检测PCR 产物,3µL 上样检测电泳图.通过Miseq PE300 进行测序,基于Trimmomatic 来操控原始序列数据,后续拼接在此次实验中采用的软件为FLASH[15].利用PICRUSt2 预测微生物群落功能能力,并利用KEGG 数据库进行注释,对微生物群落和氮代谢功能进行预测.
1.5 数据分析与处理
高通量测序数据统计分析利用Majorbio 公司的i-sanger 平台进行.测序得到的PE reads 首先根据overlap 关系进行拼接,同时使用fastp(https://github.com/OpenGene/fastp,version 0.20.0)对序列质量进行质控和过滤,使用 Mothur(versionv.1.30.1)软件按97%的相似性将有效序列进行 OTU 聚类,得到OTU(Operational Taxonomic Units)代表序列.采用RDP classifier 贝叶斯算法对OTU 代表序列进行分类学分析,并分别在各个分类水平统计样本的群落物种组成,比对数据库为Silva 数据库.基于OTU 信息,使用Mothur v1.30.1 计算Shao 丰富度和Simpson指数[16].对OTU 丰度表进行标准化,去除16S marker gene 在物种基因组中的copy 数目的影响;每个OTU对应greengene id,获得OTU 对应的COG 家族信息和KEGG Ortholog(KO)信息;计算各COG 的丰度和KO 丰度.根据KEGG 数据库的信息,获得KO、Pathway、EC 信息,并根据OTU 丰度计算各功能类别的丰度.针对Pathway,运用PICRUSt 可获得代谢通路的3 个水平信息,分别得到各个水平的丰度表.将样品序列抽平后使用q2-diversity 计算α 多样性指数.为获得 OTUs 对应的物种分类信息,基于QIIME 平台和Silva 数据库,采用RDP classifier 对OTUs 代表序列进行物种分类注释,并分别在各个分类水平统计样本的群落组成.运用 Origin8.5 和TBtools 进行分析和制图.水质数据单因素方差分析和多重的比较采用SPSS22.0 进行,利用Excel2016和Origin8.5 进行分析和制图.
2 结果与讨论
2.1 PSBR 污染物去除效果及影响因素分析
2.1.1 PSBR 污染物去除性能变化 如图2 所示,R3 和R4 反应器稳定运行期间,平均出水CODCr浓度分别为49.35,36.63mg/L,F 检验法(方差分析)结果显示,R1、R3 和R4 出水CODCr存在极显著性差异(P<0.01),5%和10%光合细菌的投加强化了活性污泥对CODCr的去除.运行稳定时,R1~R5 反应器平均出水 NH4+-N 浓度分别为 5.88,5.01,3.29,2.88,9.58mg/L.光合细菌通过光合作用和呼吸作用两种代谢途径吸收污水中的NH4+-N,为自身生长代谢提供能量[17],同时活性污泥的生长也需要吸收NH4+-N.反应器运行后期对TN 的去除效果均强于初期,推测光合细菌投加可能有助于细菌的反硝化作用,进水中足够的CODCr也有利于脱氮效率的稳定[18],促进了反应器对TN 的去除效果.五组反应器的除磷效果在培养的前16d 基本保持不变,可能是聚磷菌(PAOs)正处于适应阶段.运行中期(19~46d),五组反应器对TP 的去除均有不同程度的提高,其中R4 反应器出水TP 由22d 的2.19mg/L 减少至46d 的0.74mg/L.
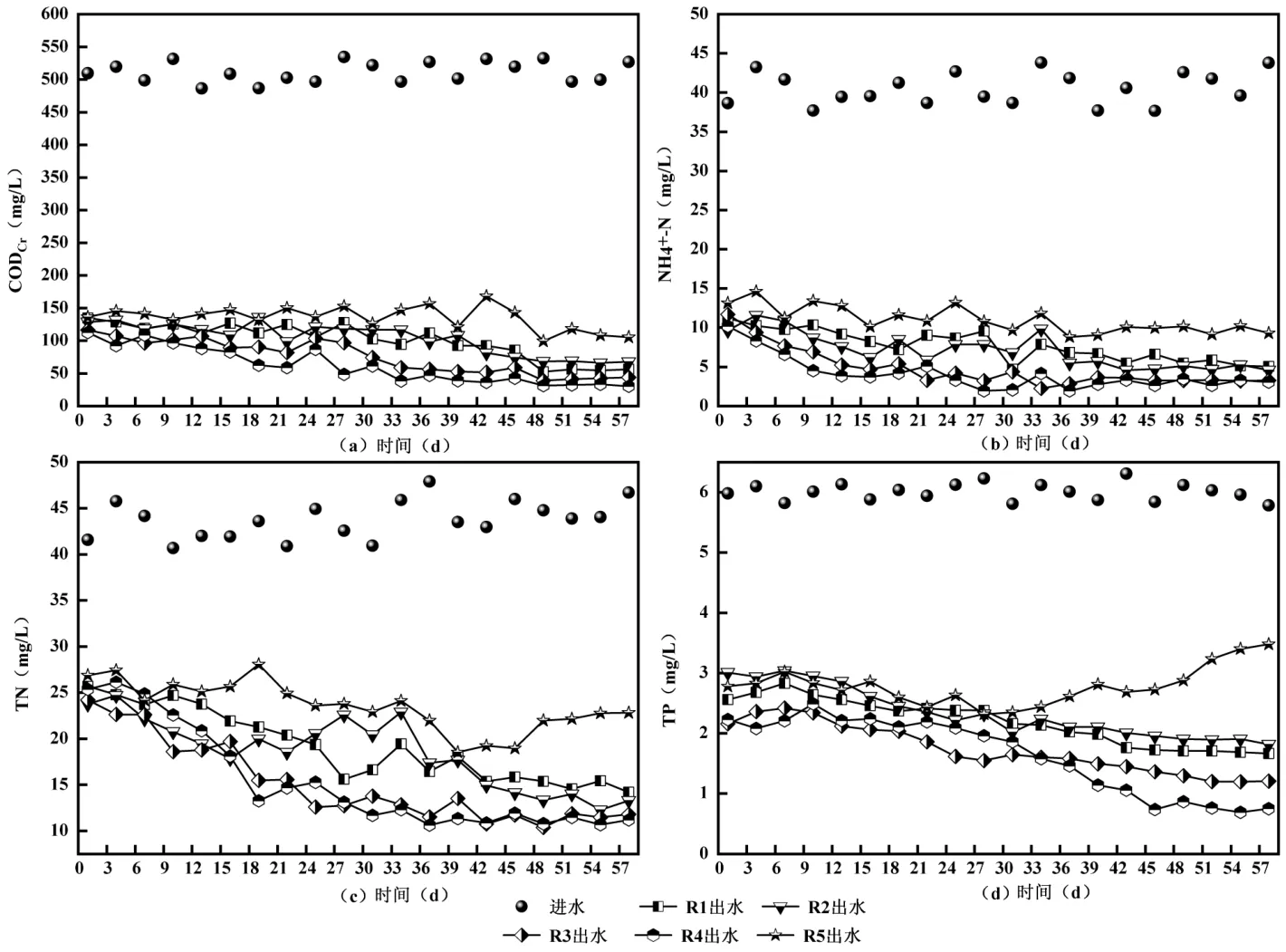
图2 光合细菌投加量对(a)CODCr、(b)NH4+-N、(c)TN、(d)TP 去除效果Fig.2 Effect of photosynthetic bacteria dosing on(a) CODCr,(b) NH4+-N,(c) TN and(d) TP removal

图3 pH 值、光强和DO 对NH4+-N、NO2--N、NO3--N 和TN 去除效果影响Fig.3 Effect of pH, light intensity and DO on NH4+-N,NO2--N, NO3--N and TN removal effects
2.1.2 PSBR 脱氮性能影响因素 实验选用NH4Cl为唯一氮源,PSBR 系统进水NH4+-N 浓度约为40mg/L.pH 值对PSBR 系统脱氮性能影响较为明显,pH为5,6时,PSBR系统NH4+-N去除率仅为31.3%,48.2%,出水 TN浓度为 34.4,29.7mg/L,并有少量NO3--N 积累,系统硝化反应受到抑制;pH 7,8 时,系统脱氮效果较好,出水TN 浓度为19.6,21.3mg/L;当初始pH 值增加至9 时,NH4+-N 去除率为88.7%,出水NO3--N和TN浓度为21.82, 26.80mg/L,说明系统硝化性能最好,但反硝化进程受到阻碍,出水TN 浓度并没有降低.综上,随着pH提高,PSBR 系统出水TN浓度呈先降低后升高趋势,从趋势可以看出pH 值与出水NH4+-N浓度呈负相关,与出水NO3--N浓度呈正相关.光照强度为2000~5000lux,出水NH4+- N、NO3--N 和TN 浓度与光照强度呈正相关,当光照强度为5000lux时,NH4+-N 和TN 去除率最高,分别为84.86%和57.99%,NO3--N 积累量较低为8.94mg/ L.光照强度提高至6000lux 时,污染物出去率并无显著提高,出水NH4+-N、NO3--N 和TN 浓度与光照强度为4000lux时无显著差异(P>0.05).DO 浓度对PSBR 系统脱氮性能影响与pH 值趋势相似,当DO 为3mg/L 时,PSBR系统NH4+-N 和TN 去除率分别为78.74%,59.41%,TN出水浓度较低.DO浓度与出水NH4+-N浓度呈负相关,与出水NO3--N 浓度呈正相 关.
2.2 微生物群落多样性与组成分析
2.2.1 微生物Alpha 多样性 5 组样品共获得优化序列469807 个,平均序列长度416bp,用于物种分类的OTUs 单元总计1411 个,分属34 门、89 纲、211目、325 科、539 属、871 种.Coverage 指数(样品基因文库覆盖率)可以反映测序结果中微生物样本库的覆盖范围,本次测序Coverage 指数均大于99%,能准确地反映出污泥样品中微生物群落的真实生物特征.通过单样本的Alpha 多样性指数对R1~R5 样品中活性污泥微生物群落多样性和丰富度进行分析(表1),常用四种指数分别为Chao1、Ace、Simpson和Shannon.Chao1 和Ace 指数反映了样品中微生物菌群丰富度,Ace 指数可以估计群落中含有OTUs 数目[19],Simpson 和Shannon 指数反映了样品中微生物菌群多样性[20].R1~R5 样本的Chao1 指数、Ace 指数和Shannon指数呈R5>R4>R3>R2>R1,且Simpson指数呈R1>R2>R3>R4>R5,说明投加光合细菌的活性污泥微生物丰富度和多样性均高于活性污泥反应器.

表1 微生物Alpha 多样性指数Table 1 Table of microbial Alpha diversity indices
2.2.2 门水平微生物群落组成 如图4 所示,5 组样品中变形菌门Proteobacteria 为最优势菌门,其相对丰度占比分别为35.62%、34.70%、38.71%、45.92%和49.44%.Proteobacteria 是污水处理厂活性污泥中常见的优势菌群,较多数反硝化细菌和脱氮微生物属于 Proteobacteria[21].R1~R5 五组样品中Proteobacteria 相对丰度依次增加,是由于投加的光合细菌属于变形菌门中 α 纲(红螺菌目),Proteobacteria 的大量存在是保证反应器能够正常运行的基础.拟杆菌门Bacteroidota 是一类化能有机营养菌,也是污水生物处理工艺中较常见的优势菌门,能参与有机物降解、反硝化、固氮等过程,其相对丰度占比分别为20.76%、15.59%、21.14%、19.34%和15.73%.Bacteroidota 具有较强的碳水化合物代谢能力,是降解CODCr的主要菌群.R3 和R4 样品中较高的相对丰度占比为反应器高效降解污染物提供良好的基础.

图4 门水平微生物群落组成Fig.4 Microbial community composition at the phylum level
2.2.3 属水平微生物群落组成 在属水平上5 组样品共鉴定出539 个菌属.5 组样品共有菌属为373个,5 组样品有23 个菌属至少在1 组样本中相对丰度大于1%,为样品中的优势菌属.投加光合细菌后,其对微生物的群落结构产生了影响(图5).腐螺旋菌科Saprospiraceae 为最优势菌属,其在五组样品中的相对丰度占比分别为10.92%、7.37%、17.74%、12.4%和16.6%,其次为鸟氨酸球菌属Ornithinibacter 和OLB12.红假单胞菌属Rhodopseudomonas 相对丰度占比分别为 1.99%、5.32%、7.18%、8.28%和2.23%.norank_f__Saprospiraceae 属于严格好氧型细菌,兼具反硝化与除磷功能,能够将复杂有机物降解为小分子碳源[22-24].Ornithinibacter 菌属在污水处理维持微生物群落结构,保证了系统的稳定运行.OLB12 菌属一般在污水处理系统中好氧池丰度较高,是一类好氧硝化优势菌属,在缺氧条件下则无法发挥作用[25].硝化菌多为自养型微生物,可能受实验环境影响,5 组样本中AOB 种类较少,且各菌属相对丰度较低.属于亚硝化单胞菌科(Nitrosomonadaceae)的硝化单胞菌属Nitrosomonas,是污水处理工艺中常见的AOB 菌属.R1 中Nitrosomonas 的相对丰度为 0.18%,而在 R4 中相对丰度增加至 0.54%.Rhodopseudomonas 菌属为本实验所投加的光合细菌,成为微生物群落主要优势菌属之一.R2~R4 样品中Rhodopseudomonas 菌属的相对丰度占比随光合细菌投加量的增大而提高,较高的丰度占比对反应器出水稳定有良好的促进作用.R5 样品中Rhodopseudomonas 菌属相对丰度占比较R4 样品反而下降了6.05%,这可能是因为较大的光合细菌投加量对原有活性污泥微生物群落有较大的冲击,破坏了其稳定结构,最后导致Rhodopseudomonas菌属相对丰度大幅下降.黄亮等[26]从石油炼化污水中筛选到1 株有机物高效降解菌B2,以其为特定微生物接种于活性污泥系统中,生物强化活性污泥系统对复配水污染物的去除,当菌株投加量超过3%时,污水CODCr去除性能和可生化性下降,可能是污水中碳源不足,使活性污泥与B2 菌之间产生竞争关系.

图5 属水平微生物群落组成Fig.5 Composition of genus level microbial community
2.3 微生物群落功能变化分析
2.3.1 KEGG 功能代谢通路 与代谢相关的功能通路相对丰度均在77.9%以上,代谢功能较高占比反映了微生物新陈代谢是各类菌属与环境之间主导的物质和能量交换主要过程.不同光合细菌投加量下微生物群落代谢功能具有差异性(图6).5 组样本均得到45 条KEGG 第二等级代谢通路,但R2~R5中各代谢通路的丰度相应地均高于R1.说明光合细菌增强了微生物群落代谢功能.样本中碳水化合物代谢(Carbohydrate metabolism)丰度占总功能的7%~9%,碳水化合物代谢是细胞生长和繁殖所需的能量和碳元素来源[27],菌体内的氨基酸代谢可以为氨基酸、核酸和维生素的生物合成提供前体细胞,而且还可以产生大量重要的芳香族化合物[28].这些代谢功能较高丰度是细胞赖以生长和繁殖的基础.能量代谢(Energy metabolism)、辅助因子和维生素代谢(Metabolism of cofactors and vitamins)和翻译(Translation)等同样是活性污泥微生物的重要代谢途径,其丰度分别在4.7%、4.4%和3.1%左右,几乎处在一个相同的水平仅次于碳水化合物和氨基酸代谢,能量代谢是生命活动最基本的特征,是维持一切生命活动的必要条件[29].

图6 KEGG 第二等级代谢通路注释Fig.6 KEGG second-order metabolic pathway annotation
2.3.2 氮代谢性能变化 脱氮是活性污泥的重要功能之一,参与氮代谢的功能基因在废水处理中起着至关重要的作用[30].本文利用PICRUSt2 对氮代谢通路及其相关功能基因进行了预测分析,通过与KEGG 库中65 个直系同源基因(KEGG Orthology)对比,5 组样本均获得48 个氮代谢功能基因.由于本实验选用氯化铵为唯一氮源,活性污泥中存在着4种氮代谢途径:硝化作用、反硝化作用、同化硝酸盐还原、异化硝酸盐还原.图7 为主要氮代谢通路、功能基因及其丰度.探究光合细菌对活性污泥氮代谢作用的影响,发现硝化过程中涉及的相关功能基因有amo(amoA/B/C)、hao 和nxr(nxrA/B). amo 基因编码的氨单加氧酶(AMO)能将NH4+-N 氧化为NH2OH,同时NH2OH 在hao 基因编码的羟胺氧化还原酶(HAO)催化下迅速氧化为NO2--N,nxr 基因编码的亚硝酸盐氧化还原酶(NXR)能最终将NO2--N氧化为NO3--N.在硝化过程中,NXR功能基因nxr 保持很高的丰度.NXR 是一个具有多重功能的酶,既可以催化亚硝酸盐的氧化,还可以催化硝酸盐的还原.amo 基因丰度与氨氧化速率呈正相关[31],AMO 和HAO 催化发生的氨氧化是硝化过程的限速步骤[32].光合细菌的投加使参与硝化过程的基因丰度明显提高,加快氨氧化速率,推动硝化过程的进行.与反硝化过程有关的基因包括nar(narG/H/I)、nap(napA/B/C) 、 nxr(nxrA/B) 、 nor(norB/C) 与nosZ.nar 和nap 基因是参与反硝化过程第一步反应的两种关键基因,能分别编码不同类型异化硝酸盐还原酶,将NO3--N 还原为NO2--N.nir 基因编码的亚硝酸盐还原酶(NIR)可进一步将NO2--N 还原为NO,nor 和nosZ 基因分别编码一氧化氮还原酶(NOR)和一氧化二氮还原酶(NOS),对应的参与NO还原和N2O 还原过程,使得反硝化的最终完成.同时,活性污泥中还存在大量有关异化硝酸盐还原过程的基因(nirB/D、nrfA/H)与同化硝酸盐还原过程的基因(narB、NR、nasA/B、nirA).异化硝酸盐还原可作为微生物的呼吸作用,同化硝酸盐还原可将氮素转化为生物质[33],这两个过程最终都将NO2--N还原为NH4+-N.
2.4 讨论
2.4.1 投加量及环境因子对PSBR性能影响 光合细菌投加量是影响PSBR 污染物去除的关键因素,生物处理系统内含有较为庞大微生物群体,在它们的协同作用下可使微生物群落功能基因和通路丰度增加.Yao 等[34]提出菌株投加量是生物处理技术成功的有效策略.投加量过少,光合细菌较难适应外界环境变化,或者在和活性污泥土著微生物竞争营养物质时衰亡,导致投加菌株接种率降低使污染物去除效果不明显[35],在本研究R2 反应器中红假单胞菌属Rhodopseudomonas(本实验投加的光合细菌)相对丰度占比较低,未形成最优势菌属;过量光合细菌会超额吸收污水中的碳源,抑制活性污泥正常生长代谢功能,影响PSBR 硝化反硝化性能,对活性污泥微生物群落结构和功能造成严重破坏[36].在本实验中15%投加量的R5 中微生物群落功能基因丰度和微生物多样性降低很好地验证了这一结论;综上所述,适当的光合细菌投加量会在一定程度上提高微生物对有机物污染物降解效果.
pH 值、光照和DO 是PSBR 脱氮性能的重要影响因子.利用活性污泥法处理污水时,pH 值的变化对微生物的生命活动有很大的影响,本研究中,pH 值与NH4+-N 去除率呈显著正相关(P<0.05),与NO3--N 去除率呈显著负相关(P<0.05).在好氧硝化过程中,酸性条件下过量的H+会抑制硝化细菌的活性,从而减慢氨氧化和硝化速率,且硝化阶段会消耗一定的碱度,过低的pH 无法保证充足的碱度,导致NH4+-N 去除率偏低[37].卫燕红等[38]研究显示光合细菌生长受pH 值影响显著,初始pH5 和9 时,光合细菌菌生长缓慢,当pH7~8 时,生长速度最快,活菌数最高,光合细菌适合在中性或弱碱环境中生存.综上,pH 值可直接影响活性污泥微生物硝化和反硝化进程,改变PSBR系统脱氮性能.
光合细菌是一种光合革兰氏阴性原核生物,通过胞内叶绿素和类胡萝卜素等各种光合色素进行无氧光合作用,将光能转化为化学能,用于其重要的细胞代谢活动[39].在适当的范围内,随着光强的增加,光合细菌可快速生长,当光强超过一定限度时,能量供应超过了细胞内酶的能力,导致光转换效率随光强的增加而降低[40].本研究中PSBR 系统污染物去除率与光照强度呈正相关,但光照强度存在阈值,当光照强度为6000lux 时,PSBR 系统污染物去除率并无明显提高.光照强度对PSBR 运行的影响基本与前人研究结论相吻合.在污水生物处理过程中,DO 水平高低密切影响硝化和反硝化进程,DO浓度较高,污泥活性高且微生物代谢旺盛,有机物去除速率较快.当同时考虑硝化反硝化反应及有机物去除时,硝化细菌比增长速率受DO 浓度影响不可忽视,研究显示活性污泥中硝化细菌约占5%左右,大部分硝化细菌处在生物絮凝体内部.增加DO 浓度,生物絮凝体穿透能力也将增加,硝化反应速率会被提高,生物反硝化过程必须保证严格缺氧环境,氧和硝酸盐会同时竞争电子供体,硝酸盐还原酶的合成及其活性也同时受到分子态氧的抑制影响[41].
2.4.2 PSBR 微生物群落组成与功能解析 门水平上变形菌门、拟杆菌门、放线菌门、绿弯菌门和厚壁菌门为优势菌群(图4),这与已有研究的结果较吻合[42].有研究认为变形菌门能调节微生物群落结构,其在有机物和氨氮去除中发挥着重要作用[43].本研究中变形菌门在 SBR 和 PSBR 中占比都较高(34.70%~49.44%),且丰度无显著变化(P>0.05),说明光合细菌投加不会改变微生物群落最优势菌门种类,变形菌门中具备氮素转化功能的氨氧化细菌、亚硝酸盐氧化菌和反硝化菌等,在PSBR 系统脱氮上发挥重要作用.SBR 和PSBR 中前四优势菌门相对丰度呈现显著差异(P<0.05).有研究表明,拟杆菌门也参与降解有机物、反硝化和固氮等过程,并在促进污泥成熟稳定方面发挥作用[44].光合细菌投加使放线菌门和绿弯菌门相对丰度显著升高(P<0.05),使R3 和R4 反应器取得较好的脱氮效果.
光合细菌投加提高了活性污泥微生物群落多样性和丰富度,这与已有的研究结果一致.有研究显示污水处理系统中存在能够分泌信号分子的菌体,菌间具有群体感应现象,当引入外源工程菌株进行生物强化使种群密度达到感应阈值,从而激活相关基因的表达,调控菌群生态关系.光合细菌投加可能使PSBR 系统内微生物群体感应信号增强,促进了细胞间信息传递与交流,提高微生物生长代谢活性,使微生物群落结构发生变化.
利用PICRUSt2 对系统运行稳定时细菌群落功能进行预测(图6).SBR 和PSBR 一级功能层基因丰度无显著差异(P>0.05),主要功能基因均为有机系统及代谢相关功能,说明有机系统和代谢在反应器稳定运行过程中起着极其重要的作用.SBR 和PSBR二级功能层的功能基因丰度存在差异,氨基酸代谢和硝化系统等功能基因丰度较高.本研究中进水为人工配水,提供无机氮源和碳源,整个过程主要是硝化菌和反硝化菌及其相关功能菌的富集过程,其它菌群被淘汰或者活性处于较低水平,这可能是功能基因丰度变化的原因之一.SBR 和PSBR 中微生物细菌参与氮素代谢的相关功能基因,共48 个KO 注释为氮代谢.由于本实验选用氯化铵为唯一氮源,活性污泥中存在着4 种氮代谢途径:硝化作用、反硝化作用、同化硝酸盐还原、异化硝酸盐还原.光合细菌刺激下使绝大部分氮代谢功能基因丰度增加,表明生物强化提升了活性污泥的整体氮代谢潜力.
3 结论
3.1 10%光合细菌投加下反应器对 CODCr、NH4+-N 和TN 去除率优于对照组,光合细菌与活性污泥微生物提高了PSBR 系统染物去除性能.
3.2 光合细菌投加使活性污泥系统微生物群落结构和特定微生物丰度产生差异,微生物群落丰富度和多样性得到提升.光合细菌有利于norank_f__Saprospiraceae、Ornithinibacter 和OLB12 等功能菌属的生长富集,为系统高效脱氮提供了良好菌群基 础.
3.3 光合细菌能促进活性污泥微生物功能基因表达的上调,特别是新陈代谢和群体感应等方面,通过调节信号传导机制改善菌群互作关系和部分功能代谢通路.光合细菌还能够提升关键氮代谢基因(amo、hao 和nap 等)和酶(AMO、HAO 和NAP)的丰度,使得活性污泥微生物在环境中硝化/反硝化潜在能力增强.

