基于时序互信息构建基因调控网络
缑葵香,宫秀军,汤 莉
(1. 天津大学理学院,天津 300072;2. 天津大学计算机科学与技术学院,天津 300072)
时间序列(time series)微阵列数据,可以反映一组基因在生命活动周期的时间序列条件下的表达水平的变化,其表达水平变化的时间延迟关系可反映基因调控关系,因而近年来被广泛地用于基因之间转录调控关系寻找和基因调控网络的构建.构建和分析基因调控网络,从分子水平认识细胞内的生理活动和功能,了解通路中的相互作用,以及如何使生物体产生变化.基于微阵列数据构建基因调控网络,逐渐成为生物信息学的研究热点.
很多数学模型如布尔网络模型、微分方程模型、静态贝叶斯网络模型和动态贝叶斯网络模型[1]等被提出用于构建基因调控网络.相对来讲,布尔网络模型是一种简单的离散模型,对系统的模拟是定性的、较为粗糙的.微分方程所给出的模型是确定性的、连续的,然而由于细胞中的分子受到热力学波动和噪声过程的支配,基因表达是一个随机过程,微分方程模型不能很好地描述基因表达的随机性.静态贝叶斯网络是一个有向无环的概率图形,它用概率来表示调控关系,更符合生物学实际.静态贝叶斯网络模型可以成功地从非时序数据中推断出随机变量之间的因果关系,但是不能对动态时间数据进行建模,因而不能成功地从时间序列的微阵列数据中构建基因调控网络.动态贝叶斯网络(dynamic Bayesian network,DBN)是在静态贝叶斯网络的基础上,引入了时间维而形成的动态网络,动态贝叶斯网络能够学习随机变量间的概率依存关系及其随时间变化的规律,并以图的方式直观地反映这种关系,由于微阵列数据的时间特性,动态贝叶斯网络可以更精确地描述基因调控网络,所以基于动态贝叶斯网络从时间序列微阵列数据中构建基因调控网络成为当前的一个研究热点.目前,已有许多基于动态贝叶斯网络构建基因调控网络的相关算法[2-5],但这些算法都需要将连续的基因表达数据离散化,没有充分利用数据所包含的信息.
笔者提出了一个基于动态贝叶斯网络构建基因调控网络的算法,该算法基于时序互信息来学习动态贝叶斯网络,在计算基因间的时序互信息时,该算法考虑了时间序列微阵列数据的时间特性,并利用协方差矩阵计算互信息,没有将基因表达数据离散化,与基因表达数据的连续性相符合,减少了信息的损失.
1 动态贝叶斯网络及相关研究
1.1 动态贝叶斯网络
动态贝叶斯网络是贝叶斯网络的扩展,它包含随时间变化的隐含的状态,使隐含的随机进程的熵的比率最大化,它是有向有环图.设一个时间序列X ∈ Rs×n,s为时间点的数目,n为基因的数目.(t)表示基因i关于时间t的表达水平的变量,在时刻t,n个基因表达水平的向量为 X (t) = (X1(t) , … ,Xn(t ))T,基因 i在 s个时间点上表达水平的向量为 Xi=(Xi(1),… , Xi(s ))T.
动态贝叶斯网络假定:①网络中的有向弧随着时间的变化而展开,即假定动态贝叶斯网络是一阶马尔科夫的,基因 i在 t时刻的表达水平只依赖于它的父节点在t-1时刻的表达水平,而与 t-1时刻前的表达状态完全独立;②基因之间的动态因果关系在所有的时间点上都没有变化,即随机变量的集合以及相应的概率转移函数,对每个时间点来说都是相同的.根据以上假设,网络的条件概率分布可表示为

式中 P ai(t − 1 )为基因 Xi(t)的父节点.一个典型的动态贝叶斯网络如图1所示.
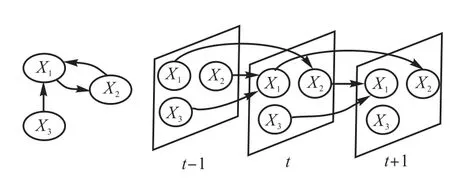
图1 一个简单的动态贝叶斯网络Fig.1 A simple dynamic Bayesian network
从数据中学习动态贝叶斯网络分为结构学习和参数学习两个过程.给定一个时间序列的数据集D = ( X ( 1),… ,X (n)),从 D中学习动态贝叶斯网络结构就是找到一个与 D最佳匹配的结构 G = ( V,E),其中V = { X1,… ,Xn}为顶点的集合,Xi表示基因 i,E = { (Xi(t − 1 ),Xj(t) ) |Xi(t − 1 ) → Xj(t)}为 一 组 有 向 弧的集合,Xi(t − 1 ) → Xj(t )表示 t-1时刻的基因 i对 t时刻的基因 j有调控关系,即基因 i在 t-1时刻的表达水平影响了基因j在t时刻的表达水平.研究人员对用动态贝叶斯网络从时间序列基因数据中构建基因调控网络的模型做了大量的研究工作.
1.2 相关研究
Wu等[2]讨论了利用 BDE(Bayesian dirichlet equivalence)打分函数来构建动态贝叶斯网络的贪婪的爬山搜索算法.给定一个时间序列的数据集D = ( X ( 1),… ,X (n)),从 D中构建动态贝叶斯网络就是发现一个与D最匹配的模型 M =(G,Θ),G是网络结构,Θ是条件概的集合.BDE打分函数的方法对父节点的数目比较敏感,容易陷入局部最优,计算复杂性指数级 O (μ2N),N为网络中节点的数目,μ为指定的父节点的上界.计算复杂度高,而且这种方法需要将基因表达数据离散化.
Wu等[2]还讨论了利用 MCMC(Markov chain Monte Carlo)方法构建动态贝叶斯网络的 MH(metropolis hastings)算法.如果一个过程的“将来”仅依赖“现在”而不依赖“过去”,则此过程具有马尔可夫性,或称此过程为马尔可夫过程,X(t)=f(X(t-1)).时间和状态都离散的马尔科夫过程称为马尔科夫链.设 S是一个可数集,{ X (t),t = 1 ,2,… ,s } 是一组随机变量的集合,如果满足 P (X (t)| X (t − 1),… ,X (1))=P(X(t) |X (t − 1 )),则称{ X (t),t = 1 ,2,… ,s }为一个马尔科夫链.对于一个马尔科夫链,如果 P (X(t)|X(t −1))与 t的取值无关,则称这个马尔科夫链为齐氏马尔科夫链.动态贝叶斯网络的转移网络就是一个齐氏的马尔科夫链.
这种方法也需要将基因表达数据离散化,运算时间复杂度高,即使在数据充分的情况下,学习的结果也不是很理想.
Zhu等[3]根据生物学的细胞活化信号路径等先验知识,在基于贝叶斯网络构建基因调控网络时,缩小了搜索空间,降低了时间复杂度,扩大了构建基因调控网络的规模
Wang等[4]针对趋势相关(两基因在其表达水平随时间上升与下降的变化趋势上相关)关系在重建基因调控网络中十分重要却尚未被挖掘利用的问题,提出了几何模式动态贝叶斯网络(Gp-DBN)方法.Gp-DBN将每个基因的表达数据转换为一个几何模式,依据几何模式确定潜在的调控子和调控时滞,并通过推理这些几何模式之间的相关关系来发现基因间的调控关系.该方法解决了挖掘具有趋势相关的基因调控关系的问题,能够很大程度地提高重建的基因调控网络的性能.
Liu等[5]指出在基因调控网络中不仅单个蛋白质对基因具有调控作用,而且一些蛋白质的组合对基因也具有调控作用,Liu把这些蛋白质的组合看作基因调控网络中的隐藏变量.Liu提出了一个Semi-fixed模式将基因调控网络表示为具有隐藏变量的贝叶斯网络,并用EM算法构造Semi-fixed模式的基因调控网络.
Dojer等[6]提出一种从基因扰动实验的数据中构建动态贝叶斯网络的算法.在构建动态贝叶斯网络前,作者先用 Husmeier离散化方法对基因表达数据做了预处理,在构建贝叶斯网络时,作者使用 BDE打分函数,并用从扰动实验的数据集中独立计算每个节点(基因)的父节点集的方法代替启发式搜索的方法,减少了学习时间,减少了启发式搜索的方法带来的时间复杂性.
以上的基于动态贝叶斯网络构建基因调控网络的算法均需对基因表达数据离散化,而时序的微阵列数据是连续的,从而不能充分利用数据信息.这里笔者提出了TSMI-GRN(time series mutual informationgene regulation network)算法,该算法基于时序互信息构建基因调控网络,所构建的网络为动态贝叶斯网络,TSMI-GRN算法不需要对基因表达数据离散化,减少了数据信息的损失.
2 TSMI-GRN算法
2.1 时序互信息
设D为一个时间序列基因表达数据集,包含n个基因{1,2,…,n},s个时间点,Xi(t)表示基因 i关于时间 t的表达水平的变量,这里要在 D上用 TSMIGRN算法构建这 n个基因的调控网络.设为一多维平稳时间序列 .
要在数据 D上构建动态贝叶斯网络 G = ( V,E),V = { 1,2,… ,n },为 n个基因的集合,E= {(Xi(t −1),Xj(t) ) |Xi(t − 1) → Xj(t),i = 1,… ,n, j = 1,… ,n}.在 构 建网络时,需要研究 Xi(t − 1 )与 Xj(t)之间是否存在关系,即考虑t-1时刻基因i的表达水平对t时刻基因j的表达水平的影响,根据动态贝叶斯网络的假设,t时刻的状态只与 t-1时刻的状态有关,这里考虑在 t-1时刻除去i与j两个基因外所有基因的表达水平的条件下,来研究 Xj(t)和 Xi(t − 1 )之间的条件独立性,这里给出t时刻基因j与t-1时刻基因i的时序互信息的定义.
定义1 设 Yij( t − 1)= { Xk(t − 1 ),k ≠ i ,j} ,Xj(t)和Xi(t − 1 )在 Yij(t − 1 )条件下的条件互信息为
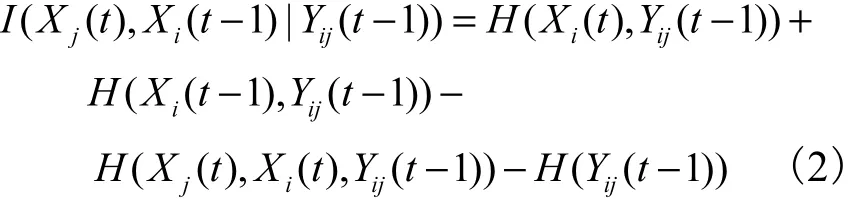
对于给定的阈值ε>0,如果I(Xj( t),Xi(t−1)|Yij(t −1))<ε,称基因j与i相互独立,否则称j与i是相关的.
在式(2)中,令

则式(2)可简化为

这里假定随机变量 X1,… ,Xn的联合分布是n维高斯分布,线性熵 Hl( X1, X2,… ,Xn)和熵H(X1,X2,… ,Xn)在理论上是等价的.线性熵
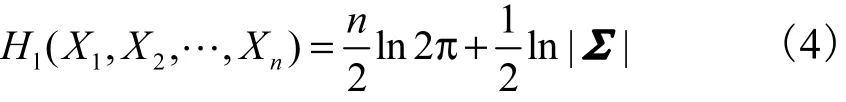
式中:Σ为 n维随机变量 ( X1, X2,… ,Xn)的协方差矩阵.
用 Σ、Σi、 Σj和 Σij分 别 表 示 前 面 所 构 造 变 量Z(t) 、 Zi(t) 、 Zj(t) 和 Zij(t)的协方差矩阵,根据式(3)和式(4),给定 Yij(t − 1 )的条件下,Xj(t)和 Xi(t − 1 )之间的条件互信息为

2.2 TSMI-GRN算法
定义 2定义了基于时间序列互信息所构建的网络结构.
定义2 设X(t) = { X (t) , X (t) , … ,X (t) }T(t ∈Z)
1 2n为一多维平稳时间序列,图 G = ( V,E),对应的顶点集V ={1,2,… ,n},对 于 给 定 ε>0,如 果 Il(Xj(t),Xi(t − 1 )|Yij(t − 1 ))> ε ,则(Xi(t − 1 ) → Xj(t) ) ∈ E ,如 果Il(Xj( t),Xi( t − 1 )|Yij(t − 1 ))< ε ,则(Xi(t − 1 ) → Xj(t ))∉E.
根据时序互信息和图结构的定义,提出了基于TSML-GRN算法从单个基因表达数据集中构建基因调控网络.TSML-GRN算法描述如下.
步骤 1 对于每个基因 j,计算 Il(Xj( t),Xj(t −1)|Yj(t − 1 ), j = 1 ,… ,n ).如果Il(Xj( t),Xj( t − 1 )|Yj(t − 1))>ε,则(Xj(t − 1 ) → Xj(t) ) ∈ E ,表明t-1时刻的基因j的表达水平对t时刻的基因j的表达水平具有调控作用.
步骤2 对于给定的基因i和j,i≠j,计算Il(Xj( t),Xi( t − 1 )|Yij(t − 1 )),其中 j = 1 ,2,…, n ,i = 1,2,… ,n.如 果 Il(Xj( t),Xi( t− 1 )|Yij(t− 1))> ε ,则(Xi(t− 1 )→ Xj(t ))∈E,表明 t-1时刻基因 i的表达水平对 t时刻基因 j的表达水平具有调控作用.计算 Il(Xi( t),Xj(t −1)|Yij(t − 1 )),如 果 Il(Xi( t),Xj(t − 1 )|Yij(t −1))>ε ,则(Xj(t − 1 ) → Xi(t) ) ∈ E ,表明t-1时刻基因j的表达水平对t时刻基因i的表达水平具有调控作用.
步骤3 根据步骤1与步骤2,构建了一个初始的动态贝叶斯网络结构 G = ( V,E),由于基因调控网络具有稀疏性[7],再利用一个打分函数对网络结构G进行优化.设定每个节点的父节点的上界μ=3,对于G中父节点数目大于3的节点,选取所有小于等于3的父节点的非空子集,选取使打分函数I(X , subsetpa(X ) /max{ H (X ) ,H(subsetpa(X ) )}[8]为最大分数的子集作为该节点父节点集,得到最终的网络结构.
在上述的TSML-GRN算法中,步骤1计算的是t-1时刻基因j与t时刻基因j的网络关系,步骤2计算的是t-1时刻基因i与t时刻基因j,以及t-1时刻基因 j与 t时刻基因 i的网络关系.经过步骤1和步骤2的计算,基于时序互信息构建了一个初始的基因调控网络,但这个网络中每个节点的父节点的数目较大,而基因调控网络具有稀疏性,所以步骤3引入了一个打分函数来优化步骤1与步骤 2构建的初始网络结构,得到最终的网络结构.下面在真实的基因表达数据集上测试 TSMI-GRN算法,根据实验结果,对算法的有效性进行分析.
3 实验分析
在酵母菌周期细胞微阵列数据集 Cdc15上测试TSML-GRN算法,该数据集从斯坦福微阵列数据库(http://smd/stanford.edu/)中获得.这里选择了数据集Cdc15分别包含 14个基因和 25个基因的两个子集进行测试.测试结果与基因组数据库 KEGG中描述的通过实验得到的酵母菌周期细胞中一些基因的调控网络(http://www.genome.jp/dbget-bin/show_ pathway?dce04111)相比较,来分析实验结果.在 TSMIGRN算法中,假定了基因表达数据满足正态分布.利用 SAS软件中的 Kolmogorov-Smirnov方法对实验数据进行了是否为高斯分布的检验,检验结果表明基因表达水平数据大致服从对数正态分布,证明这一假设是合理的.在实验中,分别选取了不同的阈值ε为0.2、0.3、0.5、0.6、0.8 和 1.0 对 TSMI-GRN 进行了测试.将测试结果与 KEGG数据库中已有的结果进行比较,发现0.5ε=时的实验结果与KEGG数据库中的结果最接近.图 2比较了选取不同阈值时网络的准确度.
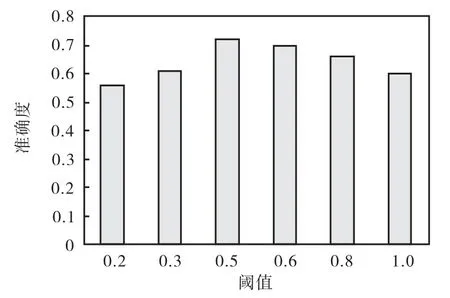
图2 不同阈值下构建网络的准确度Fig.2 Accuracy of the constructed network with different thresholds
在基因调控网络的结构学习过程中,为了定量评价网络构建的准确性,使用灵敏度(sensitivity)和特异度(specificity)作为评价指标.为此,需要首先计算以下4个值.
(1)真阳性TP(true positive):构建的网络中正确的边数.
(2)真阴性 TN(true negative):构建的网络中正确的不存在的边数.
(3)假阳性 FP(false positive):构建的网络中错误的边数.
(4)假阴性 FN(false negative):构建的网络中错误的不存在的边数.
由此,可以定义灵敏度和特异度的计算式为

显然,灵敏度和特异度的值越高,网络的构建结果就越好.
图 3为在含有 14个基因、24个时间点的微阵列数据集上用 TSML-GRN算法构建的基因调控网络.与KEGG数据库中的网络相比,本文构造的网络中有11条正确的边,4条添加的边.
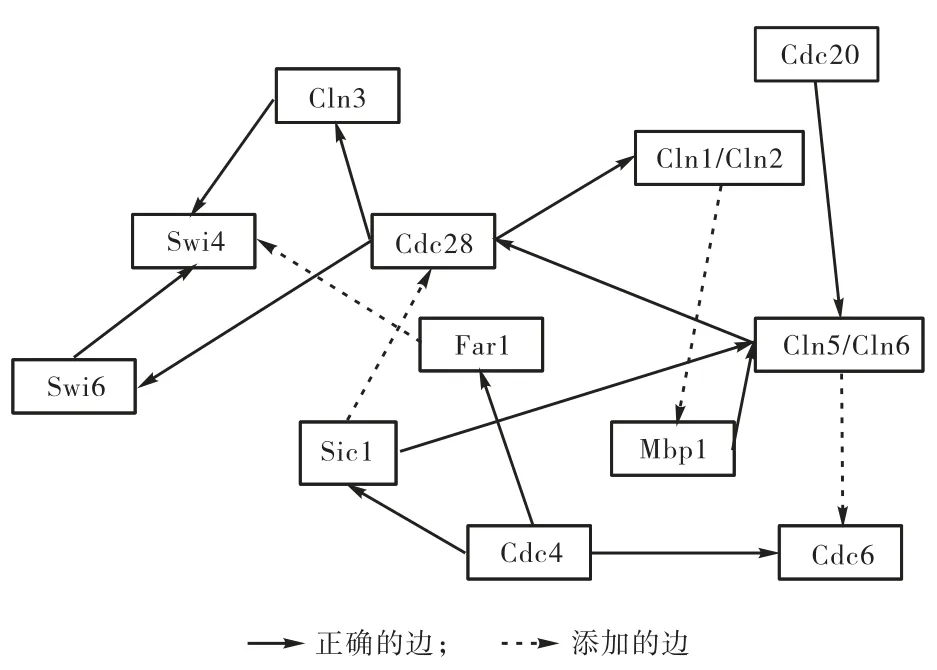
图3 TSMI-GRN算法构建的14个基因调控网络Fig.3 Regulation network containing fourteen genes constructed with TSMI-GRN algorithm
本文还在上述数据集上测试了 MH算法[2]和BDE打分爬山搜索算法[2],表1比较了3种算法构建的基因调控网络的灵敏度与特异度.

表1 灵敏度与特异度Tab.1 Sensitivity and specificity %
根据表 1,可以看出基于时序数据的互信息算法的灵敏度高于其他两种算法,说明 TSMI-GRN 算法能够更准确地构建基因调控网络.
图4为在酵母菌周期细胞的实验数据上,测试TSMI-GRN算法构建的基因调控网络.选取的实验数据集含有 25个基因、24个时间点.运用 TSMI-GRN算法在该数据集上构建的基因调控网络,与数据库KEGG中的网络相比,其中有14条正确的边,6条方向相反的边,8条添加的边.在8条添加的边中,发现Cdc28与 Cdc20之间的调控关系在文献[9]实验中被发现,Chk1与 Rad9之间的调控关系在文献[10]实验中被发现,对于学习到的其他反向边与添加的边,对生物学实验具有一定的指导作用.以上的实验结果表明,TSMI-GRN算法是有效的.

图4 TSMI-GRN算法构建的包含25个基因的调控网络Fig.4 Regulation network containing twenty five genes constructed with TSMI-GRN algorithm
4 结 语
综合前面的实验结果与分析可见,TSMI-GRN算法可以从时间序列微阵列数据中更准确地构建基因调控网络.该算法的优点为:①不需要将基因表达数据离散化,与基因表达数据的连续性相符合,因而可以更准确地构建基因调控网络;②在计算两个基因的互信息时,使用时序数据的互信息,与传统的互信息比较,增加了时间的特性,与时间序列的数据更吻合;③TSMI-GRN算法利用协方差矩阵计算两个基因间时序互信息,并且考虑了其他所有基因对这两个基因间互信息的影响;④实验结果证明,时序互信息算法的灵敏度高于MH算法以及BDE打分爬山搜索算法的灵敏度;⑤TSMI-GRN算法发现了 Cdc28与Cdc20、Chk1与Rad9之间的调控关系,这在一些生物学实验中得到了验证.因而 TSMI-GRN算法是一种有效的算法,在构建基因调控网络中发挥积极作用.
经过实验验证,TSMI-GRN算法是一种有效的算法,但 TSMI-GRN算法有一定的局限性,它假定微阵列数据集服从高斯分布,计算两个基因间的互信息时用线性熵来近似代替信息熵.
[1] 刘万霖,李 栋,朱云平,等.基于微阵列数据构建基因调控网络[J].遗传,2007,29(12):1434-1442.
Liu Wanlin,Li Dong,Zhu Yunping,et al. Constructing gene regulation network from microarray data[J]. Hereditas,2007,29(12):1434-1442(in Chinese).
[2] Wu Huihai,Liu Xiaohui. Dynamic Bayesian networks modeling for inferring genetic regulatory networks by search strategy:Comparison between greedy hill climbing and MCMC methods[J]. International Journal of Computational Intellgence,2009,5(2):189-199.
[3] Zhu Dongxiao,Li Hua. Improved Bayesian network inference using relaxed gene ordering[J]. International Journal of Data Mining and Bioinformatics,2010,4(1):44-59.
[4] 王开军,张军英,赵 峰,等.几何模式动态贝叶斯网络推理基因调控网络[J].西安电子科技大学学报:自然科学版,2007,34(6):922-925.
Wang Kaijun,Zhang Junying,Zhao Feng,et al. Geometric-pattern dynamic Bayesian networks reasoning gene regulatory networks[J]. Journal of Xidian University:Natural Science,2007,34(6):922-925(in Chinese).
[5] Liu Tiefei,Sung Wingkin,Mittal Ankush. Model gene network by semi-fixed Bayesian network [J]. Expert Systems with Applications,2006,30(1):42-49.
[6] Dojer N,Gambin A. Mizera A,et al. Applying dynamic Bayesian networks to perturbed gene expression data[J].BMC Bioinformatics,2006,7:249-259.
[7] Chan Z S H, Collins L, Kasaboy N. Bayesian learning of sparse gene regulatory networks[J]. Biosystems,2007,87(2/3):299-306.
[8] Liang S,Fuhrman S,Somogyi R. Reveal,a general reverse engineering algorithm for inference of genetic network architectures[J]. Pacific Symposium on Biocomputing,1998,3:18-29.
[9] Rudner A D,Murray A W. Phosphorylation by Cdc28 activates the Cdc20-dependent activity of the anaphasepromoting complex[J]. The Journal of Cell Biology,2000,149(7):1377-1390.
[10] Loegering D,Arlander S J. Rad9 protects cells from topoisomerase poison-induced cell death[J]. The Journal of Biological Chemistry,2004,279(18):18641-18647.

