蛋白质组学在去泛素化酶研究中的应用
李衍常,高媛,徐忠伟,兰秋艳,2,徐平,2
1 军事医学科学院放射与辐射医学研究所 蛋白质组学国家重点实验室 国家蛋白质科学中心 (北京) 北京蛋白质组研究中心 蛋白质药物国家工程研究中心,北京 102206
2 武汉大学药学院 组合生物合成与新药发现教育部重点实验室,湖北 武汉 430072
蛋白质组学在去泛素化酶研究中的应用
李衍常1,高媛1,徐忠伟1,兰秋艳1,2,徐平1,2
1 军事医学科学院放射与辐射医学研究所 蛋白质组学国家重点实验室 国家蛋白质科学中心 (北京) 北京蛋白质组研究中心 蛋白质药物国家工程研究中心,北京 102206
2 武汉大学药学院 组合生物合成与新药发现教育部重点实验室,湖北 武汉 430072
李衍常, 高媛, 徐忠伟, 等. 蛋白质组学在去泛素化酶研究中的应用. 生物工程学报, 2014, 30(9): 1341−1350.
Li YC, Gao Y, Xu ZW, et al. Application of proteomics in deubiquitinases research. Chin J Biotech, 2014, 30(9): 1341−1350.
泛素-蛋白酶体系统是细胞内蛋白质特异性降解的主要途径,参与并调控细胞周期、免疫应答、信号传递和DNA修复等真核生物体内几乎所有的生命活动。去泛素酶的存在使泛素化修饰成为可逆过程,保证了泛素系统及其相关生理过程的动态平衡,其表达紊乱也是诱发多种疾病的主要原因。对去泛素化酶进行系统、全面的研究是理解其作用机制并将其作为治疗药物靶点的前提。蛋白质组学技术的快速发展为系统深入研究去泛素化酶提供了条件,特别是在去泛素化酶的相互作用网络和底物特异性研究等方面发挥了独特的作用。因此,文中结合课题组研究工作,对去泛素化酶的分类及功能进行介绍并总结了蛋白质组学在去泛素化酶研究中的应用进展。
泛素-蛋白酶体系统,去泛素化酶,蛋白质组学,蛋白质相互作用,质谱
泛素(Ubiquitin, Ub)是一种含76个氨基酸,序列高度保守且广泛存在于真核生物中的小分子蛋白[1-3]。泛素-蛋白酶体系统 (Ubiquitinproteasome system, UPS) 作为真核生物内特异、高效并依赖能量的蛋白质降解途径,参与了细胞周期调控、免疫应答、信号传递和DNA修复等众多重要的生命活动[4-7]。2004年,以色列科学家Aaron Ciechanover、Avram Hershko和美国科学家Irwin Rose因发现泛素介导的蛋白质降解途径而获得诺贝尔化学奖。
泛素修饰是可逆过程,去泛素化酶(Deubiquitinase, DUB) 的存在保证了UPS及其相关生理过程的动态平衡稳定[4,8-11]。目前已知酵母存在约20种DUB,人类存在约100种DUB。根据结构和催化机制,DUB可分为5大类,参与了泛素生成、修剪、回收和底物保护等多个过程[8]。DUB底物特异性主要表现为泛素链形式 (Met1-linear fusion,K6、K11、K27、K29、K33、K48和K63)[12]的特异性和底物蛋白的特异性。如何系统研究众多DUB的特异性及其调控规律已经成为该领域的热点,也是难点。由于DUB是导致多种疾病的关键靶点,因此开发DUB特异性的抑制剂来调控其在代谢通路中的功能为药物开发提供了新思路。
目前,以质谱技术(Mass spectrometry,MS)为代表的蛋白质组学 (Proteomics) 在系统揭示生命过程和解释基因表达调控分子机制中发挥着重要作用[13-15]。随着高分辨率质谱的迅猛发展,蛋白质组学深度测序和精确定量技术在UPS研究中展现出独特的优势并呈现出良好的发展势头。2010年国际首届蛋白质组学与泛素相关会议PPDUP (Proteomics of protein degradationand ubiquitin pathways) 在加拿大温哥华举行[16-17]。同样,蛋白质组学在DUB的相互作用网络研究和抑制剂的开发等方面也取得了众多成果。因此,本文将结合实验室的研究工作,对DUB进行简要介绍并总结蛋白质组学在DUB研究中的应用进展。
1 去泛素化酶介绍
1.1 去泛素化酶的分类
根据催化机制,蛋白酶 (Protease) 包含天冬氨酸、丝氨酸、苏氨酸、半胱氨酸和金属蛋白酶5大类[18]。DUB也属于蛋白酶,主要包括半胱氨酸蛋白酶和金属蛋白酶2种。目前从酵母和人类中分离鉴定出了多种DUB,基于其结构相似性和可能的作用机制,主要分为泛素羧基末端水解酶(Ubiquitin C-terminal hydrolase, UCH)、泛素特异性蛋白酶 (Ubiquitin-specific protease,USP)、卵巢肿瘤蛋白酶(Otubain protease,OTU)、Machado-Joseph disease蛋白酶 (MJD) 和MPN(+)/JAMM蛋白酶 (JAMM)五种类型,其中前4种属于半胱氨酸蛋白酶,JAMM为金属类蛋白酶。
1.1.1 泛素羧基末端水解酶 (Ubiquitin C-terminal hydrolase, UCH)
UCH是最早被发现和表征的DUB。已知酵母中为YUH1,人类中的4个DUB分别是UCHL1、UCHL3、UCHL5和BAP1。UCH通过裂解C末端76位甘氨酸,将泛素分子从底物蛋白上释放出来。研究显示该类DUB底物偏好分子量较小的底物蛋白 (20–30氨基酸)[9]。UCH活性位点上的狭窄裂隙和环状结构直径的限制在一定程度上起到了特异性识别小分子底物的功能[19]。UCH主要功能是将未成熟的串联泛素前体或者泛素-核糖体蛋白复合物进行修剪得到成熟的泛素。文献报道YUH1不仅在泛素生成过程中发挥作用,还特异性地对类泛素NEDD8/RUB1的生成起调控作用[20]。UCHL1特异性存在于神经元中,其活性与神经性疾病帕金森症 (Parkinson’s disease,PD) 有关[21-23]。
1.1.2 泛素特异性蛋白酶 (Ubiquitin-specific protease,USP)
USP家族是目前已知的去泛素化酶中成员最多且结构最具多样性的一类。已知酵母中为16个,人类约为60个USP。这些酶分子都含有两个短而保守的催化片段,即半胱氨酸盒和组氨酸盒,其中的催化三联残基 (半胱氨酸、组氨酸、天冬氨酸) 能将泛素分子从底物蛋白上移除[24]。研究显示在进化进程上USP和E3的数目呈现出一致性增加,提示两者在功能上存在一定的联系[23]。该类DUB中研究较多的如USP7/HAUSP、USP2和USP28等。USP7通过对E3酶MDM2和p53的去泛素化作用,使p53处于动态平衡,进而发挥抑癌作用[19,25-27]。USP28在结肠癌患者与乳腺癌患者体内过量表达,它通过抑制SCF-Fbxw7连接酶的泛素化活性来稳定cyclin E1蛋白和c-Myc蛋白[28]。
1.1.3 卵巢肿瘤蛋白酶 (Otubain protease,OTU)
OTU是根据生物信息学预测,最早在果蝇卵巢内发现的一类DUB[29]。酵母中有2个OTU,分别为OTU1和OTU2;人类中约有14个OTU。该家族也具有由三联催化活性位点组成的核心结构域,与UBP家族蛋白在结构上有很大相似性,并且已证明能起到去泛素化的作用。该类DUB中研究较多的有Cezanne、A20和VCIP135等。它们分别参与了NF-κB信号通路调控、有丝分裂中高尔基体重组等重要的生命过程[29]。
1.1.4 Machado-Joseph disease蛋白酶 (MJD)
MJD同样是基于生物信息学预测鉴定的,含有一个称为Josephin结构域的DUB[30]。目前有去泛素化酶活性的MJD只有Ataxin-3,而其他经预测发现的可能的MJD也含有保守的催化核心,但实验发现催化活性很低。在酵母内还没有MJD的报道。目前已发现Ataxin-3可以水解泛素化溶菌酶,还可以与去泛素化酶抑制剂Ubal结合[30-31]。
1.1.5 MPN(+)/JAMM蛋白酶 (JAMM)
JAMMs是金属类蛋白酶。酵母的RPN11和哺乳动物细胞内的POH1为其典型代表,都是构成26S蛋白酶体的重要组成部分[9,32]。其催化中心包括两个保守的组氨酸残基和天冬氨酸残基以及二价锌离子。其他重要的JAMM还有AMSH和BRCC36等,主要参与调控胞吞和细胞周期检测点等过程[33-34]。
1.2 去泛素化酶功能介绍
DUB利用其催化核心表面的亲核基团 (半胱氨酸) 来攻击泛素的羧基末端,通过裂解切断泛素链与底物蛋白之间的连接或者泛素链之间的连接,使泛素分子从泛素化蛋白底物上或蛋白酶体降解底物中脱离出来,重新进入细胞内蛋白调节的循环中。
1.2.1 去泛素化酶调控过程
DUB参与了UPS发生反应的的各个环节,包括:①泛素前体处理;②回收错误连接到亲核试剂如谷胱甘肽上的泛素;③编辑或拯救待降解的泛素化蛋白;④拆卸非锚定的游离泛素链,回收泛素。具体过程如图1所示[8]。
上述每个过程都有多种特定的DUB参与,这体现了其分工明确又互相协调的作用特点。在泛素前体加工和成熟过程中,UCH类的DUB发挥了重要作用[8,21]。在编辑或者拯救待降解蛋白方面,大量的DUB特异性地参与了多种代谢通路,体现出了DUB对泛素链或者底物的特异性。如USP7特异性地参与了p53和MDM2 (p53的E3) 的去泛素化过程,以阴阳协调的方式来实现对p53的调控[10,25]。对于游离的非锚定泛素链的回收,Wilkinson研究团队[7,35]系统揭示了IsoT (Isopeptidase)在该过程的作用。在酵母中与IsoT同源的DUB是UBP14,我们的研究结果同样显示UBP14对游离的泛素链的特异性现象,同时发现UBP14对不同链接形式存在一定的偏好性 (另文发表)。
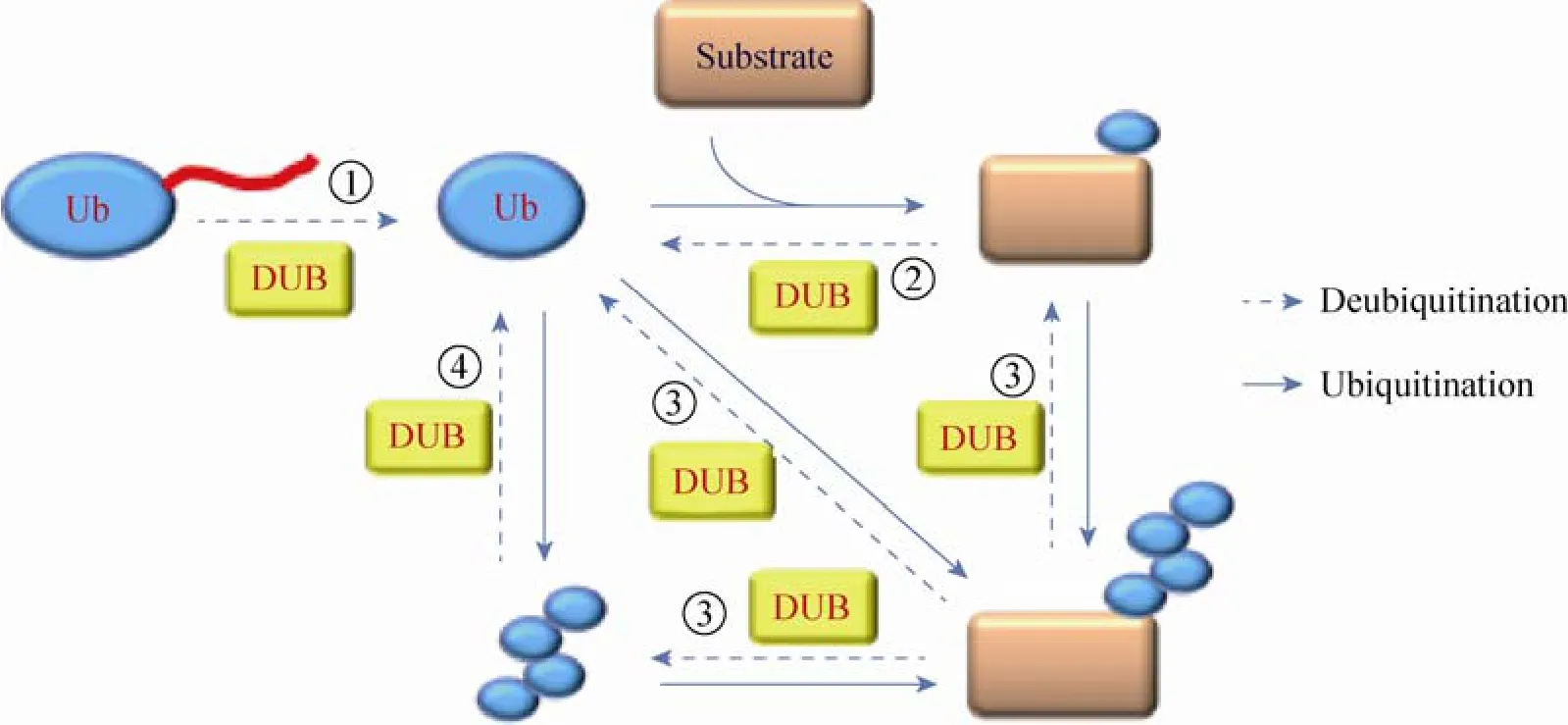
图1 去泛素化酶参与泛素加工的过程[8]Fig. 1 DUB involved in the ubiquition processing[8].
1.2.2 去泛素化酶与疾病关联
由于UPS参与调控了细胞内多种生命过程,其表达紊乱会引起人体内的多种病理改变,比如癌症、神经退行性病变以及免疫功能紊乱等。由于DUB参与了泛素降解过程的各个环节,作为致癌或抑癌E3连接酶的直接抑制物,同样也可以作为抗癌治疗的作用靶点。目前研究发现DUB参与并调控了众多疾病,如表1所示。
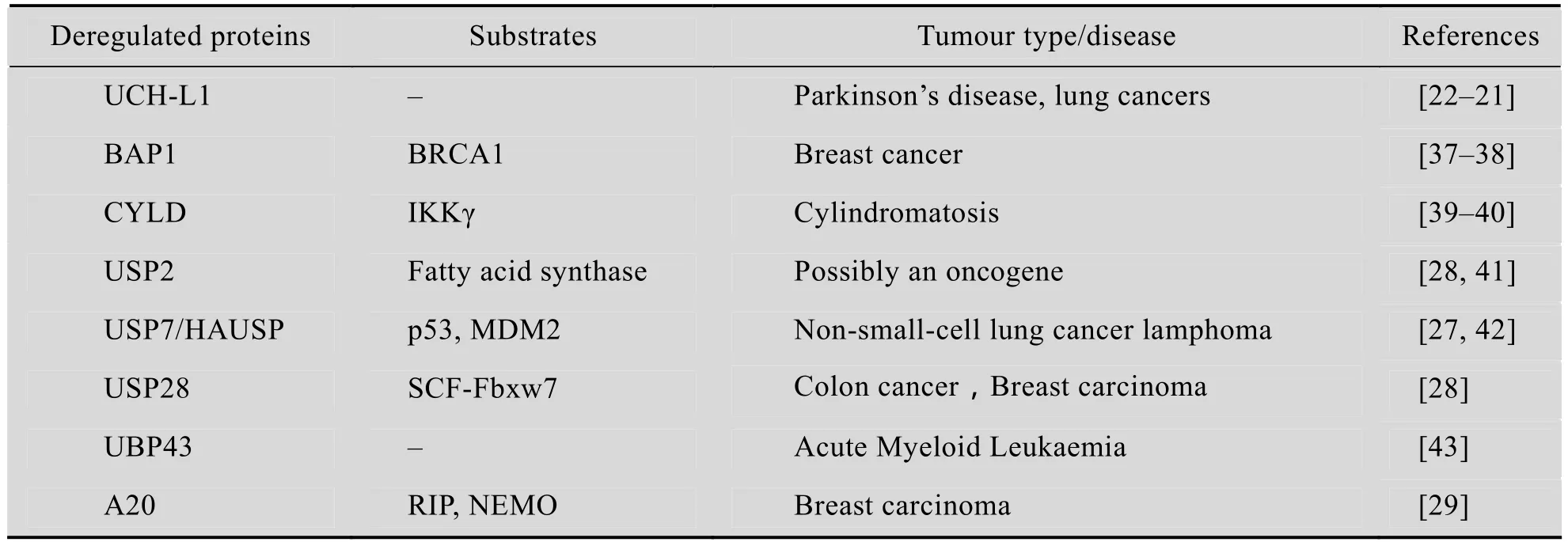
表1 去泛素化酶与疾病之间的关系Table 1 The relationship between DUB and diseases
目前,以UPS内的多种酶 (主要是E3、DUB和Proteasome) 为靶点,开发了一系列的诊断及治疗方法来检测或治疗肿瘤。比如已经成功上市的蛋白酶体抑制剂硼替佐米 (Bortezomib,商品名Velcade),可以用来治疗多发性骨髓瘤等血液系统肿瘤。其作用机制是可逆性地抑制26S蛋白酶体活性位点,阻止NF-κB抑制剂被蛋白酶体降解来发挥抗癌作用[36]。Velcade的成功带动了一大批蛋白酶体抑制剂的研发,突出的例子有PR-171、NPI-0052和CEP-18770等[44]。同样,对UPS起重要调控作用的DUB,也可以作为众多疾病的治疗靶点。因此,开发针对不同DUB的特异性抑制剂可能是研究信号途径调控和疾病分子机制的重要手段。如果能够在整体水平上系统研究DUB的作用机制、底物特异性以及生物学效应,将会为我们选择特定的DUB进行特异性抑制剂的筛选研究提供佐证。
2 蛋白质组学在去泛素化酶研究中的应用
经过近20年的发展,蛋白质组学已经成为生命科学研究中的重要力量。目前,对蛋白质组的深度测序、精确定量以及生物学功能研究已经成为蛋白质组学研究发展的主流[13,15]。高效液相色谱的分辨能力和高精度质谱的快速鉴定能力极大地促进了蛋白质组学的发展[13,45-48],各种定量策略的开发提高了相对定量和绝对定量的应用范围和定量精度[46-47]。同时,有效的蛋白质翻译后修饰富集策略为蛋白质组的功能研究打下坚实的基础[49-50,54]。
蛋白质组学在UPS的位点鉴定、泛素链修饰形式以及底物特异的研究中发挥着重要的作用。泛素亲和结构域 (Ubiquitin binding domain,UBD)[50-52]和K-εGG抗体[53-54]的开发使泛素化位点的鉴定突飞猛进。利用质谱选择反应监测技术 (Selected-reaction monitoring,SRM) 实现了对不同泛素链连接形式的鉴定与绝对定量[12,55]。此外,蛋白质组学在泛素相关研究中的应用还包括相互作用蛋白的鉴定和特定通路中泛素化底物的筛选等诸多方面[49-54]。
2.1 去泛素化酶的相互作用组
2009年,Sowa等[56]在Cell杂志上发表了系统研究人类75个DUB的功能和相互作用蛋白鉴定的工作。作者将75个DUB分别构建到带有FLAG-HA (血凝素) 标记的质粒上,然后转染到293T细胞内,在非变性条件下亲和纯化,利用LC-MS/MS技术系统分析鉴定不同DUB的相互作用蛋白。为了得到高可信的相互作用蛋白,他们还开发了生物信息软件ComPASS来区分非特异性背景蛋白或者污染物。该研究鉴定到了774个高可信的相互作用蛋白质。通过对鉴定的相互作用蛋白质的功能分析,发现DUB及其相互作用蛋白参与了蛋白质降解、转录调控、DNA修复和内质网介导的降解等生物过程。该工作利用蛋白质组学的方法第一次系统地揭示了DUB在人体内的系统分布和相互作用关系,为后续深入研究DUB的功能及其调控网络提供了依据和方向。
除此之外,利用蛋白质组学技术鉴定DUB相互作用蛋白的研究还有很多[57-61]。Xie等[57]利用iTRAQ (Isobaric tags for relative and absolute quantitation) 试剂分别标记SMURF1正常组和过表达组,定量比较分析SMURF1相关的蛋白复合体,发现了与其有密切相互作用的去泛素化酶FAM/USP9X。该去泛素化酶能够拮抗SMURF1自身的泛素化修饰从而保护SMURF1不被蛋白酶体降解。Gomez-Ferreria等[58]利用亲和纯化技术得到CEP192相互作用蛋白,通过在线液质联用技术鉴定到其与K63特异的去泛素化酶CYLD存在相互作用。在敲除CYLD和CEP192的细胞中都发现了有丝分裂的纺锤丝生成受抑制的现象,提示了CEP192通过去泛素化酶CYLD在纺锤丝合成中的重要作用。
2.2 去泛素化酶底物特异性鉴定
DUB底物的特异性主要表现在泛素链形式的特异性和底物蛋白的特异性,亦或两者的结合[7]。质谱技术在系统鉴定蛋白和识别各种泛素链连接形式具有独特优势,因此蛋白质组学为系统研究DUB与底物之间的特异性关系提供了条件。
Mevissen等[59]在Cell杂志上发表了系统评估16个OTU家族DUB与泛素链之间的特异性关系的工作。通过结合质谱鉴定泛素链组成的数据(未发表,E. Bennett,University of California, San Diego),利用体外反应实验证明了OTU家族与泛素链之间的特异性关系。他们发现OTU结构域上S1’和S2泛素结合位点决定了OTU家族区分不同泛素链形式的特异性。Fiil等[60]利用SILAC (Stable isotope labeling by amino acids in cell culture) 定量策略,亲和纯化Met1-ub相关蛋白,实验揭示了OTU家族去泛素化酶抑制泛素线性连接形式 (Met1-Ub) 的聚集,同时通过亲和纯化线性泛素链鉴定到RIPK2是NOD2 (Nucleotideoligomerization domain-containing protein 2) 相关的主要底物,证明了OTU去泛素化酶能够抑制Met1-Ub在RIPK2的聚集,从而证实了OTU家族去泛素化酶通过去除底物NOD2上的线性泛素连接形式来调控免疫反应,并抵抗炎症信号。同样,我们利用SILAC定量技术和质谱SRM技术系统研究酵母内不同DUB对泛素链形式的特异性,也发现该DUB对不同链接形式存在一定的偏好性 (另文发表)。
除了对泛素链形式的特异性选择外,特定代谢通路中DUB特异性底物的鉴定也同样可以利用蛋白质组学技术进行研究。如Debelyy等[61]利用凝胶电泳分离和质谱鉴定技术分析了过氧化物酶体的组成,发现去泛素化酶UBP15能够特异性与其发生相互作用。通过体外验证实验证明了UBP15能够特异性识别PEX5 (Peroxisomal targeting sequence receptor, Pex5p),并在PEX5从过氧化物酶体重新返回细胞质的过程中发生去泛素化作用。本实验室同样开展了利用LC-MS/MS质谱平台系统鉴定DUB底物的工作,试图通过系统鉴定不同DUB底物蛋白来揭示DUB与底物蛋白的特异性关系 (另文发表)。
2.3 基于化学方法的DUB功能的蛋白质组学
2002年,Borodovsky等[62]结合蛋白质组学和化学技术发展了一种“基于化学的功能蛋白质组学”(Chemistry-based functional proteomics)的方法,来鉴定DUB及其相互作用蛋白。他们开发了一种称为HA-Ub-derived probe的功能探针。该探针是在N端含红细胞凝集素标签(Hemagglutinin-tag, HA-tag)的Ub,C末端再带上一个半胱氨酸反应基团。该反应基团能与DUB共价结合,并固定特异目的蛋白。最后经抗HA琼脂糖介质分离后利用质谱鉴定获取的蛋白质。该方法成功鉴定了24种DUB,还找到19种可能与DUB相互作用的蛋白。同样,Hemelaar等[63]利用该方法发现了包含OTU结构域的DUB,并用来评估DUB的活性。如果结合现在已有的高精度蛋白质组学平台,这种活性探针策略可在DUB发现及其相互作用蛋白研究方面发挥更大的作用。
2.4 去泛素化酶抑制剂
正如前文所述,开发关于DUB的抑制剂能够为我们更好地研究DUB在代谢通路中的功能提供利器。如何将抑制剂开发成药物也是科研工作者关注的焦点[18,64-68]。Altun等[67]基于活性-化学蛋白质组学 (Activity-based chemical proteomics)来鉴定和选择能够对特定DUB存在抑制活性的抑制剂。他们选取了小分子广谱DUB抑制剂PR-619为对照,利用无标定量 (Label-free quantification) 的方法来评估P22077对各种DUB的抑制情况,发现其对USP7存在特异性的抑制效应。由于USP7在癌症发生发展过程中的重要作用,因此该抑制剂具有进一步发展为癌症治疗药物的潜力。
2012年,Perry等[68]借助质谱确定了蛋白酶体相关去泛素化酶USP14为抑制剂WP1130的作用靶点,发现使用DUB抑制分子WP1130能显著抑制Norwalk病毒在鼠巨噬细胞内的复制。因此,我们发现通过使用特殊的DUB抑制剂可以特异地阻滞特定病毒感染,为抗病毒治疗提供新靶点。
3 前景与展望
综上所述,UPS内的DUB涉及到许多重要的生理过程和部分疾病与肿瘤的发生。因此研究针对DUB关键节点的调控成为抗癌治疗的重点。借鉴抗癌药物Velcade抑制蛋白酶体活性的思路,可为基于重要DUB作为靶点的抗癌药物开发提供思路。为实现这个目标,要对DUB的功能及其在生物体内复杂的动态过程的作用进行系统深入的研究。因此,将蛋白质组学技术应用到DUB的研究中显得特别重要。
随着蛋白质组学技术的快速发展,蛋白质组的深度测序和精确定量已经成为可能。因此,利用蛋白质组学技术可以对DUB及其相互作用网络进行深度覆盖和精确定量,系统阐释DUB的功能与疾病调控机制。利用功能化的蛋白质组学来揭示DUB底物特异性,更多地发现可能的DUB调控规律与特点,通过开发特异的DUB抑制剂还可为研究USP相关疾病的治疗提供新的技术手段。
REFERENCES
[1] Hershko A, Ciechanover A. The ubiquitin system. Annu Rev Biochem, 1998, 67: 425–479.
[2] Varshavsky A. The ubiquitin system. Trends Biochem, 1997, 22(10): 383–387.
[3] Hochstrasser M. Ubiquitin-dependent protein degradation. Annu Rev Genet, 1996, 30: 405–439.
[4] Reyes-Turcu FE, Ventii KH, Wilkinson KD. Regulation and cellular roles of ubiquitin-specific deubiquitinating enzymes. Annu Rev Biochem, 2009, 78: 363–397.
[5] Chen ZJ. Ubiquitin signalling in the NF-κB pathway. Nat Cell Biol, 2005, 7(8): 758–765.
[6] Pickart CM. Mechanisms underlying biquitination. Annu Rev Biochem, 2001, 70: 503–533.
[7] Haas AL, Wilkinson KD. DeTEKting ubiquitination of APC/C substrates. Cell, 2008, 133(4): 570–572.
[8] Amerik AY, Hochstrasser M. Mechanism and function of deubiquitinating enzymes. Biochim Biophys Acta, 2004, 1695(1/3): 189–207.
[9] Nijman SMB, Luna-Vargas MPA, Velds A, et al. A genomic and functional inventory of deubiquitinating enzymes. Cell, 2005, 123(5): 773–786.
[10] Komander D, Clague MJ, Urbé S. Breaking the chains: structure and function of the deubiquitinases. Nat Rev Mol Cell Biol, 2009, 10(8): 550–563.
[11] Reyes-Turcu FE, Wilkinson KD. Polyubiquitin binding and disassembly by deubiquitinating enzymes. Chem Rev, 2009, 109(4): 1495–1508.
[12] Xu P, Duong DM, Seyfried NT, et al. Quantitative proteomics reveals the function of unconventional ubiquitin chains in proteasomal degradation. Cell, 2009, 137(1): 133–145.
[13] Mann M, Kulak NA, Nagaraj N, et al. The coming age of complete, accurate, and ubiquitous proteomes. Mol Cell, 2013, 49(4): 583–590.
[14] Aebersold R, Mann M. Mass spectrometry-based proteomics. Nature, 2003, 422: 198–207.
[15] Gao X, Zhang XL, Zheng JJ, et al. Proteomics in China: ready for prime time. Sci China Life Sci, 2010, 53(1): 22–33.
[16] Kaiser P, Mayor T. Gold for ubiquitin in Vancouver: First Conference on Proteomics of Protein Degradation and Ubiquitin Pathways held June 6-8, 2010 in Vancouver, University of British Columbia, organized By Lan Huang, Thibault Mayor, and Peipei Ping. Mol Cell Proteomics, 2011, 10(5): R110 003863.
[17] Bennett EJ, Mayor T. Proteomics propels protein degradation studies in San Diego. Mol Cel Proteomics, 2012, 11(12): 1523–1528.
[18] Puente XS, López-Otín C. A genomic analysis of rat proteases and protease inhibitors. Genome Res, 2004, 14(4): 609–622.
[19] Hu M, Gu LC, Li MY, et al. Structural basis of competitive recognition of p53 and MDM2 byHAUSP/USP7: implications for the regulation of the p53-MDM2 pathway. PLoS Biol, 2006, 4(2): e27.
[20] Linghu B, Callis J, Goebl MG. Rub1p processing by Yuh1p is required for wild-type levels of Rub1p conjugation to Cdc53p. Eukaryot Cell, 2002, 1(3): 491–494.
[21] Liu Y, Fallon L, Lashuel HA, et al. The UCH-L1 gene encodes two opposing enzymatic activities that affect alpha-synuclein degradation and Parkinson's disease susceptibility. Cell, 2002, 111(2): 209–218.
[22] Leroy E, Boyer R, Auburger G, et al. The ubiquitin pathway in Parkinson's disease. Nature, 1998, 395(6701): 451–452.
[23] Semple CA, RIKEN GER Group, GSL Members. The comparative proteomics of ubiquitination in mouse. Genome Res, 2003, 13(6B): 1389–1394.
[24] Hu M, Li PW, Li MY, et al. Crystal structure of a UBP-family deubiquitinating enzyme in isolation and in complex with ubiquitin aldehyde. Cell, 2002, 111(7): 1041–1054.
[25] Sheng Y, Saridakis V, Sarkari F, et al. Molecular recognition of p53 and MDM2 by USP7/HAUSP. Nat Struct Mol Biol, 2006, 13(3): 285–291.
[26] Kruse JP, Gu W. Modes of p53 regulation. Cell, 2009, 137(4): 609–622.
[27] Everett RD, Meredith M, Orr A, et al. A novel ubiquitin-specific protease is dynamically associated with the PML nuclear domain and binds to a herpesvirus regulatory protein. EMBO J, 1997, 16(7): 566–577.
[28] Popov N, Wanzel M, Madiredjo M, et al. The ubiquitin-specific protease USP28 is required for MYC stability. Nat Cell Biol, 2007, 9(7): 765–774.
[29] Wertz IE, O'Rourke KM, Zhou HL, et al. De-ubiquitination and ubiquitin ligase domains of A20 downregulate NF-κB signalling. Nature, 2004, 430(7000): 694–699.
[30] Burnett B, Li FS, Pittman RN. The polyglutamine neurodegenerative protein ataxin-3 binds polyubiquitylated proteins and has ubiquitin protease activity. Hum Mol Genet, 2003, 12(23): 3195–3205.
[31] Scheel H, Tomiuk S, Hofmann K. Elucidation of ataxin-3 and ataxin-7 function by integrative bioinformatics. Hum Mol Genet, 2003, 12(21): 2845–2852.
[32] Verma R, Aravind L, Oania R, et al. Role of Rpn11 metalloprotease in deubiquitination and degradation by the 26S proteasome. Science, 2002, 298(5593): 611–615.
[33] Dong YS, Hakimi MA, Chen XW, et al. Regulation of BRCC, a holoenzyme complex containing BRCA1 and BRCA2, by a signalosome-like subunit and its role in DNA repair. Mol Cell, 2003, 12(5): 1087–1099.
[34] Kouranti I, McLean JR, Feoktistova A, et al. A global census of fission yeast deubiquitinating enzyme localization and interaction networks reveals distinct compartmentalization profiles and overlapping functions in endocytosis and polarity. PLoS Biol, 2010, 8(9): e1000471.
[35] Reyes-Turcu FE, Horton JR, Mullally JE, et al. The ubiquitin binding domain ZnF UBP recognizes the C-terminal diglycine motif of unanchored ubiquitin. Cell, 2006, 124(6): 1197–1208.
[36] Kouroukis CT, Baldassarre FG, Haynes AE, et al. Bortezomib in multiple myeloma: a Practice guideline. Clin Oncol (R Coll Radiol), 2014, 26(2): 110–119.
[37] Ventii KH, Devi NS, Friedrich KL, et al. BRCA1-associated protein-1 is a tumor suppressor that requires deubiquitinating activity and nuclear localization. Cancer Res, 2008, 68(17): 6953–6962. [38] Hoeller D, Dikic I. Targeting the ubiquitin system in cancer therapy. Nature, 2009, 458(7237): 438–444.
[39] Sun SC. CYLD: a tumor suppressor deubiquitinase regulating NF-kappaB activation and diverse biological processes. Cell Death Differ, 2010, 17(1): 25–34.
[40] Glittenberg M, Ligoxygakis P. CYLD: a multifunctional deubiquitinase. Fly (Austin), 2007, 1(6): 330–332.
[41] Graner E, Tang D, Rossi S, et al. The isopeptidase USP2a regulates the stability of fatty acid synthase in prostate cancer. Cancer cell, 2004, 5(3): 253–261.
[42] Cummins JM, Rago C, Kohli M, et al. Tumour suppression: disruption of HAUSP gene stabilizes p53. Nature, 2004, 428(6982): 1 p following 486.
[43] Tokarz S, Berset C, La Rue J, et al. The ISG15 Isopeptidase UBP43 is regulated by proteolysis viathe SCFSkp2 ubiquitin ligase. J Biol Chem, 2004, 279(45): 46424–46430.
[44] D’Arcy P, Linder S. Proteasome deubiquitinases as novel targets for cancer therapy. Int J Biochem Cell Biol, 2012, 44(11): 1729–1738.
[45] Lu AP, Waanders LF, Almeida R, et al. Nanoelectrospray peptide mapping revisited: composite survey spectra allow high dynamic range protein characterization without LCMS on an orbitrap mass spectrometer. Int J Mass Spectrometry, 2007, 268(2): 158–167.
[46] Bantscheff M, Schirle M, Sweetman G, et al. Quantitative mass spectrometry in proteomics: a critical review. Anal Bioanal Chem, 2007, 389(4): 1017–1031.
[47] Bantscheff M, Lemeer S, Savitski MM,et al. Quantitative mass spectrometry in proteomics: critical review update from 2007 to the present. Anal Bioanal Chem, 2012, 404(4): 939–965.
[48] Hebert AS, Richards AL, Bailey DJ, et al. The one hour yeast proteome. Mol Cell Proteomics, 2014, 13(1): 339–347.
[49] Peng JM, Schwartz D, Elias JE, et al. A proteomics approach to understanding protein ubiquitination. Nat Biotechnol, 2003, 21(8): 921–926.
[50] Hjerpe R, Aillet F, Lopitz-Otsoa F, et al. Efficient protection and isolation of ubiquitylated proteins using tandem ubiquitin-binding entities. EMBO Rep, 2009, 10(11): 1250–1258.
[51] Shi Y, Chan DW, Jung SY, et al. A data set of human endogenous protein ubiquitination sites. Mol Cell Proteomics, 2011, 10(5): M110 002089.
[52] Guerrero C, Tagwerker C, Kaiser P, et al. An integrated mass spectrometry-based proteomic approach: quantitative analysis of tandem affinity-purified in vivo cross-linked protein complexes (QTAX) to decipher the 26 S proteasome-interacting network. Mol Cell Proteomics, 2006, 5(2): 366–378.
[53] Kim W, Bennett EJ, Huttlin EL, et al. Systematic and quantitative assessment of the ubiquitinmodified proteome. Mol Cell, 2011, 44(2): 325–340.
[54] Xu GQ, Paige JS, Jaffrey SR, et al. Global analysis of lysine ubiquitination by ubiquitin remnant immunoaffinity profiling. Nat Biotechnol, 2010, 28(8): 868–873.
[55] Xu P, Cheng DM, Duong DM, et al. A proteomic strategy for quantifying polyubiquitin chain topologies. Isr J Chem, 2006, 46(2): 171–182.
[56] Sowa ME, Bennett EJ, Gygi SP, et al. Defining the human deubiquitinating enzyme interaction landscape. Cell, 2009, 138(2): 389–403.
[57] Xie Y, Avello M, Schirle M, et al. Deubiquitinase FAM/USP9X interacts with the E3 ubiquitin ligase SMURF1 protein and protects it from ligase activity-dependent self-degradation. J Biol Chem, 2013, 288(5): 2976–2985.
[58] Gomez-Ferreria MA, Bashkurov M, Mullin M, et al. CEP192 interacts physically and functionally with the K63-deubiquitinase CYLD to promote mitotic spindle assembly. Cell Cycle, 2012, 11(19): 3555–3558.
[59] Mevissen TE, Hospenthal MK, Geurink PP, et al. OTU deubiquitinases reveal mechanisms of linkage specificity and enable ubiquitin chain restriction analysis. Cell, 2013, 154(1): 169–184.
[60] Fiil BK, Damgaard RB, Wagner SA, et al. OTULIN restricts Met1-linked ubiquitination to control innate immune signaling. Mol Cell, 2013, 50(6): 818–830.
[61] Debelyy MO, Platta HW, Saffian D, et al. Ubp15p, a ubiquitin hydrolase associated with the peroxisomal export machinery. J Biol Chem, 2011, 286(32): 28223–28234.
[62] Borodovsky A, Ovaa H, Kolli N, et al. Chemistrybased functional proteomics reveals novel members of the deubiquitinating enzyme family. Chem Biol, 2002, 9(10): 1149–1159.
[63] Hemelaar J, Galardy PJ, Borodovsky A, et al. Chemistry-based functional proteomics mechanismbased activity-profiling tools for ubiquitin and ubiquitin-like specific proteases. J Proteome Res, 2004, 3(2): 268–276.
[64] Lee MJ, Lee BH, Hanna J, et al. Trimming of ubiquitin chains by proteasome-associated deubiquitinating enzymes. Mol Cell Proteomics, 2011, 10(5): R110 003871.
[65] Yang JM. Emerging roles of deubiquitinating enzymes in human cancer. Acta Pharmacol Sin, 2007, 28(9): 1325–1330.
[66] Clague MJ, Barsukov L, Coulson JM, et al. Deubiquitylases from genes to organism. Physiol Rev, 2013, 93(3): 1289–1315.
[67] Altun M, Kramer HB, Willems LI, et al. Activitybased chemical proteomics accelerates inhibitor development for deubiquitylating enzymes. Chem Biol, 2011, 18(11): 1401–1412.
[68] Perry JW, Ahmed M, Chang KO, et al. Antiviral activity of a small molecule deubiquitinase inhibitor occurs via induction of the unfolded protein response. PLoS Pathog, 2012, 8(7): e1002783.
(本文责编 郝丽芳)
Application of proteomics in deubiquitinases research
Yanchang Li1, Yuan Gao1, Zhongwei Xu1, Qiuyan Lan1,2, and Ping Xu1,2
1 Beijing Institute of Radiation Medicine, State Key Laboratory of Proteomics, National Center for Protein Sciences Beijing, Beijing Proteome Research Center, National Engineering Research Center for Protein Drugs, Beijing 102206, China
2 Key Laboratory of Combinatorial Biosynthesis and Drug Discovery (Wuhan University), Ministry of Education, and Wuhan University School of Pharmaceutical Sciences, Wuhan 430072, Hubei, China
As the major pathway mediating specific protein degradation in eukaryotes, ubiquitin-proteasome system (UPS) is involved in various physiological and pathological processes such as cell cycle regulation, immune response, signal transduction and DNA-repair. Deubiquitinases (DUB) maintain the balance of UPS and related physiological processes via reversibly removing ubiquitin from the covalently modified protein substrates, which have been implicated in various disease processes in case of their imbalance expression. Because DUB plays critical regulating roles in the UPS pathway, they may be also the ideal drug targets for severe and intractable human diseases, such as cancer and neurodegenerative disease. With the rapid development of proteomic technology, systematical investigation of specific substrates and interacting proteins of varied DUB via mass spectrometry approach may shed light on these DUB’s biological function and regulating roles in the physiological and pathogenic states. In this review, we briefly introduce the characteristics of DUB and summarize the recent application and progresses of proteomics in DUB research.
ubiquitin-proteasome system, deubiquitinase, proteomics, interactome , mass spectrometry
December 23, 2013; Accepted: February 10, 2014
Ping Xu. Tel: +86-10-83147777-1314; Fax: +86-10-80705155; E-mail: xupingghy@gmail.com
Supported by: National Basic Research Program of China (973 Program) (Nos. 2011CB910600, 2013CB911200), National High Technology Research and Development Program of China (863 Program) (Nos. SS2012AA020502, 2011AA02A114), National Natural Science Foundation of China (Nos. 31070673, 31170780), Beijing Natural Science Foundation (No. 5112012), Foundation of State Key Lab of Proteomics (No. SKLP-Y201102).
国家重点基础发展研究计划 (973计划) (Nos. 2011CB910600,2013CB911200),国家高技术发展计划 (863计划) (Nos. SS2012AA020502, 2011AA02A114),国家自然科学基金 (Nos. 31070673, 31170780),北京市自然科学基金 (No. 5112012), 蛋白质组学国家重点实验室基金 (No. SKLP-Y201102) 资助。
Received: December 23, 2013; Accepted: February 10, 2014
Supported by: National Basic Research Program of China (973 Program) (Nos. 2011CB910600, 2013CB911200), National High Technology Research and Development Program of China (863 Program) (Nos. SS2012AA020502, 2011AA02A114), National Natural Science Foundation of China (Nos. 31070673, 31170780), Beijing Natural Science Foundation (No. 5112012), Foundation of State Key Lab of Proteomics (No. SKLP-Y201102).
Corresponding author: Ping Xu. Tel: +86-10-83147777-1314; Fax: +86-10-80705155; E-mail: xupingghy@gmail.com
国家重点基础发展研究计划 (973计划) (Nos. 2011CB910600,2013CB911200),国家高技术发展计划 (863计划) (Nos. SS2012AA020502, 2011AA02A114),国家自然科学基金 (Nos. 31070673, 31170780),北京市自然科学基金 (No. 5112012), 蛋白质组学国家重点实验室基金 (No. SKLP-Y201102) 资助。

