双峰驼凝乳酶原基因的生物信息学分析
杨 艺,普 燕,张富春
(新疆大学生命科学与技术学院,新疆生物资源与基因工程重点实验室,新疆,乌鲁木齐,830046)
随着乳品行业的高速发展,人们对发酵乳制品的需求也日益增加。近年来国内外干酪市场发展迅猛,不仅对干酪的研究日益深入,而且制作干酪所用的凝乳酶需求量大增。干酪加工中添加凝乳酶的主要目的是促使牛乳凝结,为排除乳清提供条件[1]。凝乳酶是一种从未断奶的小牛皱胃中发现的天门冬氨酸蛋白酶,可以专一地切割乳中κ-酪蛋白的Phe105-Met106之间的肽键,破坏乳中的酪蛋白胶束引起蛋白质凝聚,因此在乳制品尤其是在干酪加工制作中应用广泛[2],理想的凝乳酶应能迅速、专一地打开κ-酪蛋白Phe105-Met106键,但水解蛋白其他肽键的能力较低,即两者的活力比值要高[3]。凝乳酶凝乳后制成的干酪相当于将原料奶中的蛋白质和脂肪浓缩了十倍左右,另外干酪中还含有糖类、有机酸、常量矿物质元素等多种成分,具有很高的营养价值。凝乳酶主要为动物来源的凝乳酶,约占市场的70%,同时还有植物来源的凝乳酶、微生物来源的凝乳酶和利用基因工程技术生产的重组凝乳酶。牛凝乳酶有着强大的凝乳活性和较低的蛋白水解活性,是凝乳制作干酪的优质酶,目前对牛凝乳酶的研究已经较为成熟,市场上销售的凝乳酶主要是牛凝乳酶,而相应骆驼凝乳酶研究较少,因此骆驼凝乳酶开发利用前景广阔。Kappeler等[4]研究发现单峰驼凝乳酶与牛凝乳酶有不同的特性,单峰驼凝乳酶表现出比牛凝乳酶高70%的凝乳活性,却只有牛凝乳酶的20%非特异性蛋白酶活性。动力学分析表明,达到半饱和时,其所需的底物仅是牛凝乳酶的50%还少,并且牛凝乳酶不能凝结骆驼乳,说明骆驼凝乳酶有较高的凝乳活性。Jensen等[5]研究表明单峰驼凝乳酶的稳定性高于牛凝乳酶。
本研究主要运用生物信息学的方法对双峰驼凝乳酶基因及相应的氨基酸序列的同源性、理化性质、保守结构域、亚细胞定位、信号肽、跨膜结构域、亲水性/疏水性、二级结构以及活性位点进行预测分析。旨在为研究双峰驼凝乳酶的凝乳特性及其条件提供理论参考。
1 材料和方法
1.1 双峰驼凝乳酶原基因序列的获得
首先从Broe[6]等人在2011申请的专利中获得双峰驼凝乳酶原基因序列,由上海生工生物工程有限公司合成双峰驼凝乳酶原基因开放阅读框序列,在大肠杆菌进行原核表达,发现双峰驼凝乳酶原融合蛋白以包涵体形式表达,分子大小与理论值相符。将双峰驼凝乳酶原融合蛋白包涵体溶于尿素,但透析复性的双峰驼凝乳酶原融合蛋白经过酸化/中和处理并没有得到有活性的凝乳酶,说明该基因序列所产生的双峰驼凝乳酶原有误。随后通过检索GeneBank数据库,发现Kappeler等人正式注册的双峰驼凝乳酶原基因序列,与从专利获得的序列比对后,发现两者双峰驼凝乳酶原有六个氨基酸的差异,推测可能是这六个氨基酸的差异导致合成的凝乳酶无活性。本研究采用从GeneBank数据库中获得的双峰驼凝乳酶原基因序列进行分析。
1.2 生物信息学分析
利用 NCBI及 ProtParam、ProtScale、SignalP 4.0 Server、TMHMM2.0、等在线工具进行核酸及氨基酸序列的组成、理化性质、信号肽和亲水性/疏水性分析;利用Blast和DNAMAN在线工具以及MEGA4.0软件,进行氨基酸序列的序列比对和同源性分析。所用软件或在线工具有关信息见表1。

表1 预测基因结构和功能应用的网站及软件Table 1 The websites and softwares for predicting the gene structure and function
2 结果与分析
2.1 双峰驼凝乳酶原基因序列
双峰驼凝乳酶原基因序列是一个完整的开放阅 读框,为1 146 bp,编码381个氨基酸(见图1)。

图1 双峰驼凝乳酶原序列及其氨基酸序列Fig.1 Nucleotide acid sequence and amino acid sequence of prochymosin in camelus bactrianus
2.2 双峰驼凝乳酶原的同源性分析
通过GeneBank数据库中的Blastp程序,对双峰驼凝乳酶的氨基酸序列进行同源性分析,数据显示其与单峰驼,牦牛、牛、山羊、印度水牛、绵羊凝乳酶氨基酸序列相似性分别为99%、84%、84%、84%、83%和84%。利用DNAMAN软件对双峰驼凝乳酶原的氨基酸序列和其它物种凝乳酶原的氨基酸序列进行多重比对(见图2)。运用MEGA4.0软件,根据上述动物的凝乳酶原的氨基酸序列构建的系统进化树(见图3),进一步的验证了其同源性。

图2 双峰驼凝乳酶原以及其它动物种凝乳酶原的基氨基酸序列比对Fig.2 Homology analysis of camelus bactrianus prochymosin amino acid sequences and those from other animal species

图3 不同物种基于凝乳酶原氨基酸序列的系统进化树Fig.3 Phylogenetic tree of different species basedon amino acid of prochymosin in camelus bactrianus
2.3 双峰驼凝乳酶原氨基酸一级结构及理化性质预测分析
通过ExPASyProtParam预测双峰驼凝乳酶原基因编码的氨基酸序列的组成和理化性质,并与单峰驼、牛等动物凝乳酶原基因的一级结构进行比较,结果见表2。结果显示,凝乳酶原基因均编码381个氨基酸,并且其带正、负电荷氨基酸数目相差甚微,分子量差异很小,等电点在4.8到5.9之间,其半衰期均大于10小时,表明其有较高稳定性,根据定义当不稳定系数分值小于40时,预测蛋白质比较稳定;当不稳定系数分值大于40时,则该蛋白质不稳 定[7],因此七种凝乳酶原均为较稳定的蛋白。

表2 不同物种凝乳酶原的氨基酸理化性质的比较Table 2 The physical and chemical characteristics of amino acid sequences of different prochymosin
2.3 双峰驼凝乳酶原的保守结构域分析
通过NCBI保守结构域数据库(Conserved Domain Database,CDD),分析双峰驼凝乳酶原中的保守结构域(见图4),结果显示双峰驼凝乳酶原属于胃蛋白酶A超家族,含有多个胃蛋白酶保守结构域。

图4 预测的双峰驼凝乳酶原保守结构域Fig.4 Conserved domain prediction of prochymosin in camelus bactrianus
2.4 双峰驼凝乳酶的亚细胞定位
应用Psort工具预测双峰驼凝乳酶的亚细胞定位,结果见表2,其可能定位于内质网(膜)、过氧化物酶体、细胞质膜和内质网(腔)。表3显示双峰驼凝乳酶定位于内质网(膜)的概率较高,为0.820,定位于过氧化物酶体、细胞质膜和内质网(腔)的概率甚微。

表3 双峰驼凝乳酶原的亚细胞定位Table 3 Subcellular location of prochymosin of camelus bactrianus
2.5 双峰驼凝乳酶原信号肽预测和分析

图5 双峰驼凝乳酶信号肽预测Fig.5 Signal P prediction of prochymosin in camelus bactrianus
通过Signal P4.0Server软件预测双峰驼凝乳酶信号肽(见图5),结果显示:第17位丝氨酸残基具有最高的原始剪切位点分值0.661,第8位亮氨酸残基具有最高的信号肽分值0.946,第17位丝氨酸残基具有最高的综合剪切位点分值0.778。由于氨基酸残基的原始剪切位点和信号肽的分值均,可推测双峰驼凝乳酶基因所编码的蛋白存在信号肽,属于分泌蛋白,与Foltman关于牛凝乳酶的研究一致,该研究表明牛凝乳酶存在一个16个氨基酸组成的前导信号序列,在通过细胞膜分泌中很重要[15]。
2.6 双峰驼凝乳酶原跨膜结构域预测和分析
跨膜结构域是膜内在蛋白与膜脂相结合的主要部位,它固着于细胞膜上起锚定作用,一般由20个左右的疏水氨基酸组成。通过Expasy软件中的TMHMM Server v.2.0工具预测双峰驼凝乳酶原基因的跨膜螺旋区(见图6)。预测结果显示,双峰驼凝乳酶原基因氨基酸序列中不存在跨膜螺旋区,蛋白质整体381个氨基酸残基全部在膜外,为非跨膜蛋白。

图6 双峰驼凝乳酶原跨膜结构域预测Fig.6 Predicted transmembrane domain of prochymosin in camelus bactrianus
2.7 双峰驼凝乳酶原氨基酸序列的疏水性亲水性的预测和分析
通过ExPASyProtScale在线软件,对双峰驼凝乳酶原基因所编码的氨基酸序列的疏水性亲水性进行预测,其结果见图7,分析双峰驼凝乳酶的结果显示,双峰驼凝乳酶,最高的分值3.300,为第7位的亮氨酸,疏水性最强;最低的分值-2.789,为第115和116位的精氨酸和苯丙氨酸,亲水性最强。GRAVY值是测蛋质的疏水性标准,通常GRAVY值分布在2至-2之间,如为正值,则该蛋白为疏水蛋白;如为负值,则为亲水蛋白。其GRAVY值为 -0.096,从整体来看,亲水性氨基酸残基的分布在整条肽链上,且在整体上多于疏水性氨基酸残基,推测双峰驼凝乳酶是一种可溶性蛋白。
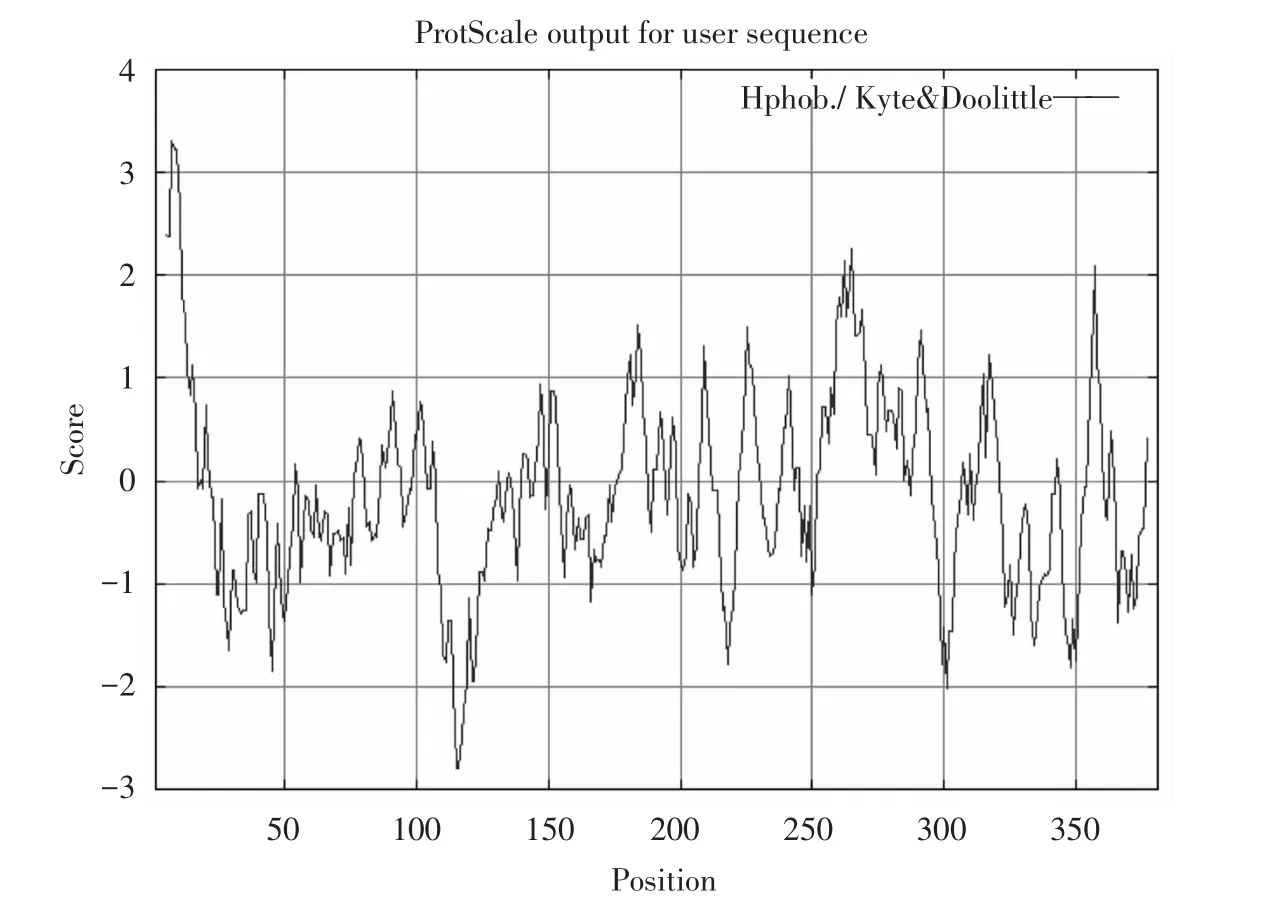
图7 双峰驼凝乳酶原氨基酸序列的疏水性亲水性预测Fig.7 Predicted hydrophobicity/hydrophilicity of the amino acidsequence of camelus bactrianus prochymosin
2.8 双峰驼凝乳酶原二级结构预测和分析
蛋白质的二级结构主要包括α螺旋,延伸链,β转角,无规则卷曲以及模序等蛋白质局部结构组件。通过GOR4方法对双峰驼凝乳酶原二级结构进行在线分析(见表4,图8),在双峰驼凝乳酶原二级结构中,无规则卷曲占的比例最高为53.02%,α螺旋占17.32%,延伸链占29.66%。由此可推测,无规卷曲是双峰驼凝乳酶原二级结构中最大量的结构元件,α螺旋和延抻链分散于整个蛋白质中。

表4 双峰驼凝乳酶二级结构典型构象统计结果Table 4 Secondary structure prediction of camelus bactrianus prochymosin
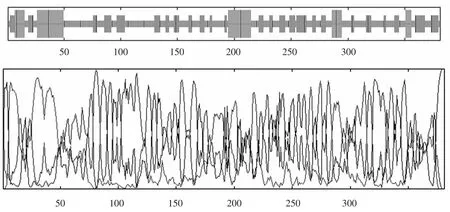
图8 双峰驼凝乳酶原二级结构预测和分析Fig.8 Predicted secondary structure of camelus bactrianus prochymosin
2.9 双峰驼凝乳酶原的活性位点的分析
通过NPS的PROSCAN对双峰驼凝乳酶原进行活性位点的分析(见表5),结果显示,双峰驼凝乳酶原有6类活性位点,分别是N-糖基化位点、cAMP和cGMP依赖性蛋白激酶磷酸化位点、蛋白酶C磷酸化位点、酪蛋白激酶II磷酸化、N–十四酰化位点、真核生物和病毒天门冬氨酰蛋白酶活性位点。这可能与凝乳酶的分泌以及自剪切成为有活性的酶等生物学功能相关。
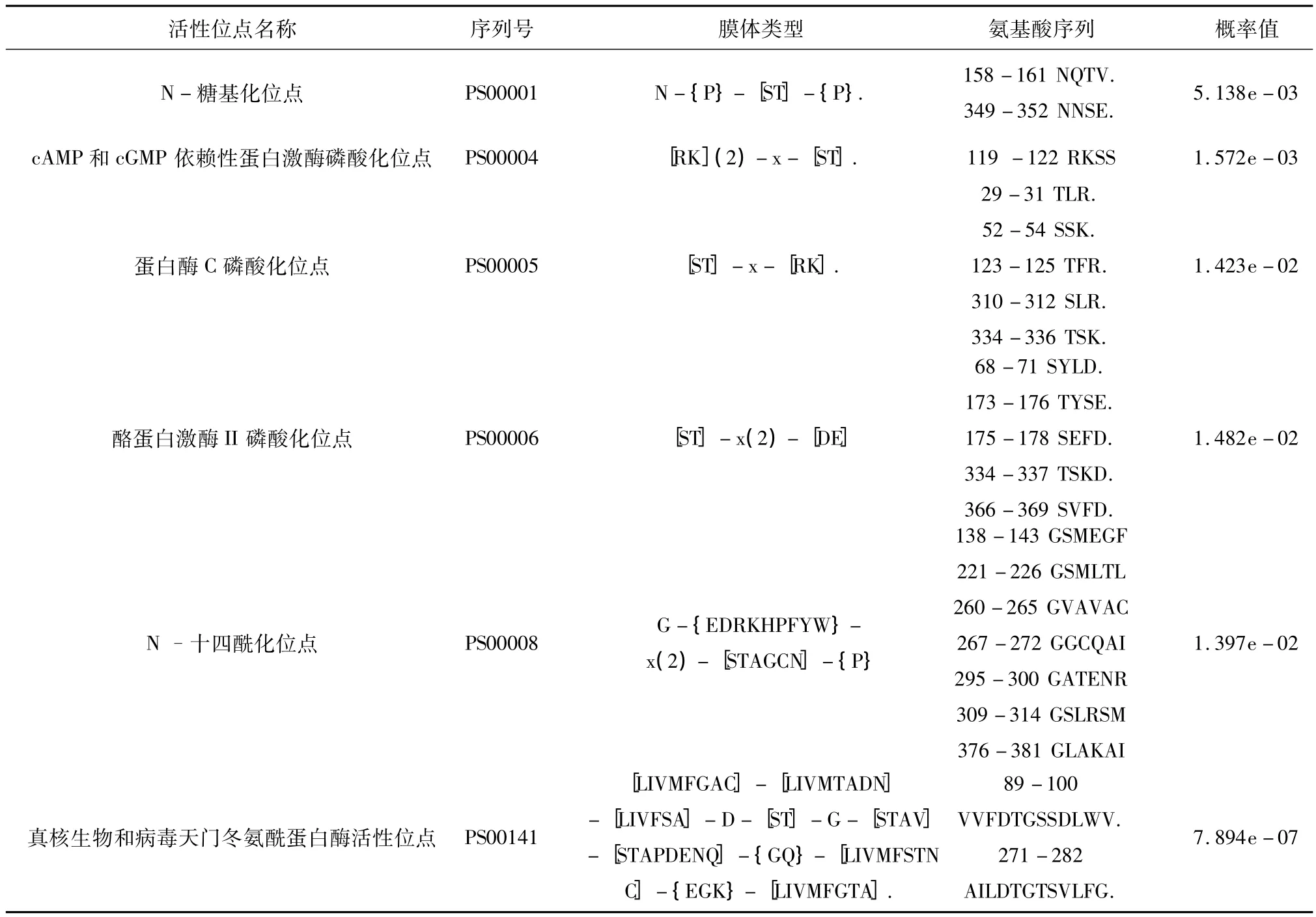
表5 双峰驼凝乳酶原在NPS中的活性位点预测分析Table 5 Scanning of camelus bactrianus prochymosin for site/signatures against proscan database in NPS
3 结论
骆驼乳具有多种生理活性物质,营养价值远远高于一般的动物乳制品。这些独特成分使其在防病抗病方面起着积极的作用[16]。Jesper等研究的结果表明,单峰驼凝乳酶作为一种替代品已经被成功地销售,因为其能提供较高的干酪产量和制备的干酪苦味少,风味很好,并且蛋白水解的活性较低[17]。Bansal N.等研究的结果表明,单峰驼凝乳酶适合用于契达奶酪的制作,不仅蛋白水解水平较低,并且具有有良好的风味[18]。目前骆驼凝乳酶研究较少,对于双峰驼凝乳酶原基因的生物信息分析尚未见报道,因此骆驼凝乳酶开发利用前景广阔。
本研究运用生物信息学分析软件,系统地研究了双峰驼凝乳酶原的基因序列和氨基酸序列。结果显示,双峰驼凝乳酶原基因全长1 146 bp,编码381个氨基酸,属于胃蛋白酶A超家族,可能定位于内质网(膜)的稳定亲水性蛋白,不含跨膜结构域。无规卷曲是其二级结构中最大量的结构元件,α螺旋和延抻链分散于整个蛋白质中,编码蛋白有6类活性位点,主要分为N-糖基化位点,蛋白激酶磷酸化位点,蛋白酶磷酸化位点,N-十四酰化位点和蛋白酶活性位点。通过分析双峰驼凝乳酶原基因及其编码蛋白质的特征为研究其凝乳特性及其条件提供理论参考,对凝乳酶凝乳功能的研究具有重要的意义。
References)
[1] 钟继才.凝乳酶在干酪生产中的应用[J].中国乳品工业,2006,6(1):54-56.ZHONG Jicai.Application of rennin in cheese making[J].China Dairy Industry,2006,6(1):54-56.
[2] WILLIAMS M G,WILSHER J,NUGENT P,et al.Mutagenesis,biochemical characterization and X-ray structural analysis of point mutants of bovine chymosin[J].Protein Eng,1997,10(9):991-997.
[3] 周俊清.产高效凝乳酶菌株获得方法的探讨[J].食品科学,2005,26(3):253-256.ZHOU Junqing.Discussion on method of yielding high effect chymosin-producing strain [J]. Food Science,2005,26(3):253-256.
[4] KAPPELER S R,BRINK H M V D,RAHBEK-NIELSEN H,et al.Characterization of recombinant camel chymosin reveals superior properties for the coagulation of bovine and camel milk [J].Biochemical and Biophysical Research Communications,2006,342(2):647-654.
[5] JANGHOLM JENSEN J, MØLGAARD A, NAVARRO POULSEN J C,et al.Camel and bovine chymosin:the relationship between their structures and cheese-making properties[J].Biological Crystallography,2013,D69,901-913.
[6] BROE M L,BRINK J V D,HARBOE M R.Marianne Kirsten Harboe.Coagulation of Milk [P].United States,US2011/0008492A1,2011-1-13.
[7] 陈珊珊,郭晋隆,李国印,等.甘蔗过氧化氢酶基因的电子克隆及生物信息学分析[J].生物信息学,2012,10(1):65-70.CHEN Shanshan,GUO Jinlong, LIGuoyin,etal.Electronic cloning and characterization of CAT gene from Saccharum officinarum using bioinformatics tools [J].Chinese Journal of Bioinformaties,2012,10(1):65-70.
[8] GASTEIGER E,HOOGLAND C,GATTIKER A,et al.Protein identification and analysis tools on the ExPASy server[J].The Proteomics Protocols Handbook,2005,571-607.
[9] PETERSEN T N,BRUNAK S,HEIJINE G V,et al.Signal P 4.0: discriminating signalpeptidesfrom transmembrane regions [J].Nature Methods,2011,8(10):785-786.
[10] NAIR R,ROST B.Better prediction of sub-cellular localization by combing evolutionary and structural information[J].PROTEINS:Structure,Function,and Genetics,2003,53:917-930 .
[11]郝爱平,熊超超.小鼠Pitx2基因的生物信息学分析[J].生物技术,2012,22(6):13-16.HAO Aiping,XIONG Chaochao.Bioinformatics analysis of pitx2 gene in mouse[J].Biotechnology,2012,22(6):13-16.
[12] MA Tingting,ZHOU Yijun,GAO Fei,et al.Bioinformatical analysis on sequence and functions ofperoxidase in Arabidopsis [J]. Agricultural Biotechnology,2012,13(8):1645-1650.
[13] TAMNRA R,DUDLEY J,NEI M,et al.MEGA4:Molecular Evolutionary Genetics Analysis(MEGA)software version 4.0[J].Molecular Biology Evolution,2007,24(8):1596-1599.
[14]蔡明,高贝,张道远.齿肋赤藓热激蛋白60基因的电子克隆和生物信息学分析[J].生物信息学,2013,11(3):216-223.CAI Ming, GAO Bei, ZHANG Daoyuan. Electronic cloning and characterization of HSP60 gene from Syntrichia caninervis using bioinformatics tool[J].Chinese Journal of Bioinformaties,2013,11(3):216-223.
[15] FOLTMANN B,PEDERSEN V B,JACOBSEN H,et al.The complete amino acid sequence ofprochymosin [J].Proc Natl Acad Sci USA,1977,74:2321-2324.
[16]何俊霞,哈斯苏荣,那仁巴图,等.驼乳医疗保健作用的研究进展Ⅱ[J].食品科学,2009,30(23):504-507.HE Junxia,HASISURONG,NARENBATU,et al.Research progress in health care function of camel milk[J].Food Science,2009,30(23):504-507.
[17] SØRENSEN J,PALMER D S,QVIST K B,et al.Initial Stage of Cheese Production:A Molecular Modeling Study of Bovine and Camel Chymosin Complexed with Peptides from the Chymosin-Sensitive Region of K-Casein [J].Journal of Agricultural and Food Chemistry,2011,59:5636-5647.
[18] BANSAL N,DRAKE M A D A,PIRAINO P,et al.Suitability of Recombinant Camel(Camelus Dromedarius)Chymosin as a Coagulant for Cheddar Cheese [J].International Dairy Journal,2009,19:510-517.

