酵母基因编辑技术:从单基因操作到基因组重建
李晨,楼慧强中国农业大学生物学院,农业生物技术国家重点实验室,北京 100193
酵母基因编辑技术:从单基因操作到基因组重建
李晨,楼慧强
中国农业大学生物学院,农业生物技术国家重点实验室,北京 100193
DNA双链发生断裂(Double strand break, DSB)时,细胞通常会采用同源重组(Homologous recombination, HR)这一主要修复方式。基于这一原理,人们开发出多种技术引入双链断裂从而实现对基因组 DNA序列的靶向编辑。真核生物基因敲除或编辑最早通过在外源片段中引入双链断裂和同源臂在酵母中获得成功。30多年来,酵母遗传操作走过了一条从单基因单位点到多基因多位点,乃至最新的基因组合成与重建之路。本文沿着引入DNA双链断裂技术的发展与演变这一主线,对一系列基因编辑及基因组编辑技术的发展进行了简要回顾和综述,旨在理清基因编辑技术的发展脉络,也为高等哺乳动物的基因编辑技术体系的建立和完善提供参考。
DNA双链断裂;基因编辑;基因组编辑;基因组合成与重建;酵母
同源重组(Homologous recombination)是指发生在具有相同或相近序列的DNA片段之间的交换,是广泛存在于各种生命形式中的遗传重组方式之一。除了减数分裂等过程中程序性发生的同源重组,这一机制通常被细胞用于修复 DNA双链断裂(Double strand break,DSB)等极为严重的DNA损伤[1]。因此,通过在细胞中人为导入DSB即可激发该位点附近包含相同或相近DNA序列区段的同源重组。利用同源重组的序列特异性和序列交换的特性,科学家很早就建立了细胞内基因靶向敲除、置换等各种遗传操作技术[2]。
在所有真核生物中,最早实现基因敲除的是单细胞模式生物——酿酒酵母(Saccharomyces cerevisiae)[3]。酿酒酵母具有极为高效的同源重组的特点[4],表现为极高的同源重组频率和较短的同源片段长度需求(最少只需20~40 bp)。正是这些先天优势促使酿酒酵母成为遗传操作最为便利的模式系统。基因靶向敲除技术开创了反向遗传学(Reverse genetics)。随着越来越多物种全基因组序列的测定,反向遗传学逐渐替代经典的正向遗传学(Forward genetics)而成为现代遗传学的主要研究手段。这一技术很快就被拓展应用于基因组上任意位点的各种变化,包括替换、插入、添加标签等。利用这些技术,在酿酒酵母中首次构建完成了全基因组范围内非必需基因的单敲除文库(单倍体细胞)、覆盖全部基因的单敲除文库(双倍体细胞)以及每个开放阅读框(Open reading frame, ORF)3′端添加GFP标签的菌株文库[5, 6]。这些宝贵的资源给全世界酵母遗传学研究者带来了极大的便利,推动了功能基因组学、化学基因组学、相互作用组学、系统生物学等多门新学科的诞生和迅猛发展[6]。
正是源于功能基因组学时代对多基因功能研究和多基因操作的需求,基因编辑技术朝着以下趋势快速发展改进:从单基因向多基因;从纯手工向机器自动化;从低通量向高通量;从小规模向大规模等。本文以长期在遗传操作技术上领衔的模式系统酵母为例对基因编辑技术的发展历程和趋势进行了简要综述。
1 外源片段中引入DSB
1.1 单一位点编辑
第一代基因编辑技术的原理如图 1所示。首先选择拟敲除基因的上下游约 500~1000 bp片段为同源臂,将同源臂克隆到携带筛选标记基因的质粒载体中。敲除载体构建完成后,选择合适的限制性内切酶切割引入双链断裂。将线性化的DNA片段通过转化导入目标细胞。转入细胞的外源DNA片段被细胞内DSB识别因子识别,启动修复机制,将携带有筛选标记的外源片段通过同源序列之间的交换整合到酵母基因组DNA。阳性转化子可以通过筛选标记(如营养缺陷和抗性等)获得并进一步通过菌落 PCR等技术验证基因编辑的准确性。当受体细胞(如酿酒酵母)同源重组频率较高,对同源臂长度要求较低,如40 bp左右的同源臂,则不必进行载体克隆,利用一步PCR产物转化即可实现基因敲除。
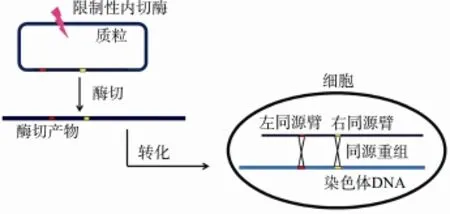
图1 单一位点编辑
因此根据某单一靶标位点 DNA序列来设计外源片段两端同源臂,并通过向外源片段中引入DSB,即实现单一位点的重组交换。
1.2 多位点编辑
遗传学家们很早就认识到细胞内基因并不是单独起作用,而是存在着复杂的相互作用,并且基因与基因间的相互作用在细胞的各项生物学过程中都起着极为关键的作用。因此,人们已经越来越不满足于对单个基因的遗传分析。
在对单一位点编辑的基础上,人们也开始将同源重组技术应用于基因组上多位点的编辑。DNA测序技术的快速发展一方面为人们破译了越来越多物种的基因组全序列,同时也推动了基因组修饰技术的发展。根据基因组上的多个不同位点,设计多对同源臂,并将含有 DSB的多个外源片段导入细胞,促进多个外源片段与基因组之间发生多位点同源重组。下面以多重自动化基因组工程(Multiplex automated genome engineering,MAGE)为例介绍一下自动化大规模遗传操作技术的发展。在多重自动化基因工程技术中,多个外源片段在λ同源重组系统Beta蛋白介导下,随DNA复制整合到细菌或酵母基因组上。2009年,哈佛医学院著名遗传学家George Church领导的研究团队在Nature杂志上发表了这个在基因组编辑上取得重大突破的方法[7]。这种寡核苷酸片段介导的等位基因替换(Oligo-mediated allelic replacement)能够以很高的效率在基因组上引入一系列遗传修饰。在众多含有变异序列的寡核苷酸片段的混合物中,与靶序列具有较高同源性的寡核苷酸片段能够以更高的效率与染色体 DNA发生同源重组而整合进入染色体DNA。这一过程可以伴随新一轮的细胞分裂循环进行,即只要存在含有变异的外源片段,置换重组就可以持续进行(图2)。MAGE方法的这一特点使得人们能够快速实现基因组的多样化改造。总体看来,MAGE方法具有高效率、低成本的优势,而且还能够同时对基因组上多个位点进行自动的多种不同的修饰,实现全基因组水平的序列多样性[7]。对于寡核苷酸片段,可以通过对序列原本存在的DNA片段进行酶切或PCR操作来获得,也可以引入人为设计合成的DNA片段,进一步加大遗传多样性。
MAGE方法除了能够对基因组进行编辑以获得更多的新基因型外,也能够进行基因组的从头合成(de novo genome synthesis)。片段两两之间存在同源臂,可通过同源重组的方式完成超大片段甚至完整染色体的组装(图3)。因为序列变异可伴随着每一次细胞分裂被循环引入和积累,MAGE方法也被看做一个加速进化的平台。在MAGE方法的基础上,研究人员利用细菌自身带有的接合能力促进细胞更高效地接受外源基因。这种改进后的方法称为接合组装基因组工程(Conjugative assembly genome engineering, CAGE)[8]。CAGE通常被应用于工程菌株的遗传育种和优化。譬如将含有不同变异的菌株混合,利用细菌天然的结合生殖能力,将这些目标变异整合到同一基因组中。该方法的优势是无需人工体外作用,并且可实现菌株之间大规模的基因组区域的转移重组[9]。与MAGE方法联合使用,可以显著缩短全基因组改造所需时间。

图2 多位点基因组编辑

图3 多位点基因组编辑技术用于从头合成基因组
2 在染色体DNA中引入DSB
在基因组DNA较小、同源重组效率相对较高的低等真核生物中,上文所述通过外源线性片段的形式引入DSB启动同源重组的基因敲除技术相对容易获得。但在多细胞生物中,随着基因组的逐渐增大和同源重组频率下降,获得阳性转化子的效率显著降低。例如,在小鼠胚胎干细胞中的基因敲除效率大约为百万分之一。此时,通常可以采用阻断其他DNA修复途径如非同源末端连接(Non-homologous end joining, NHEJ)等方法人为提高同源重组效率。敲除Ku70/80等NHEJ修复方式中关键基因可极大提高同源重组频率[10],从而被广泛用于细菌、丝状真菌在内的多种模式生物中。这一方法由于人为引入Ku70/80敲除背景,增加了对目标基因遗传分析的干扰;此外该方法也不适用于更为复杂的哺乳动物。因此,在基因组靶标位点特异地引入DSB,大幅提高该区段的同源重组频率成为新一代基因编辑技术的最佳策略。这一技术对于基因组极为庞大的高等哺乳动物细胞的研究尤为重要。
2.1 PCR片段介导的染色体断裂(PCR-mediated chromosome splitting, PCS)
Baudin等[11]于 1993年提出了利用同源区域来实现PCR介导的基因断裂方法。与传统的染色体断裂方法相比,PCR介导的断裂方法的最大特点是无需克隆目的基因,只要具有目的基因的序列信息,即可以实现目标位置的精确断裂。PCS方法切割染色体包含了两步PCR和一步转化过程(图4)。首先根据靶位点设计引物进行第一步PCR扩增得到左右同源臂;根据第一步PCR产物的部分序列设计第二步PCR的引物,通过融合PCR[12],将待引入染色体中的中心粒序列、端粒序列(约800 bp,含有5′-(CCCCAA)6-3′)及筛选标记序列(约400 bp)分别引入。得到含有筛选标记融合后的两个重组片段,分别带有与染色体DNA靶位点部分序列匹配的同源臂;将两个片段转化细胞,即可利用同源重组完成对染色体DNA的剪切过程,得到具有端粒及中心粒的新染色体[13]。
在PCS方法中,维持断裂后产生的新染色体的稳定性极为重要。中心粒、端粒及自主复制起始序列(Autonomously replicating sequence, ARS)是染色体的 3个必要元件,对细胞中染色体的稳定维持具有重要的作用。每条染色体正常只含有一个中心粒序列和两个端粒序列。经染色体切割产生的新染色体,必然会缺少中心粒和端粒序列。最初的PCS方法的目的即是:为剪切后获得的染色体添加中心粒序列和端粒序列,保证断裂染色体的基本功能的完整性和稳定性。

图4 PCS方法实现染色体DNA的定点断裂
2.1.1 ARS-PCS方法
最初的PCS方法主要是保证了被剪切后所得的染色体具有中心粒和端粒。尽管每条染色体通常具有多个 ARS序列,仍有可能切割得到不含有 ARS的新染色体。在没有ARS的情况下,染色体不能正常地进行自主复制。如将动物或植物来源的染色体片段克隆到酵母人工染色体(Yeast artificial chromosome,YAC)上时,这种情况经常会发生。为解决这个问题,人们在 PCS方法的基础上改进出ARS-PCS方法,即在上述的PCS方法中,将H4 ARS的序列设计入第二步PCR的引物中[14]。这样,在随后的片段与染色体DNA发生同源重组后,可以使得原先不具有ARS的那部分染色体被加上外源的ARS元件,保证了染色体的正常复制和传递。
2.1.2 Cre/loxP-PCS方法
筛选标记对于所有基因组编辑技术都是必需的为了实现筛选标记的重复利用,人们将Cre/loxP位点特异性重组系统引入到PCS方法中。Cre/loxP位点特异性重组系统是条件性及时空特异性基因打靶的核心技术,通过loxP序列的定位及Cre酶的重组作用,实现对目标靶位的切割。在应用PCS方法或ARS-PCS方法时,在筛选标记的上游和下游分别添加一个loxP序列。同时在细胞中转入可诱导的GAL1启动子控制的 Cre重组酶表达质粒。这样在完成阳性克隆的筛选后,启动 Cre酶表达,即可去除该筛选标记,进而实现筛选标记的重复利用[14]。基于同样的原理,Cre/loxP也可实现基因组上任何片段的删除,可以通过融合 PCR在欲敲除片段两端加入loxP序列,在Cre酶的作用下完成靶序列的删除。
2.2 PI-Sce I方法
归巢核酸内切酶系统也可以帮助人们实现条件性的染色体断裂。PI-Sce I酶为一种较常用的归巢核酸内切酶,其识别序列较长,且在天然的酵母基因组中并不存在,因此,这种识别位点独特性及专一性使得其易于控制。另外,编码该酶的基因表达是受MET3启动子的调控的,这就使得此技术更加适用于条件性、时空特异性的染色体剪切。通过特定的启动子在合适的时间调节酶切作用,防止断裂染色体产生的时间不同而造成错开和丢失[15]。而在利用PI-Sce I酶进行切割后,被切割后残留的酶识别序列可能会干扰细胞中的端粒反应,或是被细胞内的相应机制认定为DNA损伤,这是需要考虑的方面[16]。总体看来,基因组重排方法在菌种改良等获取新的适应性基因组组成中具有极大的意义,也是一种技术上的巨大进步。
2.3 CRISPR/Cas9法产生染色体断裂
在细菌或古菌中发现的一种 RNA指导的自我防御的机制为遗传学家提供了新的有力武器,这就是CRISPR(Clustered regularly interspaced short palindromic repeat sequences)及Cas蛋白系统(CRISPR-associated systems)[17]。该系统的一大优势是利用单向导RNA (Single- guide RNA, sgRNA)引导新型核酸内切酶[18],大幅提高了切割位点的特异性。这一优势对于庞大的高等哺乳动物基因组编辑尤为重要。CRISPR/Cas9系统是目前应用较多的条件性基因打靶系统,其优点在于简单高效,且无需使用筛选标记。在酿酒酵母体系中,DiCarlo等[19]首先证明了利用CRISPR/Cas9系统介导的单基因敲除或敲入的高效性,其中用双链寡核苷酸片段实现了依赖同源重组的DSB的修复(图5)。Jakočiūnas等[20]成功地利用多CRISPR/Cas9系统在一步转化中同时完成了酵母基因组 5个位点的编辑。在后来的研究中,人们也逐渐地将这个系统应用到高通量的功能基因组学研究中[21]。对于复杂的高等哺乳动物来说,基于CRISPR/Cas系统的基因编辑技术更是革命性的突破,开启了包括人类在内动物遗传学研究和应用的新纪元。
如上介绍的这些染色体 DNA剪切技术获得断裂的染色体后,可用于进行构建新的染色体、基因删除或大规模重排(图6),也可以与其他的技术结合应用达到不同基因编辑目的。
2.4 染色体重排技术
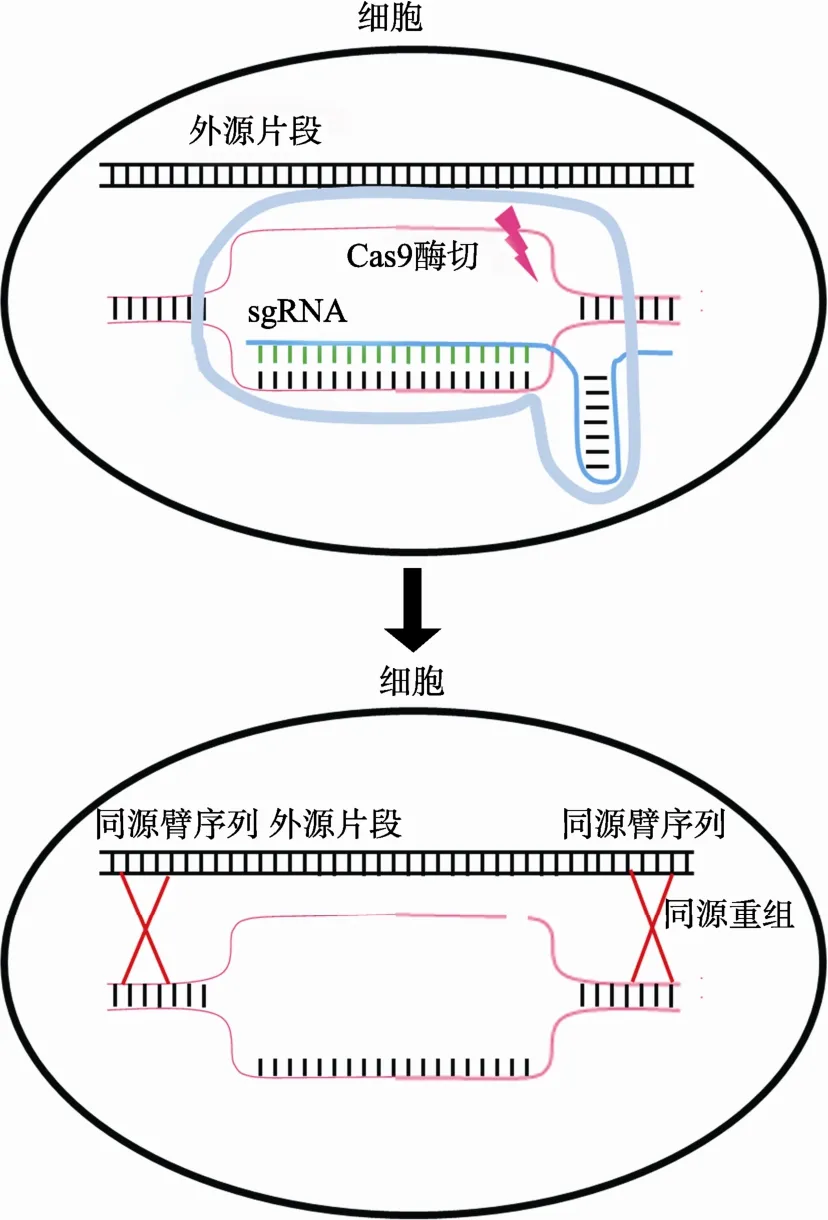
图5 借助CRISPR/Cas9系统向基因组中引入DSB

图6 断裂染色体的多种用途
酿酒酵母的许多表型均是由多基因控制的,尤其是那些在工业应用上有用途的性状,如乙醇耐受性、高温耐受性等。传统思路是对这些性状相关的基因进行逐个分析,现在可以采用基因组层面的方法来分析这些数量性状遗传,进而筛选出对不同环境具有较好适应性的经过“重排”的新的基因型。由此,在染色体切割技术的基础上,人们开创了染色体重排技术(Chromosome shuffling)。
染色体重排技术是为了实现将某一菌株的特定染色体片段用另一个菌株上的对应的区域来替换,探索亚染色体水平上的基因型-表型之间的关系。这个方法应用在酵母体系中时,主要是通过酵母的 a交配型及 α交配型之间的杂交来完成的。首先来自a型酵母的染色体和来自 α型酵母的染色体在相同的位置被剪切开,产生携带有不同筛选标记的断裂染色体;将两种交配型酵母进行杂交,得到的二倍体在完全培养基上传代培养后,剪切产生的部分微型染色体可能会丢失。这些细胞变成了半合子状态(Hemizygote),其丢失的那段染色体可能会被来自另一种类型的细胞的等位片段填补上,从而实现了等位序列的替换。该技术侧重于研究由一个选定的染色体片段区的替换或半合子的形成而引起的表型的改变,在实验室中或工业生产中均具有一定的应用价值。
2.5 基因组重建技术
在上述染色体重排技术中,为了研究某一片段或某几个片段的作用,对染色体DNA进行一处或几处的替换等编辑。如果对全基因组范围内的所有染色体在多个位点引入DSB,诱导其进行全基因组范围内的较多片段的替换或重排,即可实现基因组的重新排列,用于大规模地研究全基因组范围内的基因功能[23]。这种排列是随机的,可以创造多种不同的酵母基因组。1994年,Stemmer等[24]首先提出DNA重排技术(DNA shuffling)[24]。该技术起初在一定程度上借助了生物体自然进化过程中减数分裂期等位基因之间的DNA片段交换的模型。而后,1998年,为了研究生物体中控制性状的多基因之间的关系及作用情况,Stemmer等[25]又进一步提出了基因组重建技术(Genome reconstitution)。目前,这种技术主要应用在细菌和酵母体系中。通过上述染色体断裂技术,将基因组DNA断裂成多条微型染色体。这些断裂的染色体大片段可能以新的顺序排列组合,也可能以微型染色体的形式单独存在。在选择性培养基上生长时,微型染色体会有不同程度的丢失和重新组合比例,在一定的程度范围内可以增加随机得到不同结构的新基因组组成的多样性。这种方法可以快速地获得新型基因组。获得种类众多的基因型组成后,用不同的选择压力去筛选适应不同的环境条件的基因组组成。经过不同培养条件的筛选,可以得到若干种正向突变菌株,而这些正向突变的菌株再次杂交或减数分裂,可以进一步地筛选出某特定性状被增强的菌株[26]。这种方法有效地提高了菌株间基因组的重组几率,可以有效地缩短育种周期,较为迅速地获得优良突变菌株,在很大程度上弥补了经典诱变方法的缺陷。
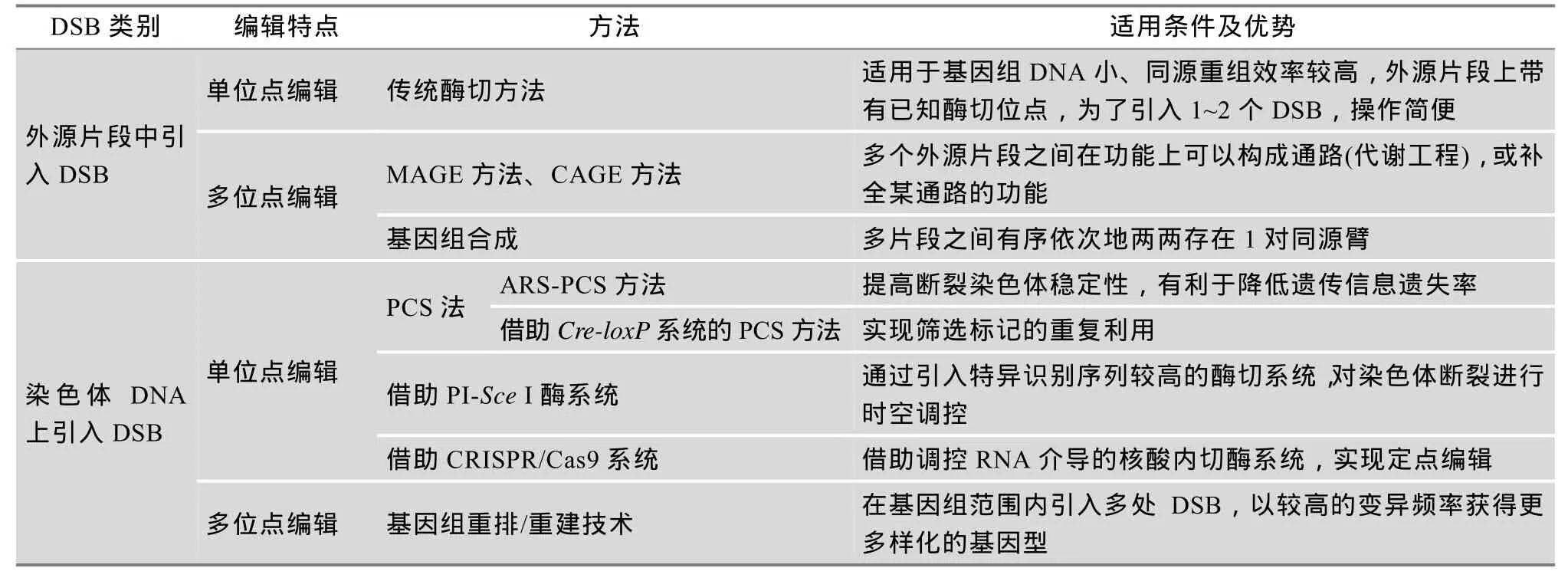
表1 不同基因及基因组编辑技术的特点
3 基因及基因组编辑技术的意义
基因及基因组编辑技术实际上是以人工方式来促进酿酒酵母等物种基因组的进化[6],加速了适应环境的基因型的产生。这种人工进化的方式在实验室中及工业上均有极大的促进作用[27]。譬如在工程菌株的育种和优化,获得在较酸性环境下仍能快速生长的乳酸高产菌株;培育多重抗性乙醇酵母,提高发酵性能等。
表1汇总了本文介绍的这几种不同的基因及基因组编辑技术。这些技术的发展及改进对基因功能研究及工业上的菌种培育具有极大的意义。目前在基因组层面进行多位点大规模操作的技术,如基因组重排技术,只能在酿酒酵母等少数遗传操作体系非常成熟的单细胞微生物中得以实现。而在高等生物中,受到基因组的高度复杂性及靶标特异性的限制,靶向基因敲除技术直到近几年受益于CRSPR/Cas技术才得以快速发展[28]。全基因组范围的功能基因组学发轫于酿酒酵母[23]。但在大约15年之后的今天,基于 CRSPR/Cas9技术的功能基因组学研究就扩展到了包括人类细胞在内的高等哺乳动物[21]。我们完全有理由相信,基因及基因组编辑技术还将不断创造新的技术革命,引领21世纪生命科学的发展。
[1] Lisby M, Rothstein R. Cell biology of mitotic recombination. Cold Spring Harb Perspect Biol, 2015, 7(3): a016535.
[2] Duina AA, Miller ME, Keeney JB. Budding yeast for budding geneticists: A primer on the Saccharomyces cerevisiae model system. Genetics, 2014, 197(1): 33-48.
[3] Rothstein RJ. One-step gene disruption in yeast. Methods Enzymol, 1983, 101: 202-211.
[4] Orr-Weaver TL, Szostak JW, Rothstein RJ. Yeast transformation: A model system for the study of recombination. Proc Natl Acad Sci USA, 1981, 78(10): 6354-6358.
[5] Huh WK, Falvo JV, Gerke LC, Carroll AS, Howson RW, Weissman JS, O'Shea EK. Global analysis of protein localization in budding yeast. Nature, 2003, 425(6959): 686-691.
[6] Botstein D, Fink GR. Yeast: An experimental organism for 21st century biology. Genetics, 2011, 189(3): 695-704.
[7] Wang HH, Isaacs FJ, Carr PA, Sun ZZ, Xu G, Forest CR, Church GM. Programming cells by multiplex genome engineering and accelerated evolution. Nature, 2009, 460(7257): 894-898.
[8] Pál C, Papp B, Pósfai G. The dawn of evolutionary genome engineering. Nat Rev Genet, 2014, 15(7): 504-512.
[9] Ma NJ, Moonan DW, Isaacs FJ. Precise manipulation of bacterial chromosomes by conjugative assembly genome engineering. Nat Prot, 2014, 9(10): 2285-2300.
[10] Sidhu YS, Cairns TC, Chaudhari YK, Usher J, Talbot NJ, Studholme DJ, Csukai M, Haynes K. Exploitation of sulfonylurea resistance marker and non-homologous end joining mutants for functional analysis in Zymoseptoria tritici. Fung Genet Biol, 2015, 79: 102-109.
[11] Baudin A, Ozier-Kalogeropoulos O, Denouel A, Lacroute F, Cullin C. A simple and efficient method for direct gene deletion in Saccharomyces cerevisiae. Nucleic Acids Res, 1993, 21(14): 3329-3330.
[12] Lu Q. Seamless cloning and gene fusion. Trends Biotechnol, 2005, 23(4): 199-207.
[13] Sugiyama M, Ikushima S, Nakazawa T, Kaneko Y, Harashima S. PCR-mediated repeated chromosome splitting in Saccharomyces cerevisiae. BioTechniques, 2005, 38(6): 909-914.
[14] Sugiyama M, Yamagishi K, Kim YH, Kaneko Y, Nishizawa M, Harashima S. Advances in molecular methods to alter chromosomes and genome in the yeast Saccharomyces cerevisiae. Appl Microbiol Biotechnol, 2009, 84(6): 1045-1052.
[15] Yamagishi K, Sugiyama M, Kaneko Y, Harashima S. Conditional chromosome splitting in Saccharomyces cerevisiae using the homing endonuclease PI-Scei. Appl Microbiol Biotechnol, 2008, 79(4): 699-706.
[16] Bianchi A, Negrini S, Shore D. Delivery of yeast telomerase to a DNA break depends on the recruitment functions of Cdc13 and Est1. Mol Cell, 2004, 16(1): 139-146.
[17] 李君, 张毅, 陈坤玲, 单奇伟, 王延鹏, 梁振, 高彩霞. CRISPR/Cas系统: RNA靶向的基因组定向编辑新技术.遗传, 2013, 35(11): 1265-1273.
[18] 殷利眷, 胡斯奇, 郭斐. CRISPR-Cas9基因编辑技术在病毒感染疾病治疗中的应用. 遗传, 2015, 37(5): 412-418.
[19] DiCarlo JE, Norville JE, Mali P, Rios X, Aach J, Church GM. Genome engineering in Saccharomyces cerevisiae using CRISPR-Cas systems. Nucleic Acids Res, 2013, 41(7): 4336-4343.
[20] Jakočiūnas T, Bonde I, Herrgård M, Harrison SJ, Kristensen M, Pedersen LE, Jensen MK, Keasling JD. Multiplex metabolic pathway engineering using CRISPR/Cas9 in Saccharomyces cerevisiae. Metab Eng, 2015, 28: 213-222.
[21] Shalem O, Sanjana NE, Zhang F. High-throughput functional genomics using CRISPR-Cas9. Nat Rev Genet, 2015, 16(5): 299-311.
[22] Mali P, Yang LH, Esvelt KM, Aach J, Guell M, DiCarlo JE,Norville JE, Church GM. Rna-guided human genome engineering via Cas9. Science, 2013, 339(6121): 823-826.
[23] Winzeler EA, Shoemaker DD, Astromoff A, Liang H, Anderson K, Andre B, Bangham R, Benito R, Boeke JD, Bussey H, Chu AM, Connelly C, Davis K, Dietrich F, Dow SW, El Bakkoury M, Foury F, Friend SH, Gentalen E, Giaever G, Hegemann JH, Jones T, Laub M, Liao H, Liebundguth N, Lockhart DJ, Lucau-Danila A, Lussier M, M'Rabet N, Menard P, Mittmann M, Pai C, Rebischung C, Revuelta JL, Riles L, Roberts CJ, Ross-MacDonald P, Scherens B, Snyder M, Sookhai-Mahadeo S, Storms RK, Véronneau S, Voet M, Volckaert G, Ward TR, Wysocki R, Yen GS, Yu KX, Zimmermann K, Philippsen P, Johnston M, Davis RW. Functional characterization of the S. cerevisiae genome by gene deletion and parallel analysis. Science, 1999, 285(5429): 901-906.
[24] Stemmer WP. DNA shuffling by random fragmentation and reassembly: In vitro recombination for molecular evolution. Proc Natl Acad Sci USA, 1994, 91(22): 10747-10751.
[25] Crameri A, Raillard SA, Bermudez E, Stemmer WPC. DNA shuffling of a family of genes from diverse species accelerates directed evolution. Nature, 1998, 391(6664): 288-291.
[26] Zhang YX, Perry K, Vinci VA, Powell K, Stemmer WPC, del Cardayré SB. Genome shuffling leads to rapid phenotypic improvement in bacteria. Nature, 2002, 415(6872): 644-646.
[27] Steensels J, Snoek T, Meersman E, Nicolino MP, Voordeckers K, Verstrepen KJ. Improving industrial yeast strains: Exploiting natural and artificial diversity. FEMS Microbiol Rev, 2014, 38(5): 947-995.
[28] Cong L, Ran FA, Cox D, Lin SL, Barretto R, Habib N, Hsu PD, Wu XB, Jiang WY, Marraffini LA, Zhang F. Multiplex genome engineering using CRISPR/Cas systems. Science, 2013, 339(6121): 819-823.
(责任编委: 刘钢)
From gene editing to genome reconstitution: evolving techniques in yeast
Chen Li, Huiqiang Lou
State Key Laboratory of Agro-Biotechnology, College of Biological Sciences, China Agricultural University, Beijing 100193, China
Homologous recombination is one of the main repair pathways in response to DNA double strand break (DSB) in eukaryotes. Based on this, a series of techniques to introduce DSB have been developed in order to edit the DNA sequence of genome. In eukaryotes, the gene editing technique was first established in S. cerevisiae by transformation of a foreign DNA fragment containing the sequence homologous to the targeted site more than thirty years ago. The core of all currently available editing methods lies in the introduction of DSB. Here, we try to convey a historic view of various editing techniques from its original version to the up-to-dated genome synthesis and reconstitution. We believe that this review will help to illustrate the trend of the development of genome editing techniques, which will provide a valuable reference for developing similar techniques in mammals.
DNA double strand break; gene editing; genome editing; genome synthesis/reconstitution; yeast
2015-06-09;
2015-07-24
国家自然科学基金项目(编号:31271331)和国家级大学生创新训练项目(编号:201410019015)资助
李晨,本科生,iGEM(International Genetically Engineered Machine Competition)中国农业大学团队队长。E-mail: li2006chen@126.com
楼慧强,博士,教授,研究方向:酵母分子遗传学。E-mail: lou@cau.edu.cn
10.16288/j.yczz.15-274
时间:2015-8-7 15:18:39
URL:http://www.cnki.net/kcms/detail/11.1913.R.20150807.1518.006.html

