CYC类基因在被子植物花发育中的研究进展
徐蕴如 李玥莹 逄洪波 张雨欣 刘羽莲
(沈阳师范大学化学与生命科学学院,沈阳 110034)
CYC类基因在被子植物花发育中的研究进展
徐蕴如 李玥莹 逄洪波 张雨欣 刘羽莲
(沈阳师范大学化学与生命科学学院,沈阳 110034)
CYCLOIDEA(CYC)类基因属于TCP基因家族成员,在花发育过程中具有重要作用。CYC类基因在被子植物进化过程中发生基因复制事件,形成CYC1、CYC2和CYC3三大分支,其中CYC2分支成员在花对称性形成方面具有主要调控作用。CYC1和CYC3分支成员开展研究较少。综述了国内外CYC类基因的研究现状及其存在问题,并对CYC类基因的研究前景做了展望。
CYC类基因;花对称性;TCP基因家族;进化
被子植物中存在着丰富多彩、形态各异的花。由于花是被子植物最精密、复杂的繁殖器官,这一直吸引着大批的生物学家,特别是进化发育生物学方面学者的注意力。研究表明,花发育的过程是由一个高度精密的调控网络所决定的[1-3]。在花发育的过程中,转录因子发挥着重要作用,它们时空表达的差异通常与花的形态变化密切相关[4,5]。在这些转录因子中,TCP家族转录因子由于在很多物种中具有调控细胞分裂和花对称性的作用而备受瞩目[6-12]。
TCP属于植物特有的一类转录因子,它是以玉米中的TEOSINTE BRANCHED1基因(TB1)[13]、金鱼草中的CYCLOIDEA基因(CYC)[6,7]和水稻中的PROLIFERATING CELL FACTORS 1 和2 基因(PCF1和PCF2)的首字母命名的[14]。TCP家族的蛋白包含一个高度保守的TCP结构域,即碱性螺旋-环-螺旋(bHLH)结构,此结构与DNA结合和蛋白的二聚化相关。其中,TB1在玉米的驯化过程中对于抑制侧芽的生长和雄花的形成具有决定作用[13];CYC在金鱼草中控制花的背部器官特征[6];PCF1和PCF2能够与水稻的PROLIFERATING CELL NUCLEAR ANTIGEN(PCNA)基因的启动子结合,而PCNA基因对于DNA的复制与修复、染色质的结构维持、染色体隔离及细胞周期循环都有重要作用[14]。
根据结构域的不同,TCP家族转录因子的成员可以分成两种类型:即TCP-P型和TCP-C型。而TCP-C型可以进一步分成ECE和CINCINNATA(CIN)两个分支[15,16]。其中,位于ECE分支中的TCP转录因子是被子植物特有的。CYC类基因属于TCP-C型中ECE分支上的成员,它除了具有TCP结构域、R结构域外,还含有核心真双子叶植物所特有的谷氨酸-半胱氨酸-谷氨酸的基序(ECE motif)[16-19](图1)。
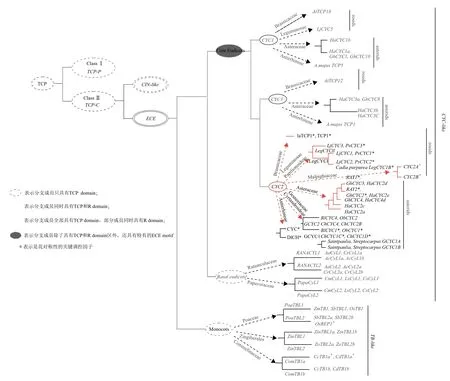
图1 TCP转录因子分类及CYC-类基因在被子植物中发生的代表性基因复制事件[6-63](图片根据文献[10]修改)
在植物的进化过程中,基因复制事件具有重要作用[20]。由于类群分化时间长短不同,各自经历了不同次数的基因复制事件,导致不同类群中含有的基因家族成员数目各不相同。在藻类的鼓藻(Cosmarium)、苔藓类的小立碗藓(Physcomitrella patens)和蕨类的卷柏(Selaginella)中,TCP仅含有5-6个家族成员[21],但在种子植物中,TCP家族成员多达十几个至几十个。基因复制事件的频繁发生能够产生很多重复基因,复制的重复基因或者丢失、或者在选择的作用下产生功能冗余、或者新功能化[22-27]。系统发育分析结果表明,被子植物中的CYC类基因发生过两次大的基因复制事件,形成CYC1、CYC2和CYC3三大分支[19],在不同类群中,每一分支又分别发生了次数不等的基因复制事件,最终形成不同拷贝[28-33](图1)。本文综述了国内外关于CYC类基因在被子植物中的研究现状及其存在问题,并对CYC类基因的研究前景进行了展望。
1 CYC2是被子植物花对称性关键调控因子
被子植物的花型,根据其对称面的有无,可以分为3种:辐射对称花(actinomophy),具有多个对称面;两侧对称花(zygomophy),只有一个对称面;不对称花(asymmetry),没有对称面[34-36]。野生型的金鱼草(Antirrhinum majus)具有典型的两侧对称花,是研究花对称性的模式植物。目前,关于金鱼草花型的分子调控机制已经研究得比较透彻,主要是由调控花背部特性的CYC基因、DICHOTOMA(DICH)基因、RADIALIS(RAD)基因和调控腹部特性的DIVARICATA(DIV)基因决定的[6,7]。其中,CYC和DICH基因均属于CYC2分支成员,它们起源于基因复制事件,具有冗余功能。CYC、DICH基因在金鱼草两侧对称花的发育过程中起着决定性作用,其中CYC基因起主要作用。
随着金鱼草花对称性分子调控机制的阐明,研究人员相继在其他类群中开展了大量的研究。如今,关于花对称性的研究已经取得很大进展,研究的对象主要涉及金鱼草及其近缘类群[6,7,12,37,38],菊科[31,39-42]、苦苣苔科[43-48]、豆科[9,49-51]、川续断目的忍冬科和川续断科[18,19,28,29]、毛茛目中的近缘类群罂粟科和紫堇科[52-54]、十字花科[55,56]、金虎尾科[57-59]等,甚至扩展到单子叶植物禾本科[30,60]、姜科[61]、鸭跖草[62]、兰科[63]中(图1),这些研究结果显示,在真双子叶植物,甚至在亲缘关系较远的基部被子植物、单子叶植物中,CYC2都是调控花对称性的关键因子。在被子植物进化过程中,CYC的同源基因被各自招募到不同类群中,参与调控两侧对称花的形成[12,64]。
CYC2基因通过调控花瓣和雄蕊的形状来参与花对称性的形成。在不同的类群中,CYC同源基因对器官的发育可能具有某些共同的作用,都与花瓣的生长有关,但功能具有物种差别,如在金鱼草中,CYC基因对背部花瓣的生长具有促进作用[6,7,65],但是在拟南芥、苦苣苔科等其他类群,CYC同源基因对于背部花瓣的生长具有抑制作用[19,44,48]。即使在同一类群中,CYC同源基因对不同器官的作用也可能不同。如金鱼草的CYC基因对背部花瓣的生长具有促进作用,而对背部雄蕊的生长具有抑制作用[6,7]。器官的形状不仅仅涉及到细胞的生长和分裂,还与器官的生长极性、区间生长速率及机械约束等因素有关,但是关于形态和相关调控因子的具体关系了解得很少,研究人员以金鱼草花为例,首次将计算数学与分子遗传学等数据相结合,从数量方面阐明了CYC等花对称性基因在金鱼草花型中发挥的调控作用[37,38]。
CYC2基因的表达模式和表达时间决定了花的最终对称性。从表达模式上看,两侧对称花类群中CYC2基因都集中在花器官的背部表达[7,48,50],而辐射对称花类群,表达模式相对比较复杂,CYC2基因的表达或是从背部扩增到整个花器官[47,59,66],或是不表达[67],或是先表达后消失[43,59,68]。在表达时间上,拟南芥(Arabidopsis thaliana)中CYC的同源基因AtTCP1早期在花原基背部短暂的表达没有影响辐射对称花的形成[68],无独有偶,五数苣苔(Bournea leiophylla)BlCYC基因在花发育早期的背部表达也没有影响最终辐射对称花的形成[43],最近在金虎尾科的研究中也发现了这一现象[59]。这些情况均说明了CYC2基因早期的表达模式不足以影响最终的花对称性。而烟叶唇柱苣苔ChCYC从花发育早期到晚期、持续不断的在背部器官的表达与两侧对称花的形成从另一方面证实了上述的观点[48],CYC2基因对花对称性形成的影响需要在整个花发育过程中持续不断的作用。
2 CYC类基因的其他作用
TCP转录因子在植物发育过程中具有广泛、重要的作用。因此,CYC类基因除了对花的对称性具有重要的调控作用之外,在其他方面也应该具有重要的作用。如参与控制叶片的生长[69]、花瓣融合[39]。在拟南芥中,CYC的同源基因AtTCP1参与了激素——油菜素内脂的代谢通路,通过调控油菜素内脂表达量的多少对植物发育的各个方面进行调控[70]。复制事件产生的重复基因经常发生亚功能化和新功能化,也暗示着CYC类基因功能的多样性。金鱼草中存在两个拷贝(CYC和DICH);而拟南芥24个TCP基因中只有3个是CYC类基因[19];豆科植物百脉根(Lotus japanicus)中存在4个CYC类基因[9];菊科植物向日葵(Helianthus annuus)中CYC类基因有10个拷贝[40],而非洲菊(Gerbera hybrida)中却只含有4个拷贝[39]。在这些重复基因中,以往的研究关注的都是CYC2分支的功能,而对于另外两大分支CYC1和CYC3开展的功能研究很少,只在拟南芥中发现CYC1同源基因TB1抑制玉米侧枝的形成[71],而CYC3的功能研究还未见报道。而对菊科植物向日葵、川续断科中的研究表明,CYC1基因在类群中再次发生了基因复制事件,并且基因表达模式发生了分化[28,40],这些都暗示了CYC1基因可能参与到植物发育的更多方面。
3 CYC2类基因在实践中的应用
花型是花的重要经济性状之一,一直是花卉育种研究的热点。目前在花型育种方面,较多采用的是传统育种,主要选取一些具有好看花型的植株进行杂交育种,关于花型的分子育种开展较少。CYC2类基因是花对称性的关键调控因子,对被子植物的花型(如花瓣的大小、形状、雄蕊的发育等)具有重要调控作用。因此,可以运用基因工程技术将CYC2类基因引入到花卉育种中来,从而培育出更多、更好的新品种。
豆科是严格的自花授粉植物,很多豆科植物是人类食物的来源,传统的大豆育种存在着异花传粉难题,开展CYC2类基因的研究有望解决这一问题[72]。已经在百脉根和豌豆中分离到调控花两侧对称的关键调控基因,可以通过花型改变,使其成为辐射对称花,增大异花授粉可能,从而提高大豆产量。
4 展望
从目前的研究中可以发现,尽管人们已经对某些类群中的CYC类基因进行了一些系统而细致的研究,但在CYC类基因的功能和进化的研究中仍然存在一些问题亟待解决。
4.1 开展系统的功能、进化研究
TCP转录因子在植物发育的过程中具有广泛、重要的作用[8,13,14,16,17,67,68],CYC类基因属于TCP-C型家族成员,应该广泛参与到植物的各种发育代谢过程中。但是目前已有的关于CYC类基因的研究几乎全部位于CYC2分支上,主要调控花的对称性形成,而关于CYC1和CYC3分支功能的研究很少,CYC1和CYC3分支上基因的功能仍不清楚[19]。因此,对CYC1和CYC3分支上的CYC类基因开展系统的功能、进化研究将有助于人们全面理解CYC类基因在被子植物发展史上的重要作用。
4.2 选取更多、更具代表性的植物来研究
被子植物核心真双子叶植物中已有的研究表明,CYC2基因的祖先表达模式是背特性,即在花器官的背部表达[48,68]。但在已有的、少数的几个单子叶植物(姜科和鸭跖草)研究中,CYC2基因的表达模式是腹特性的,即在花器官的腹部器官表达[61,62]。说明被子植物中CYC2基因的表达模式非常复杂,这就需要进一步加大采样类群,选取更多、更具有代表性的植物类群进行研究,以确定这一现象是否普遍,从而解释CYC2基因何时、如何被招募到各个类群中调控花的对称性。
4.3 增加对CYC类基因上游调控元件的研究
目前关于CYC类基因的研究主要关注的是CYC2同源基因的分离、系统发育分析、表达模式及功能分析,对于上游调控元件的工作开展得很少。Yang等[48]在烟叶唇柱苣苔中的研究表明ChCYC基因具有自我调控环,能够使其在花发育过程中持续表达。Carroll等的研究表明,基因表达的样式和功能的差异主要是由调控元件造成的,而不是编码基因[73,74]。因此推测,CYC2基因在不同类群的花原基中存在着时间、空间的表达式样多变性,以及功能多样化,这很有可能是由于其上游调控元件发生变化导致的,通过对古老的发育调控网络进行修饰、改变,从而致使被子植物的花型多种多样。所以,对代表性类群的CYC类基因的上游调控元件展开研究,特别是对CYC类基因的3个分支进行比较分析,将对人们理解被子植物CYC类基因的进化历史和功能分化起到非常重要的作用。
4.4 将新方法运用到CYC类基因的研究中
研究人员将进化发育生物学与计算机数学相结合,研究了金鱼草花的三维形态建成[37,38],揭示了关于花瓣形状形成的内在分子机制。在以后的研究中,可以运用更多的新方法,如比较基因组学等方法,以不同角度看待、解决问题。
相信随着研究的不断深入,人们将会深入了解CYC类基因在花发育中的作用,提高对被子植物花发育的理解和认识,从而为以后研究整个被子植物的进化和发育提供新思路。
[1]Zhao D, Yu Q, Chen C, et al. Genetic control of reproductive meristems[M]// McManus MT, Veit B, eds. Meristematic tissues in plant growth and development. Sheffield:Sheffield Academic Press, 2001:89-142.
[2]Soltis DE, Soltis PS, Albert VA, et al. Missing links:the genetic architecture of flowers and floral diversification[J]. Trends Plant Sci, 2002, 7(1):22-31.
[3]Kaufmann K, Melzer S, Theissen G. MIKC-type MADS-domain proteins:Structural modularity, protein interactions and network evolution in land plants[J]. Gene, 2005, 347(2):183-198.
[4]Doebley J. Genetics, development and plant evolution[J]. Curr Opin Genet Dev, 1993, 3(6):865-872.
[5]Doebley J, Lukens L. Transcriptional regulators and the evolution of plant form[J]. Plant Cell, 1998, 10(7):1075-1082.
[6] Luo D, Carpenter R, Vincent C, et al. Origin of floral asymmetry in Antirrhinum[J]. Nature, 1996, 383(6603):794-799.
[7]Luo D, Carpenter R, Copsey L, et al. Control of organ asymmetry in flowers of Antirrhinum[J]. Cell, 1999, 99(4):367-376.
[8]Cubas P, Lauter N, Doebley J, et al. The TCP domain:a motif found in proteins regulating plant growth and development[J]. Plant J,1999, 18(2):215-222.
[9]Feng XZ, Zhao Z, Tian ZX, et al. Control of petal shape and floral zygomorphy in Lotus japonicas[J]. Proc Natl Acad Sci USA, 2006,103(13):4970-4975.
[10] Busch A, Zachgo S. Flower symmetry evolution:towards understanding the abominable mystery of angiosperm radiation[J]. Bioessays, 2009, 31(11):1181-1190.
[11]Hileman LC, Cubas P. An expanded evolutionary role for flower symmetry genes[J]. J Biol, 2009, 8(10):90.
[12]Preston JC, Hileman LC. Developmental genetics of floral symmetry evolution[J]. Trends Plant Sci, 2009, 14(3):147-154.
[13]Doebley J, Stec A, Hubbard L. The evolution of apical dominance in maize[J]. Nature, 1997, 386(6624):485-488.
[14]Kosugi S, Ohanshi Y. PCF1 and PCF2 specifically bind to cis elements in the rice PROLIFERATING CELL NUCLEAR ANTIGEN gene[J]. Plant Cell, 1997, 9(9):1607-1619.
[15]Cubas P. Floral zygomorphy, the recurring evolution of successful trait[J]. Bioessays, 2004, 26(11):1175-1184.
[16]Martin-Trillo M, Cubas P. TCP genes:a family snapshot ten years later[J]. Trends Plant Sci, 2009, 15:31-39.
[17] Cubas P. Role of TCP genes in the evolution of morphological characters in angiosperms[M]//Hawkins JA, Cronk QCB,Bateman RM, eds. Developmental genetics and plant evolution. London, Taylor and Francis:CRC Press, 2002:247-266.
[18]Howarth DG, Donoghue MJ. Duplication in CYC-like genes from dipsacales correlates with floral form[J]. Int J Plant Sci, 2005,166(3):357-370.
[19]Howarth DG and Donoghue MJ. Phylogenetic analysis of the“ECE”(CYC/TB1)clade reveals duplications predating the core eudicots[J]. Proc Natl Acad Sci USA, 2006, 103(24):9101-9106.
[20]Lynch M, Conery JS. The evolutionary fate and consequences of duplicate genes[J]. Science, 2000, 290(5494):1151-1155.
[21]Navaud O, Dabos P, Carnus E, et al. TCP transcription factors predate the emergence of land plants[J]. J Mol Evol, 2007, 65(1):23-33.
[22]Ohta T. Time for acquiring a new gene by duplication[J]. Proc Natl Acad Sci USA, 1988, 85(10):3509-3512.
[23]Lynch M, Force A. The probability of duplicate gene preservation by subfunctionalization[J]. Genetics, 2000, 154(1):459-473.
[24]Vandenbussche M, Theissen G, Van de Peer Y, et al. Structural diversification and neo-functionalization during floral MADS-box gene evolution by C-terminal frameshift mutations[J]. Nucleic Acids Res, 2003, 31(15):4401-4409.
[25]Taylor JS, Raes J. Duplication and divergence:the evolution of new genes and old ideas[J]. Annu Rev Genet, 2004, 38:615-643.
[26]Vandenbussche M, Zethof J, Royaert S, et al. The duplicated B-class heterodimer model:whorl-specific effects and complex genetic interations in Petunia hybrid flower development[J]. Plant Cell,2004, 16(3):741-754.
[27]Moore RC, Purugganan MD. The evolutionary dynamics of plant duplicate genes[J]. Curr Opin Plant Biol, 2005, 8(2):122-128.
[28]Carlson SE, Howarth DG, Donoghue MJ. Diversification of CYCLOIDEA-like genes in Dipsacaceae(Dipsacales):implications for the evolution of capitulum inflorescences[J]. BMC Evol Biol, 2011, 11:325.
[29] Howarth DG, Martins T, Chimney E, et al. Diversification of CYCLOIDEA expression in the evolution of bilateral flower symmetry in Caprifoliaceae and Lonicera(Dipsacales)[J]. Ann Bot London, 2011, 107(9):1521-1532.
[30]Mondragon-Palomino M, Trontin C. High time for a roll call:gene duplication and phylogenetic relationships of TCP-like genes in monocots[J]. Ann Bot London, 2011, 107(9):1533-1544.
[31]Tähtiharju S, Rijpkema AS, Vetterli A, et al. Evolution and diversification of the CYC/TB1 gene family in Asteraceae—a comparative study in gerbera(Mutisieae)and sunflower(Heliantheae)[J]. Mol Biol Evol, 2012, 29(4):1155-1166.
[32]Citerne H, Le Guilloux M, Sannier J, et al. Combining phylogenetic and syntenic analyses for understanding the evolution of TCP ECE genes in eudicots[J]. PLoS One, 2013, 8(9):e74803.
[33]Florian J, Guillaume C, Martine LG, et al. Specific duplication and dorsoventrally asymmetric expression patterns of cycloidea-like genes in zygomorphic species of ranunculaceae[J]. PLoS One,2014, 9(4):e95727.
[34]Endress PK. Evolution of floral symmetry[J]. Curr Opin Plant Biol, 2001, 4(1):86-91.
[35]Endress PK. The immense diversity of floral monosymmetry and asymmetry across angiosperms[J]. Botan Rev, 2012, 78:345-397.
[36] Citerne H, Jabbour F, Nadot S, et al. The evolution of floral symmetry[M]// Kader JC, Delseny M, eds. Advances in Botanical Research. United States:Academic Press Inc, 2010, 54:85-137.
[37]Green AA, Kennaway R, Hanna AI, et al. Genetic control of organ shape and tissue polarity[J]. PLoS Biol, 2010, 8(11):e1000537.
[38]Cui ML, Copsey L, Green AA et al. Quantitative control of organ shape by combinatorial gene activity[J]. PLoS Biol, 2010, 8(11):e1000538.
[39]Broholm SK, Tahtiharju S, Laitinen RA, et al. A TCP domain transcription factor controls flower type specification along the radial axis of the Gerbera(Asteraceae)inflorescence[J]. Proc Natl Acad Sci USA, 2008, 105(26):9117-9122.
[40]Chapman MA, Leebens-Mack JH, Burke JM. Positive selection and expression divergence following gene duplication in the sunflower CYCLOIDEA gene family[J]. Mol Biol Evol, 2008, 25(7):1260-1273.
[41]Kim M, Cui ML, Cubas P, et al. Regulatory genes control a key morphological and ecological trait transferred between species[J]. Science, 2008, 322(5904):1116-1119.
[42]Chapman MA, Tang SX, Draeger D, et al. Genetic analysis of floral symmetry in Van Gogh's sunflowers reveals independent recruitment of CYCLOIDEA genes in the Asteraceae[J]. PLoS Genet, 2012, 8(3):e1002628.
[43]Zhou XR, Wang YZ, Smith JF, et al. Altered expression patterns of TCP and MYB genes relating to the floral developmental transition from initial zygomorphy to actinomorphy in Bournea(Gesneriaceae)[J]. New Phytol, 2008, 178(3):532-543.
[44]Gao Q, Tao JH, Yan D et al. Expression differentiation of CYC-like floral symmetry genes correlated with their protein sequence divergence in Chirita heterotricha(Gesneriaceae)[J]. Dev Genes Evol, 2008, 218(7):341-351.
[45]Du ZY, Wang YZ. Significance of RT-PCR expression patterns of CYC-like genes in Oreocharis benthamii(Gesneriaceae)[J]. J S E,2008, 46(1):23-31.
[46]Song CF, Lin QB, Liang RH, et al. Expressions of ECE-CYC2 clade genes relating to abortion of both dorsal and ventral stamens in Opithandra(Gesneriaceae)[J]. BMC Evol Biol, 2009, 9:244.
[47]Pang HB, Sun QW, He SZ, et al. Expression pattern of CYC-like genes relating to a dorsalized actinomorphic flower in Tengia(Gesneriaceae)[J]. J S E, 2010, 48(5):309-317.
[48]Yang X, Pang HB, Liu BL, et al. Evolution of double positive autoregulatory feedback loops in CYCLOIDEA2 clade genes is associated with the origin of floral zygomorphy[J]. Plant Cell,2012, 24(5):1834-1847.
[49] Citerne HL, Luo D, Pennington RT, et al. A phylogenomic investigation of CYCLOIDEA-like TCP genes in the Leguminosae[J]. Plant Physiol, 2003, 131(3):1042-1053.
[50] Wang Z, Luo YH, Li X, et al . Genetic control of floral zygomorphy in pea(Pisum sativum L. )[J]. Proc Natl Acad Sci USA, 2008,105(30):10414-10419.
[51] Xu SL, Luo YH, Cai ZG, et al. Functional diversity of CYCLOIDEA-like TCP genes in the control of zygomorphic flower development in Lotus japonicus[J]. J Interg Plant Biol, 2013, 55(3):221-231.
[52]Kölsch A, Gleissberg S. Diversification of CYCLOIDEA-like TCP genes in the basal eudicot families Fumariaceae and Papaveraceae s. str[J]. Plant Biol, 2006, 8(5):680-687.
[53]Damerval C, Guilloux ML, Jager M, et al. Diversity and evolution of CYCLOIDEA-Like TCP genes in relation to flower development in Papaveraceae[J]. Plant Physiol, 2007, 143(2):759-772.
[54] Damerval C, Citerne H, Le Guilloux M, et al. Asymetric morphogenetic cues along the transverse plane:shift from dissymmetry to zygomorphy in the flower of Fumarioideae[J]. Am J Bot, 2013, 100(2):391-402.
[55]Busch A, Zachgo S. Control of corolla monosymmetry in the Brassicaceae Iberis amara[J]. Proc Natl Acad Sci USA, 2007,104(42):16714-16719.
[56]Busch A, Horn S, Muhlhausen A, et al. Corolla monosymmetry:evolution of a morphological novelty in the Brassicaceae family[J]. Mol Biol Evol, 2012, 29(4):1241-1254.
[57]Zhang WH, Kramer EM, Davis CC. Floral symmetry genes and the origin and maintenance of zygomorphy in a plant-pollinator mutualism[J]. Proc Natl Acad Sci USA, 2010, 107(14):6388-6393.
[58]Zhang WH, Kramer EM, Davis CC. Similar genetic mechanisms underlie the parallel evolution of floral phenoltypes[J]. PLoS ONE, 2012, 7(4):e36033.
[59]Zhang WH, Steinmann VW, Nikolov L. et al. Divergent genetic mechanisms underlie reversals to radial floral symmetry from diverse zygomorphic flowered ancestors[J]. Frontiers in Plant Science, 2013, 4(302):1-12.
[60]Yuan Z, Gao S, Xue DW. RETARDED PALEA1 controls palea development and floral zygomorphy in rice[J]. Plant Physiol,2009, 149(1):235-244.
[61]Bartlett ME, Specht CD. Changes in expression pattern of the teosinte branched1-like genes in the Zingiberales provide a mechanism for evolutionary shifts in symmetry across the order[J]. Am J Bot, 2011, 98(2):227-243.
[62]Preston JC, Hileman LC. Parallel evolution of TCP and B-class genes in Commelinaceae flower bilateral symmetry[J]. EvoDevo, 2012, 3:6.
[63]Mondragon-Palomino M, Theissen G. Why are orchid flowers so diverse? reduction of evolutionary constraints by paralogues of class B floral homeotic genes[J]. Ann Bot, 2009, 104(3):583-594.
[64]Hileman LC. Bilateral flower symmetry-how, when and why?[J]. Curr Opin Plant Biol, 2014, 17:146-152.
[65]Costa MMR, Fox S, Hanna AI, et al. Evolution of regulatory interactions controlling floral asymmetry[J]. Development, 2005,132(22):5093-5101.
[66]Citerne HL, Pennington RT, Cronk QC. An apparent reversal in floral symmetry in the Legume Cadia is a homeotic transformation[J]. Proc Natl Acad Sci USA, 2006, 103(32):12017-12020.
[67]Cubas P, Vincent C, Coen E. An epigennetic mutation responsible for natural variation in floral symmetry[J]. Nature, 1999, 401(6749):157-161.
[68]Cubas P, Coen E, Zapater JM. Ancient asymmetries in the evolution of flowers[J]. Curr Biol, 2001, 11(13):1050-1052.
[69] Koyama T, Sato F, Ohme-Takagi M. A role of TCP1 in the longitudinal elongation of leaves in Arabidopsis[J]. Biosci Biotechnol Biochem, 2010, 74(10):2145-2147.
[70] Guo Z, Fujioka S, Blancaflor EB, et al. TCP1 modulates brassinosteroid biosynthesis by regulating the expression of the key biosynthetic gene DWARF4 in Arabidopsis thaliana[J]. Plant Cell, 2010, 22(4):1161-1173.
[71]Aguilar-Martínez JA, Poza-Carrión C, Cubas P. Arabidopsis BRANCHED1 acts as an integrator of branching signals within axillary buds[J]. Plant Cell, 2007, 19:458-472.
[72]李家洋. 植物花对称性发育研究的进展:从理论到应用的双重价值[J]. 分子植物育种, 2006, 4(6):751-752.
[73] Carroll SB, Prud'Homme B, Gompel N. Regulating evolution[J]. Scientific American, 2008, 298(5):60-67.
[74] Carroll SB. Endless forms:the evolution of gene regulation and morphological diversity[J]. Cell, 2000, 101(6):577-580.
(责任编辑 狄艳红)
Progress on Study of CYC-like Genes During the Floral Development of Angiosperm
Xu Yunru Li Yueying Pang Hongbo Zhang Yuxin Liu Yulian
(College of Chemistry and Life Science,Shenyang Normal University,Shenyang 110034)
CYCLOIDEA(CYC)-like genes are members of TCP family and play an important role during floral development. CYC-like genes underwent gene duplication deep within angiosperm phylogeny, they diversified into three major clades, CYC1, CYC2, CYC3, and CYC2 clade genes are main regulators in floral symmetry. But the studies on CYC1 and CYC3 are limited. In this paper, recent insights into CYC-like genes and the prospective of the study on CYC genes were reviewed.
CYC gene;floral symmetry;TCP gene family;evolution
10.13560/j.cnki.biotech.bull.1985.2015.04.002
2014-07-18
国家自然科学基金项目(31100176),沈阳师范大学生态与环境研究中心主任基金项目(EERC-K-201404),沈阳师范大学大学生创新创业训练计划项目
徐蕴如,女,E-mail:1359404266@qq.com
逄洪波,女,博士,讲师,研究方向:植物进化发育生物学;E-mail:panghongbo800206@163.com

