利用表型芯片系统高通量分析H37Ra与H37Rv差异表型
王艺红,郭庆龙,周凤竹,苟宗超,王洪海,张雪莲
复旦大学生命科学学院遗传工程国家重点实验室,上海 200433
·论著·
利用表型芯片系统高通量分析H37Ra与H37Rv差异表型
王艺红,郭庆龙,周凤竹,苟宗超,王洪海,张雪莲
复旦大学生命科学学院遗传工程国家重点实验室,上海 200433
摘要:H37Rv是结核分枝杆菌标准有毒株,H37Ra是从H37Rv获得的稳定减毒株,但目前H37Ra毒力减弱原因尚不完全清楚。本研究利用表型芯片系统,高通量分析H37Ra生长表型,并与H37Rv表型比较,筛选两菌株表型差异,分析与H37Ra毒力减弱可能的相关表型及分子机制。结果发现,与H37Rv相比,H37Ra耐酸及耐渗透压能力显著下降,且不能利用丁二酸单甲酯和吐温40作为碳源。结核分枝杆菌耐酸能力直接影响其在吞噬体中的生存和代谢,耐高渗能力影响其必需营养物质的跨膜运输,代谢途径的改变影响其在宿主内的能量摄取,三者改变均可能与H37Ra毒力减弱相关。
关键词:结核分枝杆菌;表型;毒力
结核病是长期危害人类健康的主要传染病之一。2015年世界卫生组织(World Health Organization,WHO)数据表明,2014年全球约150万人死于结核病[1]。结核分枝杆菌(Mycobacteria,M.)是引起结核病的重要病原菌,其中H37Rv为结核分枝杆菌标准有毒株,并于1998年完成全基因组测序,标志着对结核分枝杆菌的研究进入后基因组时代[2]。
H37Ra是1935年由H37Rv减毒获得的稳定菌株,两者形态特征存在重要差异。H37Ra表面隆起[3],失去索状结构[4],且不与中性红染料结合[5]。1954年,Mackaness等[6]研究发现H37Ra在巨噬细胞内生存能力下降;1964年,Larson等[7]发现H37Ra在被感染小鼠中毒力丧失;1973年,Alsaadi等[8]提出H37Ra在被感染豚鼠中不具备毒力,但其毒力减弱的原因并不清楚。长期以来,多个团队对H37Ra毒力减弱可能的分子机制进行了研究[9-12]。1999年,Brosch等[13]发现重复序列IS6110能催化介导H37Rv基因组片段缺失和突变。2008年,Zheng等[14]确定了H37Ra ATCC 25177的全基因组序列,发现H37Ra基因组存在53个位点的插入和21个位点的缺失,这些突变直接影响结核分枝杆菌的细胞膜结构、生长过程及初级代谢等各项表型,可能为其毒力减弱的重要物质基础。
结核分枝杆菌生长缓慢,进行大规模的表型分析一直是较大的难题。2001年,Bochner等[15]以大肠埃希菌( Escherichia,E.)为模式细胞,提出并发展了表型芯片(Phenotype MicroArray,PM)系统。2008年,Viti等[16]对PM系统进行改进,成功将其应用于牛分枝杆菌(Mycobacterium,M.)菌株表型的分析。2013年,Khatri等[17]利用PM系统对H37Rv、牛分枝杆菌及结核分枝杆菌北京株的差异表型进行了研究。至今,PM系统已被用于1 000多种细胞表型的相关研究。
本研究利用PM系统,高通量获得减毒株H37Ra的代谢表型,与H37Rv比较,探讨H37Ra毒力减弱株的代谢表型特点,并分析遗传学差异,以获得可能与结核分枝杆菌毒力相关的信息。
1材料与方法
1.1材料
结核分枝杆菌H37Ra菌株由本实验室保存。主要试剂有IF-0a GN/GP Base(1.2×)(Biolog)、Biolog Redox Dye Mix G(100×)(Biolog)、Middlebrook 7H9 Broth(Difco)、Middlebrook 7H10 Agar(Difco),其他化学试剂均为国家分析纯(analytical reagent,AR)产品。PM板有PM1、PM2A、PM3B、PM5、PM9、PM10、PM11、PM12,购自Biolog公司。
1.2方法
1.2.1H37Ra菌悬液的制备为减少表型芯片板实验中的数据背景,对H37Ra菌悬液的制备进行改进。具体操作:H37Ra于7H10-10% OADC(/)固体板中培养3周,用接种环刮下菌苔,置于7H9-0.025%四丁酚醛,以4 mm玻璃珠振荡破碎菌团,接种于7H9-0.05%吐温80-10% OADC(//),37 ℃静置培养至对数中期,600 nm处光密度(optical density,)为0.40~0.60。离心收集菌体,用pH 6.8的0.025%四丁酚醛-20 mmol/L磷酸盐缓冲液(phosphate buffered saline,PBS)洗两遍,再用0.025%四丁酚醛-20 mmol/L PBS重悬菌体。
1.2.2表型芯片板对H37Ra表型的高通量检测表型芯片板高通量检测方法由Biolog公司提供[16,18]。具体方法:根据1.2.1制备菌悬液,25 ℃培养24 h,离心去除PBS,以IF-0a GN/GP Base重悬菌体,调整菌悬液600=0.60,于PM板各孔加入100 μL混合培养液,每孔菌悬液600=0.044,密封。37 ℃避光培养168 h,培养结束后测定各孔600。实验重复两个平行组。
1.2.3H37Ra利用碳源的验证分析根据1.2.1制备菌悬液,调整菌悬液600=0.60,接种于分别以0.5%葡萄糖、0.5%甘油、10 mmol/L乙酸盐、10 mmol/丙酸盐、0.1%吐温80为单一碳源的7H9培养基中,初始菌悬液600=0.01。37 ℃静置培养,每96 h测定菌悬液的600值。实验重复三个平行组。
1.2.4H37Ra耐受pH范围的验证分析根据1.2.1制备菌悬液,调整菌悬液600=0.60,将制备好的菌悬液分别接种于pH 4.5、pH 5.0、pH 6.0、pH 7.0、pH 8.0及常规pH的7H9-0.05%吐温80-10%OADC(//)培养基中,初始菌悬液630=0.044。37 ℃静置培养,以Dye Mix G为指示剂,每48 h测定菌液的630值。实验重复三个平行组。
2结果
2.1PM系统对H37Ra表型的高通量检测
PM系统由20块(PM1~PM20)预装有不同底物的微孔板组成,可为H37Ra提供不同的生长环境。在H37Ra培养过程中,细胞呼吸产生的NADH能导致指示剂Dye Mix G中无色的四唑染料发生不可逆衰减,转化为紫色的甲瓒,甲瓒于630 nm波长处存在特异性吸收。细胞的呼吸作用越强,产生的NADH越多,四唑染料减少量越多,培养混合物于630 nm波长处的特异性吸收越强。通过分光光度计检测630值,能快速有效监测细胞在PM系统不同环境中新陈代谢的能力[19-20]。
其中,PM1和PM2A各提供一个阴性对照孔(A1)及95种不同的单一碳源,可用于分析H37Ra对不同碳源的摄取和利用能力;PM3B提供一个阴性对照孔(A1)和95种不同的氮源组合,可用于分析H37Ra对不同氮源的摄取和利用能力;PM5用于H37Ra对不同营养补充物摄取和利用的分析;PM9可用于细胞耐受渗透压环境的分析;PM10可用于细胞耐受pH环境的分析;PM11、PM12可用于细胞耐受抗生素和胁迫环境的分析。本研究共分析了H37Ra在64种不同环境中的细胞表型。
H37Ra于PM系统中培养168 h后,测定764个微孔中培养混合物的630值。结果表明,在碳源板PM1和 PM2中,H37Ra能有效利用多种底物作为碳源,其中利用甘油(PM1-B3)、吐温80(PM1-E5)、-葡萄糖(PM1-C9)、丙酮酸(PM1-H8)、乙酸(PM1-C8)、乳酸(PM1-B9)、海藻糖(PM1-A10)、-谷氨酸(PM1-B12)和丁酸(PM2A-D12)的效率较高;在氮源板PM3中,能有效利用谷氨酸(PM3B-A12)、丙氨酸(PM3B-A7)、天冬酰胺(PM3B-A9)、谷氨酰胺(PM3B-B1)和胞嘧啶核苷(PM3B-F4)作为氮源;在渗透压板PM9中,能耐受1% NaCl和2% NaCl的渗透压,在3% NaCl的渗透压环境中呼吸微弱,在4% NaCl的渗透压环境中不能进行正常代谢;同时,H37Ra于酸性板PM10中不能耐受pH 3.5~5.5的环境,呼吸极其微弱,在pH 6.0~7.0环境中代谢能力增强(图1)。
2.2H37Ra利用碳源的验证结果
为进一步分析H37Ra利用不同碳源的生长曲线,同时对PM系统的实验结果进行验证,配制包括葡萄糖、甘油、乙酸盐、丙酸盐、吐温80在内的单一碳源培养基。利用PM系统高通量检测H37Ra表型,芯片板通过监测细胞呼吸产生的NADH量变化,来评价细胞在PM系统不同环境中新陈代谢的能力。该系统灵敏、快速,可设置较高初始菌液密度[16,18],以实现快速检测,整个实验周期仅7 d。本研究中,为全面持续观察H37Ra不同生长阶段利用不同单一碳源生长繁殖的能力,获得完整的生长曲线,采取较低的H37Ra接菌量,不同单一碳源培养基的初始菌悬液600=0.01[21]。

图1PM系统中pH及NaCl浓度对H37Ra代谢的影响
Fig.1Effects of pH and NaCl concentration on relative dye reduction in H37Ra
结果表明,H37Ra能充分利用葡萄糖和甘油作为碳源,生长情况与完全培养基几乎一致,培养24 d,H37Ra持续生长且生长速度较快。H37Ra能有效利用乙酸盐作为碳源,培养前16 d,H37Ra持续生长且生长速度较快;培养第16~24天,生长趋于平缓。H37Ra利用丙酸及吐温80作为碳源的能力较弱(图2)。在液体培养条件下,H37Ra在不同单一碳源中的生长能力:甘油>葡萄糖>乙酸盐>丙酸盐或吐温80。在PM系统中,H37Ra利用碳源代谢的能力:甘油>葡萄糖>乙酸盐>吐温80>丙酸盐。两者对吐温80和丙酸盐的利用结果存在细微差异,可能与两种检测方法中碳源浓度设置存在差异有关。同时,PM系统通过监测细胞新陈代谢能力分析细胞对底物的利用能力,整个系统更敏感,能有效区分吐温80与丙酸利用效率的细微差异。总体而言,生长曲线测定结果与PM系统结果一致,两者能相互补充验证。

图2H37Ra在不同单一碳源中的生长情况
Fig.2Growth curve of H37Ra in different carbon substrates
2.3H37Ra的pH耐受范围
配制不同pH范围的完全培养基,进一步分析H37Ra可耐受的pH范围,同时对PM系统实验结果进行验证。结果表明(图3),以H37Ra在完全培养基(pH 6.9)中的呼吸情况为阳性对照,H37Ra在pH 7.0的培养基中具有较强的呼吸代谢,在pH 6.0的培养基中呼吸代谢开始减弱,在pH 5.0及pH 4.5的培养基中不能实现正常呼吸代谢功能。在PM系统研究中,H37Ra不能耐受pH 3.5~5.5的环境,两者结果一致,能相互补充验证。
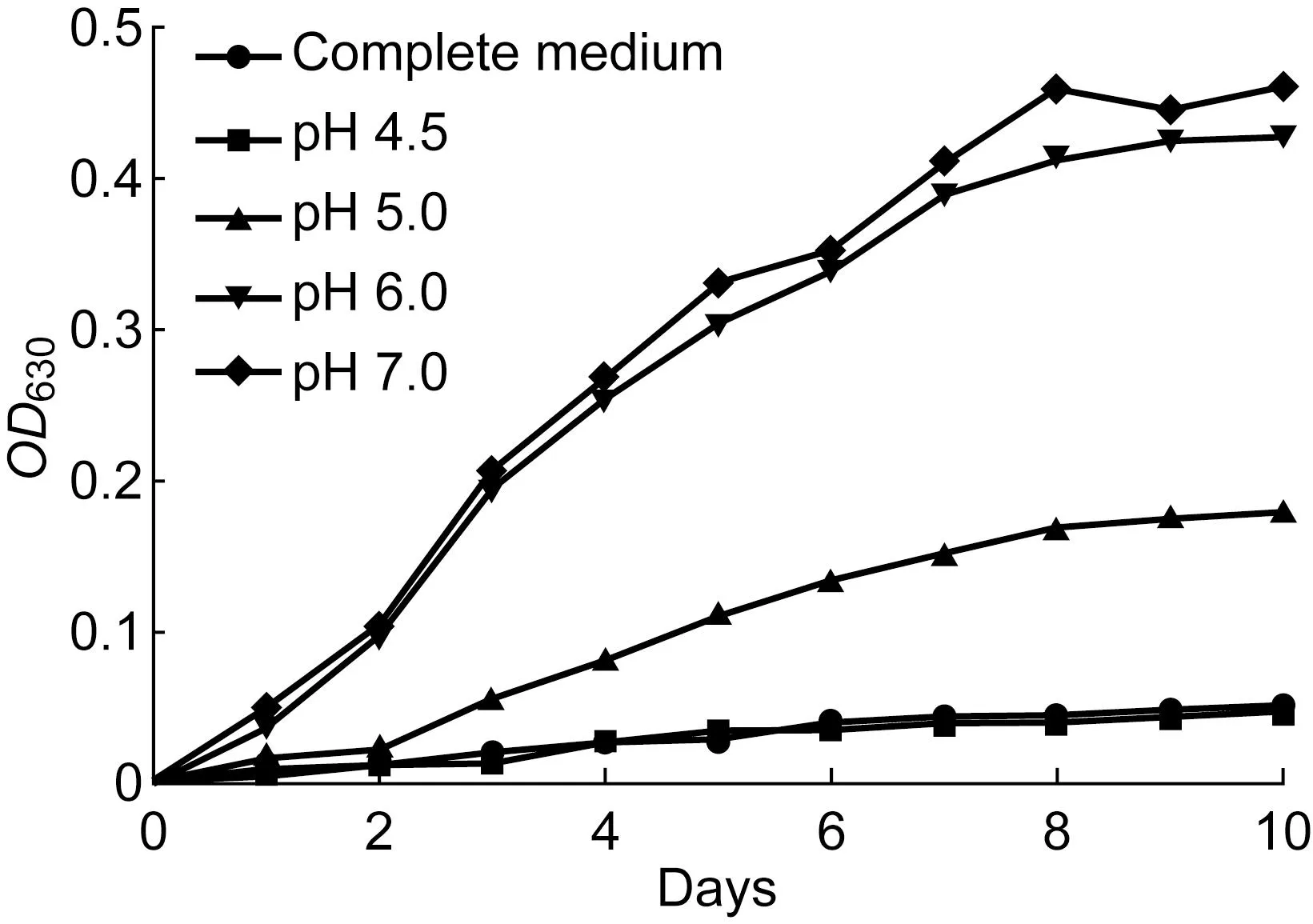
图3H37Ra在不同pH条件下的呼吸代谢情况
Fig.3Metabolism of H37Ra at different pH values
3讨论
H37Rv是结核分枝杆菌标准有毒株,H37Ra是从H37Rv获得的稳定减毒株。长期以来,多个团队致力于通过差异基因组学的分析方法,寻找H37Ra减毒的可能机制,但目前尚不完全清楚。2008年,Zheng等[14]提出H37Ra基因组中的突变直接影响结核分枝杆菌的表型特征、生长及初级代谢,可能为H37Ra毒力减弱的重要物质基础,为减毒机制研究提供了新的思路。
本研究利用PM系统高通量分析H37Ra表型,筛选H37Ra在碳源利用、氮源利用、耐受渗透压及耐受pH等方面的主要表型,并比较H37Rv与H37Ra的差异表型,探究H37Ra毒力减弱的可能机制,从而获得H37Rv中可能与毒力相关的基因。
本研究表明,H37Ra于PM系统中不能耐受pH 3.5~5.5的环境(图1)。2013年,Khatri等[17]利用PM系统对H37Rv的表型进行高通量分析,发现H37Rv在PM系统中能耐受pH 5.5的酸性环境。Portaels等[22]及Chapman等[23]也对H37Rv耐受pH范围进行了研究,表明H37Rv能耐受pH 5.0~8.4的环境。两种菌株相比,H37Ra较37Rv 耐酸能力显著下降。
结核分枝杆菌感染宿主后,通过FcR和CR3等表面受体识别,经胞吞作用进入吞噬细胞,聚集在吞噬体中,吞噬体内环境被激活,逐渐酸化,pH从6.2降至5.5[24]。吞噬体内的酸性环境极不利于结核分枝杆菌生存,直接影响其生长代谢。为了更好地适应环境,结核分枝杆菌H37Rv表现出一定程度的耐酸能力,从而具有更强的毒力;而H37Ra的耐酸能力下降,这可能是其毒力下降的原因之一。在人巨噬细胞模型中,H37Ra与H37Rv的存活率、生长速度及对巨噬细胞形态的影响均存在显著差异[25-26],且H37Ra干扰吞噬体内环境的能力受损[27]。Abramovitch等[28]研究发现,双组分调控系统PhoPR能感受低pH信号,并诱导aprABC表达,帮助细菌适应酸性环境。同时,在酸性刺激下,受PhoP调控的基因lipF、2、1、Rv2390c、whiB6等表达普遍上调[29]。H37Ra与H37Rv差异基因组学研究发现,双组分调控系统PhoPR存在基因突变[14],同时有研究确证phoPR是结核分枝杆菌的重要毒力基因[30]。因此,参与酸性调节和耐受的相关基因可能是与结核分枝杆菌毒力有关的基因。
在PM系统中,本研究还发现H37Ra失去了对高渗透压的耐受能力。H37Ra在3% NaCl的渗透压环境中呼吸极其微弱(图1),而Khatri等[17]研究表明H37Rv在PM系统中能耐受4% NaCl的高渗透压环境。细菌在感染宿主过程中需应对宿主内环境中变化的高渗环境,因此渗透压调节系统不仅与营养物质跨膜转运相关,还可能与结核分枝杆菌毒力密切相关,对能否有效侵染宿主至关重要[31]。比较H37Rv与H37Ra基因组,有一个膜蛋白基因Rv0037存在突变,而Rv0037为主要易化子超家族(major facilitator superfamily,MFS)蛋白,与调节细胞化学渗透压响应、营养物质跨膜转运相关[14]。因此,推测Rv0037突变可能与H37Ra耐渗透压能力下降有关,可能是结核分枝杆菌的毒力相关基因。
Khatri等[17]利用PM系统对H37Rv的碳源利用能力进行了高通量分析。此后,Lofthouse等[32]设计包括38种不同单一碳源和23种不同单一氮源培养基的实验,对Khatri的研究结果进行了验证。本研究比较了PM系统中H37Rv与H37Ra能高效利用的碳源或氮源种类是否存在差异,结果表明两种菌株在大部分碳源或氮源的利用上没有明显差异;但与H37Rv相比,H37Ra不能有效利用吐温40和丁二酸单甲酯作为碳源(表1)。
表1H37Rv与H37Ra菌株高效利用碳源或氮源的比较
Tab.1Differences in utilization of carbon and nitrogen substrates between H37Rv and H37Ra
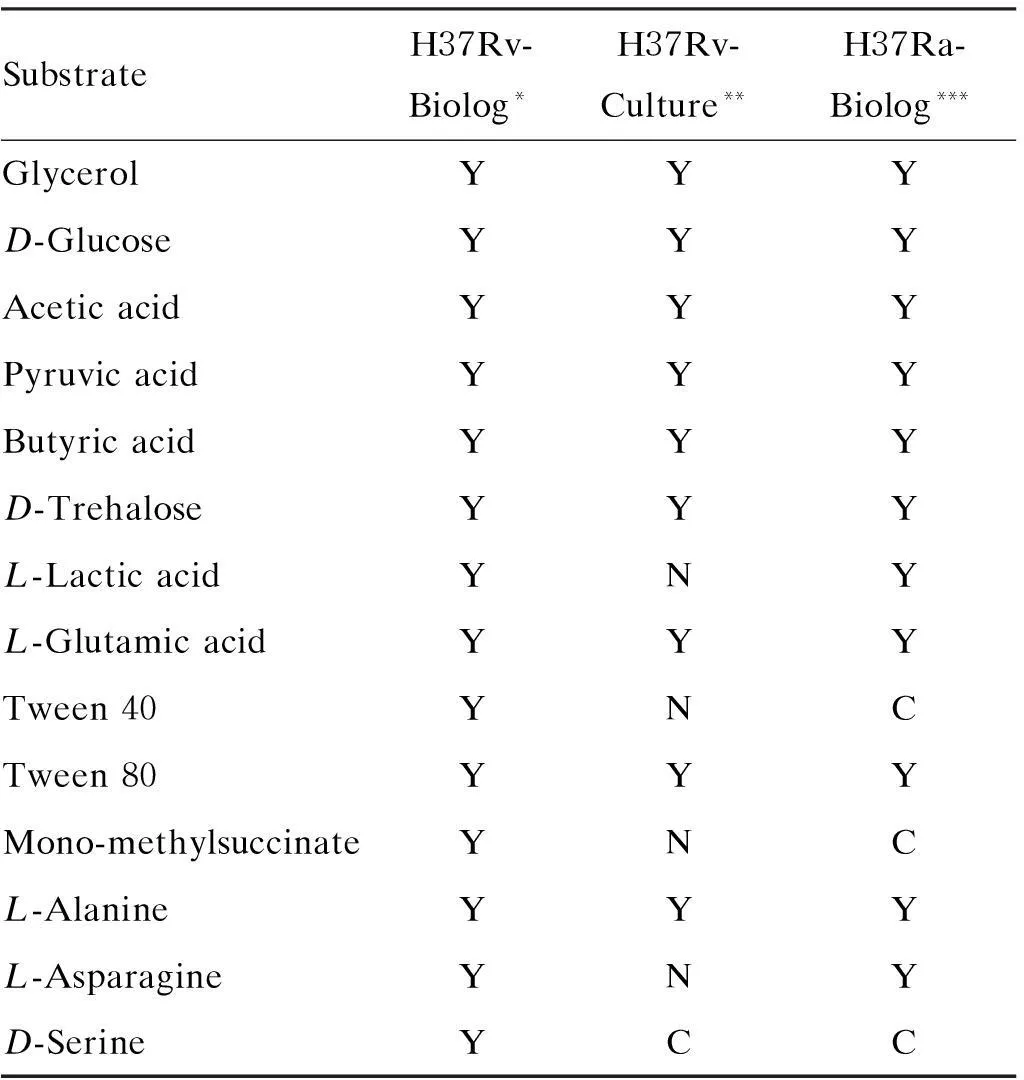
SubstrateH37Rv-Biolog*H37Rv-Culture**H37Ra-Biolog***GlycerolYYYD-GlucoseYYYAceticacidYYYPyruvicacidYYYButyricacidYYYD-TrehaloseYYYL-LacticacidYNYL-GlutamicacidYYYTween40YNCTween80YYYMono-methylsuccinateYNCL-AlanineYYYL-AsparagineYNYD-SerineYCC
*:data from Khatri’research;**:data from Lofthouse’research;***:data from the present research;Y:utilization of carbon/nitrogen source;C:no utilization of carbon/nitrogen source;N:no data.
正常条件下,结核分枝杆菌通过三羧酸循环代谢糖类获取能量;被巨噬细胞吞噬后,其在机体内主要利用宿主的脂类而不是糖类作为碳源和能量来源[33]。因此,部分脂类代谢缺陷可能是H37Ra毒力丧失的原因之一。丁二酸单甲酯和吐温40均为脂类碳源,但结核分枝杆菌中还没有与丁二酸单甲酯和吐温40代谢直接相关基因的报道,仍需深入研究才能确证丁二酸单甲酯和吐温40代谢相关基因或途径与H37Ra毒力缺失的相关性。
PM系统于2001年由Bochner等[15]提出,至今已被用于包括结核分枝杆菌等1 000多种细胞表型的研究,但目前国内外尚无PM系统用于H37Ra表型研究并与H37Rv比较的相关数据。由于结核分枝杆菌易形成菌块,菌悬液不易分散,利用PM系统进行研究往往易形成较高的数据背景,导致实验结果不准确。本研究通过改进菌悬液制备方法,减少了PM系统研究中的数据背景,提高了实验结果的准确率。通过设计碳源利用分析及耐受pH范围的实验,对PM系统的结果进行验证,表明两者相一致。研究表明,PM系统为一种快速、有效的表型分析方法,不仅为分枝杆菌高通量表型筛选研究提供了方便,也为临床分枝杆菌的分型鉴定提供了一种可靠手段。
参考文献
[1]World Health Organization.Global tuberculosis report 2015[R/OL].http://www.who.int/tb/publications/global_report/en.
[2]Cole ST,Brosch R,Parkhill J,Garnier T,Churcher C,Harris D,Gordon SV,Eiglmeier K,Gas S,Barry CE,Tekaia F,Badcock K,Basham D,Brown D,Chillingworth T,Connor R,Davies R,Devlin K,Feltwell T,Gentles S,Hamlin N,Holroyd S,Hornsby T,Jagels K,Krogh A,Mclean J,Moule S,Murphy L,Oliver K,Osborne J,Quail MA,Rajandream MA,Rogers J,Rutter S,Seeger K,Skelton J,Squares R,Squares S,Sulston JE,Taylor K,Whitehead S,Barrell BG.Deciphering the biology of Mycobacterium tuberculosis from the complete genome sequence[J].Nature,1998,393(6685):537-544.
[3]Steenken W.Lysis of tubercle bacilli in vitro[J].Exp Biol Med(Maywood),1935,33(2):253-255.
[4]Middlebrook G,Dubos RJ,Pierce C.Virulence and morphological characteristics of mammalian tubercle bacilli[J].J Exp Med,1947,86(2):175-184.
[5]Dubos RJ,Middlebrook G.Cytochemical reaction of virulent tubercle bacilli[J].Am Rev Tuberc,1948,58(6):698.
[6]Mackaness GB.The growth of tubercle bacilli in monocytes from normal and vaccinated rabbits[J].Am Rev Tuberc,1954,69(4):495-504.[7]Larson CL,Wicht WC.Infection of mice with Mycobacterium tuberculosis,strain H37RA[J].Am Rev Respir Dis,1964,90(5):742-748.
[8]Alsaadi AI,Smith DW.The fate of virulent and attenuated Mycobacteria in Guinea pigs infected by the respiratory route[J].Am Rev Respir Dis,1973,107(6):1041-1046.
[9]Kinger AK,Tyagi JS.Identification and cloning of genes differentially expressed in the virulent strain of Mycobacterium tuberculosis[J].Gene,1993,131(1):113-117.
[10]Rindi L,Lari N,Garzelli C.Search for genes potentially involved in Mycobacterium tuberculosis virulence by mRNA differential display[J].Biochem Biophys Res Commun,1999,258(1):94-101.
[11]Gao Q,Kripke K,Arinc Z,Voskuil M,Small P.Comparative expression studies of a complex phenotype:cord formation in Mycobacterium tuberculosis[J].Tuberculosis(Edinb),2004,84(3-4):188-196.
[12]Cardona PJ,Soto CY,Martín C,Giquel B,Agustí G,Andreu N,Guirado E,Sirakova T,Kolattukudy P,Julián E,Luquin M.Neutral-red reaction is related to virulence and cell wall methyl-branched lipids in Mycobacterium tuberculosis[J].Microbes Infect,2006,8(1):183-190.
[13]Brosch R,Philipp WJ,Stavropoulos E,Colston MJ,Cole ST,Gordon SV.Genomic analysis reveals variation between Mycobacterium tuberculosis H37Rv and the attenuated M.tuberculosis H37Ra strain[J].Infect Immun,1999,67(11):5768-5774.
[14]Zheng H,Lu L,Wang B,Pu S,Zhang X,Zhu G,Shi W,Zhang L,Wang H,Wang S,Zhao G,Zhang Y.Genetic basis of virulence attenuation revealed by comparative genomic analysis of Mycobacterium tuberculosis strain H37Ra versus H37Rv[J].PLoS One,2008,3(6):e2375.
[15]Bochner BR,Gadzinski P,Panomitros E.Phenotype microarrays for high-throughput phenotypic testing and assay of gene function[J].Genome Res,2001,11(7):1246-1255.
[16]Viti C,Bochner BB,Giovannetti L.Florence conference on phenotype microarray analysis of microorganisms—the environment,agriculture,and human health 19-21 March 2008,Florence,Italy[J].Ann Microbiol,2008,58(2):347-349.
[17]Khatri B,Fielder M,Jones G,Newell W,Abu-Oun M,Wheeler PR.High throughput phenotypic analysis of Mycobacterium tuberculosis and Mycobacterium bovis strains’metabolism using Biolog phenotype microarrays[J].PLoS One,2013,8(1):e52673.
[18]Bochner BR.Global phenotypic characterization of bacteria[J].FEMS Microbiol Rev,2009,33(1):191-205.
[19]Gabrielson J,Hart M,Jarelöv A,Kühn I,Mckenzie D,Möllby R.Evaluation of redox indicators and the use of digital scanners and spectrophotometer for quantification of microbial growth in microplates[J].J Microbiol Methods,2002,50(1):63-73.
[20]Zhou L,Lei XH,Bochner BR,Wanner BL.Phenotype microarray analysis of Escherichia coli K-12 mutants with deletions of all two-component systems[J].J Bacteriol,2003,185(16):4956-4972.
[21]de Carvalho LP,Fischer SM,Marrero J,Nathan C,Ehrt S,Rhee KY.Metabolomics of Mycobacterium tuberculosis reveals compartmentalized co-catabolism of carbon substrates[J].Chem Biol,2010,17(10):1122-1131.
[22]Portaels F,Pattyn SR.Growth of mycobacteria in relation to the pH of the medium[J].Ann Microbiol(Paris),1983,133(2):213-221.
[23]Chapman JS,Bernard JS.The tolerances of unclassified mycobacteria.I.Limits of pH tolerance[J].Am Rev Respir Dis,1962,86(4):582-583.
[24]Flannagan RS,Cosío G,Grinstein S.Antimicrobial mechanisms of phagocytes and bacterial evasion strategies[J].Nat Rev Microbiol,2009,7(5):355-366.
[25]Paul S,Laochumroonvorapong P,Kaplan G.Comparable growth of virulent and avirulent Mycobacterium tuberculosis in human macrophages in vitro[J].J Infect Dis,1996,174(1):105-112.
[26]Zhang M,Gong J,Lin Y,Barnes PF.Growth of virulent and avirulent Mycobacterium tuberculosis strains in human macrophages[J].Infect Immun,1998,66(2):794-799.
[27]Hart PD,Armstrong JA.Strain virulence and the lysosomal response in macrophages infected with Mycobacterium tuberculosis[J].Infect Immun,1974,10(4):742-746.
[28]Abramovitch RB,Rohde KH,Hsu FF,Russell DG.aprABC:a Mycobacterium tuberculosis complex-specific locus that modulates pH-driven adaptation to the macrophage phagosome[J].Mol Microbiol,2011,80(3):678-694.
[29]Rohde KH,Abramovitch RB,Russell DG.Mycobacterium tuberculosis invasion of macrophages:linking bacterial gene expression to environmental cues[J].Cell Host Microbe,2007,2(5):352-364.
[30]Gonzalo-Asensio J,Mostowy S,Harders-Westerveen J,Huygen K,Hernández-Pando R,Thole J,Behr M,Gicquel B,Martín C.PhoP:a missing piece in the intricate puzzle of Mycobacterium tuberculosis virulence[J].PLoS One,2008,3(10):e3496.
[31]Sleator RD,Hill C.Bacterial osmoadaptation:the role of osmolytes in bacterial stress and virulence[J].FEMS Microbiol Rev,2002,26(1):49-71.
[32]Lofthouse EK,Wheeler PR,Beste DJ,Khatri BL,Wu H,Mendum TA,Kierzek AM,Mcfadden J.Systems-based approaches to probing metabolic variation within the Mycobacterium tuberculosis complex[J].PLoS One,2013,8(9):e75913.
[33]Fisher MA,Plikaytis BB,Shinnick TM.Microarray analysis of the Mycobacterium tuberculosis transcriptional response to the acidic conditions found in phagosomes[J].J Bacteriol,2002,184(14):4025-4032.
High throughput analysis of phenotypic differences between H37Ra and H37Rv using Biolog Phenotype MicroArray
WANG Yihong,GUO Qinglong,ZHOU Fengzhu,GOU Zongchao,WANG Honghai,ZHANG Xuelian
State Key Laboratory of Genetic Engineering,School of Life Sciences,Fudan University,Shanghai 200433,China
Abstract:Tuberculosis,caused by Mycobacterium(M.)H37Rv,is a human disease of major importance worldwide.The avirulent M. H37Ra which is used commonly in the laboratory was derived from H37Rv in 1935,but the mechanism of its virulence attenuation remains unclear.In order to explore its possible mechanism of virulence attenuation,screening for different phenotypes is important.To make a rapid and high throughput phenotypic analysis between H37Ra and H37Rv,as well as catch the different phenotypes related to virulence attenuation,the Biolog Phenotype MicroArray analysis was conducted.In this research,the phenotypes of H37Ra were determined and compared with its virulent counterpart H37Rv.Of 765 substrates surveyed,H37Ra phenotypes were highly similar to that of H37Rv,but there were several differences in carbon utilization,osmosis and pH arrays.These arrays suggested that H37Ra could not utilize mono-methyl succinate and Tween 40 as carbon source in contrast to H37Rv.More interestingly,the differences between H37Ra and H37Rv in the tolerance abilities of hyperosmosis and acid were found,and H37Ra could not tolerate circumstance of pH 5.5 or 3% NaCl,but H37Rv could.These metabolic differences may have a disparate impact on the survival of M. in the hosts and be closely related to the virulence attenuation of H37Ra.
Key words:Mycobacterium;Phenotype;Virulence
基金项目:国家自然科学基金(81261120558),上海市科技支撑项目(13431900203、15431900200)
通信作者:张雪莲
Corresponding author.ZHANG Xuelian,E-mail:xuelianzhang@fudan.edu.cn
(收稿日期:2016-02-24)

