基于NRPS基因筛选与鉴定葡萄附生真菌
吕 曼,杨 柯,王 傲,史雅凝,辛志宏
(南京农业大学食品科技学院,江苏南京 210095)
基于NRPS基因筛选与鉴定葡萄附生真菌
吕曼,杨柯,王傲,史雅凝,辛志宏*
(南京农业大学食品科技学院,江苏南京 210095)
以非核糖体多肽合成酶(nonribosomal peptide synthetase,NRPS)基因为筛选靶点,从葡萄中筛选出含有NRPS基因的附生真菌,以期发现具有抗菌作用的环肽类化合物。采用平板划线技术、斜面分离纯化技术分离葡萄附生真菌,从中筛选出一株含有NRPS基因的附生真菌(编号PTLM-1)。在此基础上,构建系统发育进化树,结合形态学方法,将该菌株鉴定为腐皮镰刀菌Fusariumsolani。经过NRPS系统发育进化树分析,发现该菌株有产生抗菌环肽类化合物——环孢菌素的潜力。通过对该菌的发酵产物进行电喷雾质谱分析,进一步证实了该预测结果。该研究为定向筛选产生环肽类化合物的菌株提供了新的研究方法与理论依据。
葡萄,附生真菌,18S rDNA,ITS rDNA,分离鉴定,非核糖体多肽合成酶
食药两用植物能够产生许多具有重要生理活性的物质,在医药、食品和农业生产等领域具有重要的应用价值。然而,有些植物生长周期长且种类稀少,难以提供足够的生产原料,而且大量采伐会破坏生态环境。幸运的是,近几十年的微生物与生态学研究表明,几乎每一种植物都共附生有一种或多种微生物,这些微生物不仅能产生结构新颖的活性化合物,而且有些微生物还能产生与其宿主相同或相似的物质,尤其是食药两用植物[1],如葡萄,作为一种广泛种植、以食用为主、食药两用的作物,蕴藏着丰富的共附生微生物资源,可通过发酵技术大批量培养,经过分离纯化获得目标产物,这种策略既可保护生态环境,又能为可持续开发天然生物活性物质提供一条新途径,但目前对其附生真菌的研究主要集中在种类鉴定与葡萄病害防治上。所以,开展葡萄共附生真菌的筛选及其次级代谢产物的研究具有重要意义。
在筛选有价值的共附生微生物过程中,有效的筛选方法是决定能否发现新型活性化合物的关键[2]。通常采用的筛选方法是基于活性筛选和化学筛选或者二者的组合[3],这些方法在微生物次级代谢产物研究的过程中,筛选到许多盛产活性物质的菌株,但是随着研究的深入,这些筛选方法的固有缺陷逐渐显现出,一些已知菌株和已知化合物被不断重复发现,大大降低了发现新化合物的概率,造成了巨大的时间和资源浪费,已经成为植物真菌研究过程中最主要的制约因素[4-5]。
随着现代测序技术的发展,基因组测序、功能基因定位及其功能研究等相关技术逐渐成熟,一些微生物合成天然产物的生物合成途径得到解密,为通过功能基因筛选来预测化合物的结构类型提供了理论基础[6]。基于非核糖体多肽合成酶(NRPS)功能基因定向筛选,目标直接指向合成多肽类化合物的目标基因,成为一种发现新型天然次级代谢产物的重要策略。许多新发现的抗菌、抗肿瘤和抗癌化合物都是由NRPS途径合成的[7-8]。目前,通过依据NRPS基因筛选目标菌株,大多数集中在细菌方面,而有关真菌NRPS基因的筛选罕见报道[9-10]。
由于微生物分离鉴定工作相对复杂且周期长,因此本文结合生产实际选择含水量较高、易腐败且种植范围广的巨峰葡萄为筛选对象,以NRPS功能基因为靶点,采用平板划线分离、斜面分离纯化技术筛选葡萄附生真菌,在此基础上,提取真菌DNA,PCR扩增18S rDNA、ITS rDNA、LSU rDNA和EF-1α目标序列,在NCBI数据库中进行BLAST搜索,下载与目的菌株有较高同源性的序列,构建系统发育进化树,并结合传统的形态学方法,确定目的菌株的种属地位,以期定向筛选到具有NRPS功能基因的附生真菌。
1 材料与方法
1.1材料与仪器
巨峰葡萄购自江苏省南京市苏果超市;Omega真菌基因组试剂盒(Fungal DNA Kit 50)及引物上海捷瑞生物工程有限公司;DR001AM PCR试剂、Ver.3.0 D823A琼脂糖凝胶DNA提取试剂盒日本TaKaRa公司;pMD19-T载体、solutionⅠ、2×Taq Master Mix、大肠杆菌DH5α感受态细胞南京诺唯赞生物科技有限公司;其他分析纯试剂南京寿德实验器材有限公司;PDA固体培养基土豆200 g/L、葡萄糖20 g/L、琼脂20 g/L、食盐30 g/L,自来水配制;LB液体培养基胰蛋白胨10 g/L、酵母粉5 g/L、NaCl 10 g/L,pH7.2;LB固体培养基胰蛋白胨10 g/L、酵母粉5 g/L、NaCl 10 g/L,琼脂5 g/L,pH7.2;真菌发酵液体培养基土豆200 g/L,麦芽糖20 g/L,甘露醇20 g/L,葡萄糖10 g/L,味精5 g/L,蛋白胨5 g/L,酵母膏3 g/L,pH6.0。
DYCP-31DN电泳仪北京市六一仪器厂;JS-380C全自动数码凝胶成像分析仪上海市培清科技有限公司;TP600型梯度PCR仪日本TaKaRa公司;Microfuge 22R台式微量冷冻离心机美国Beckman公司;Mariner API-TOF型质谱仪美国应用生物系统公司。
1.2实验方法
1.2.1菌株的分离培养将巨峰葡萄于无菌操作台中破碎,梯度稀释后接种于PDA平板培养基上,于28 ℃培养2~3 d。待培养基中长出菌丝后,挑取新鲜菌丝到另一PDA平板培养基上进行划线分离,得到单菌落,并在PDA斜面试管中划线接种,4 ℃保存。
1.2.2菌株的形态学观察
1.2.2.1菌落形态特征从斜面培养基中挑取新鲜菌丝接种至另一PDA平板培养基上,于28 ℃恒温培养4~5 d,观察菌落的大小、颜色、质地等形态特征并记录。
1.2.2.2显微形态特征挑取菌落边缘菌丝,制作水浸片,然后在倒置显微镜下观察其菌丝体、分生孢子、孢子梗等。
1.2.3基因组DNA的提取从斜面中挑取新鲜菌丝接种到PDA平板培养基上,于28 ℃恒温培养箱中培养3~4 d。真菌基因组DNA的提取采用Omega真菌基因组试剂盒(Fungal DNA Kit 50)进行。将1%琼脂糖煮沸至澄清透明,加几滴EB并搅拌均匀进行染色,待30 min冷却成型后即可,将所提取的真菌基因组DNA在1%琼脂糖凝胶中电泳检测(120 V,30 min)。4 ℃保存备用,或于-20 ℃中长期保存。
1.2.4NRPS功能基因片段的扩增与基因克隆分别选用AUG003/AUG007、AUG003/AUG006、AUG005/AUG007 3对简并引物对其NRPS基因序列进行扩增,PCR反应条件为94 ℃预变性2 min,94 ℃变性30 s,55 ℃退火30 s,72 ℃延伸3 min,共35个循环,最后72 ℃延伸10 min[11]。PCR扩增反应采用25 μL的反应体系,包括ddH2O 9.5 μL、2×Taq Master Mix 12.5 μL、Primer-F(10 μmol/L)1 μL、Primer-R(10 μmol/L)1 μL、DNA 1 μL。将1%琼脂糖煮沸至澄清透明,加几滴EB并搅拌均匀进行染色,待30 min冷却成型后将上述PCR扩增产物,在1%琼脂糖凝胶中电泳检测(120 V,30 min),4 ℃保存。
1.2.518S rDNA基因片段的PCR扩增选用真核生物18S rDNA的通用扩增引物:NS1(5′-GTAG TCATATGCTTGTCTC-3′)/NS8(5′-TCCGCAGGTTCA CCTACGGA-3′)对18S rDNA片段进行扩增,PCR反应条件为94 ℃预变性4 min,94 ℃变性45 s,50 ℃退火1 min,72 ℃延伸1 min 45 s,共35个循环,最后72 ℃延伸10 min[12]。PCR扩增反应采用25 μL的反应体系,包括ddH2O 9.5 μL、2×Taq Master Mix 12.5 μL、Primer-F(10 μmol/L)1 μL、Primer-R(10 μmol/L)1 μL、DNA 1 μL。将1%琼脂糖煮沸至澄清透明,加几滴EB并搅拌均匀进行染色,待30 min冷却成型后将上述PCR扩增产物在1%琼脂糖凝胶中电泳检测(120 V,30 min),4 ℃保存。
1.2.6ITS rDNA基因片段的PCR扩增选用真核生物ITS rDNA的通用扩增引物:ITS1(5′-TCCGTA GGTGAACCTGCGG-3′)/ITS4(5′-TCCTCCGCTTA TTGATAT GC-3′)对ITS rDNA片段进行扩增,PCR反应条件为94 ℃预变性2 min,94 ℃变性30 s,59 ℃退火30 s,72 ℃延伸1 min 30 s,共35个循环,最后72 ℃延伸7 min[13]。PCR扩增反应采用25 μL的反应体系,包括ddH2O 9.5 μL、2×Taq Master Mix 12.5 μL、Primer-F(10 μmol/L)1 μL、Primer-R(10 μmol/L)1 μL、DNA 1 μL。将1%琼脂糖煮沸至澄清透明,加几滴EB并搅拌均匀进行染色,待30 min冷却成型后将上述PCR扩增产物在1%琼脂糖凝胶中电泳检测(120 V,30 min),4 ℃保存。
1.2.7LSU rDNA基因片段的PCR扩增选用真核生物LSU rDNA的通用扩增引物:NL1(5′-GCATATCAATAAGCGGAGGAAAAG-3′)/LR3(5′-GGTCCGTGTTTCAAGAC-3′)对LSU rDNA片段进行扩增,PCR反应条件为94 ℃预变性5 min,94 ℃变性1 min,53 ℃退火1 min,72 ℃延伸2 min,共35个循环,最后72 ℃延伸7 min[14]。PCR扩增反应采用25 μL的反应体系,包括ddH2O 9.5 μL、2×Taq Master Mix 12.5 μL、Primer-F(10 μmol/L)1 μL、Primer-R(10 μmol/L)1 μL、DNA 1 μL。将1%琼脂糖煮沸至澄清透明,加几滴EB并搅拌均匀进行染色,待30 min冷却成型后将上述PCR扩增产物在1%琼脂糖凝胶中电泳检测(120 V,30 min),4 ℃保存。
1.2.8EF-1α片段的PCR扩增选用真核生物EF-1α的扩增引物:EF2T(5′-GGAAGTACCAGTGATC ATGTT-3′)/EF3(5′-GTAAGGAGGASAAGACTCACC-3′)对EF-1αrDNA片段进行扩增,PCR反应条件为94 ℃预变性5 min,94 ℃变性30 s,53 ℃退火30 s,72 ℃延伸1 min,共35个循环,最后72 ℃延伸5 min[15]。PCR扩增反应采用25 μL的反应体系,包括ddH2O 9.5 μL、2×Taq Master Mix 12.5 μL、Primer-F(10 μmol/L)1 μL、Primer-R(10 μmol/L)1 μL、DNA 1 μL。将1%琼脂糖煮沸至澄清透明,加几滴EB并搅拌均匀进行染色,待30 min冷却成型后将上述PCR扩增产物在1%琼脂糖凝胶中电泳检测(120 V,30 min),4 ℃保存。
1.2.9PCR产物的回收及克隆采用琼脂糖凝胶DNA提取试剂盒回收上述PCR产物,将新鲜制备的或-70 ℃保存的100 μL大肠杆菌DH5α感受态细胞先置于冰上5 min后轻甩使细胞悬浮。再将纯化的PCR产物与pMD19-T载体连接,然后转化至感受态细胞中,冰中放置30 min,取出后42 ℃热激90 s,冰中放置5~10 min后加入890 μL LB液体培养基,37 ℃、90 r/min振荡培养1 h。将菌液均匀涂布于含AMP(100 μg/mL)的LB培养基平板上,正放15 min后倒置过夜培养,挑取白色单菌落接到含有AMP的LB液体培养基中(1 mL LB加1 μL AMP),37 ℃,120 r/min振荡培养10~12 h。吸取1 μL培养过的菌液进行PCR,所用引物为M13-RV和M13-47,电泳检测是否含有目的片段。
PCR采用25 μL的反应体系,包括ddH2O 9.5 μL、2×Taq Master Mix 12.5 μL、Primer-F(10 μmol/L)1 μL、Primer-R(10 μmol/L)1 μL、DNA 1 μL。将得到的PCR扩增产物用1%的琼脂糖凝胶电泳检测后,4 ℃保存。PCR反应条件为94 ℃预变性5 min,94 ℃变性30 s,56 ℃退火30 s,72 ℃延伸1 min,共35个循环,最后72 ℃延伸10 min。
1.2.10目的DNA序列的测序和系统发育学分析将NRPS、18S rDNA、ITS rDNA、LSU rDNA和EF-1α的单克隆PCR产物交由上海美吉生物有限公司进行测序。获得拼接序列后,将其转化为蛋白序列后进行同源性搜索,下载同源较高的蛋白序列;将18S rDNA、ITS rDNA、LSU rDNA和EF-1α序列在NCBI中进行BLAST比对,从中下载与该序列相似性较高的核酸序列。使用MEGA 6.0软件,进行多重序列匹配排列和聚类分析,采用邻接法(neighbor joining method)构建系统发育进化树,并通过自举分析(bootstrap)进行置信度检测,自举数据集为1000次。
1.2.11菌株PTLM-1的发酵与质谱分析将分离纯化得到的菌株接种在真菌发酵液体培养基中,发酵培养7 d,用丙酮提取发酵产物,过滤后直接进行进样分析。质谱条件:离子源ESI负离子模式,毛细管电压4000 V,雾化器压力25 psig,干燥气9 L/min,干燥气温度300 ℃,扫描范围50~1100 m/z。
2 结果与分析
2.1菌株形态学鉴定
2.1.1菌落形态特征由图1可知,菌株PTLM-1在PDA培养基上28 ℃培养4~5 d后,观察可见其气生菌丝为白色至浅灰色,形如羊绒。
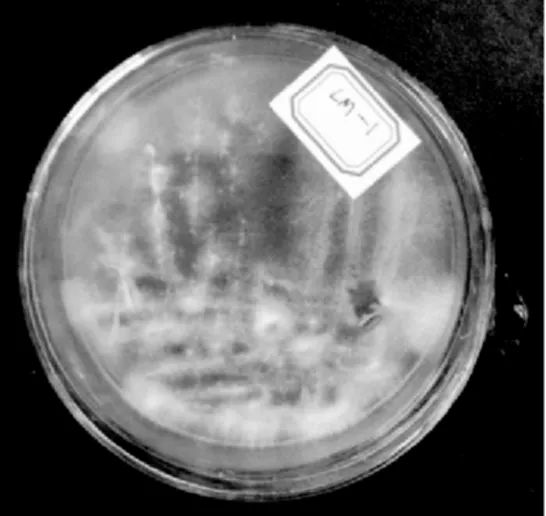
图1 菌株PTLM-1的菌落形态图Fig.1 The morphological characters of PTLM-1
2.1.2显微形态特征由图2可知,菌株PTLM-1的显微形态:分生孢子以孢尖或孢体附于菌丝上,形态各异。其中大型分生孢子分布较稀疏;小型分生孢子则呈串排列。大型分生孢子两端较尖,呈镰刀形或纺锤形,孢体稍弯曲;小分生孢子呈卵形、肾形、梨形或纺锤形等,常平铺聚集与菌丝连接在一起。

图2 菌株PTLM-1的显微形态图Fig.2 The microscopic morphological characters of PTLM-1注:A、B为菌株PTLM-1的倒置显微镜图,放大倍数为100×;C、D为菌株PTLM-1的扫描电镜图,其中C图放大倍数为8000×,D图放大倍数为2000×。
2.2NRPS片段扩增与系统发育学分析
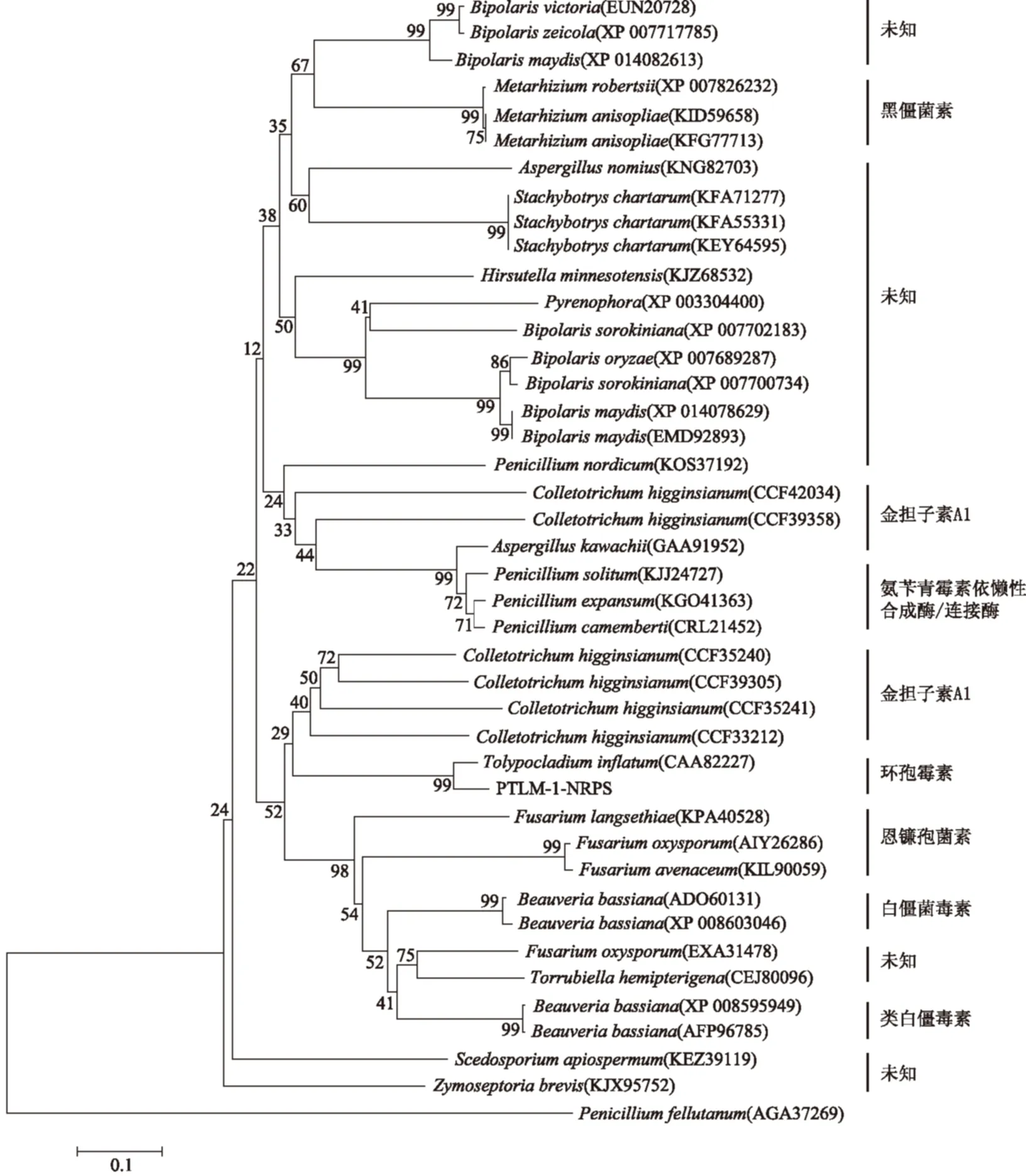
图4 基于PTLM-1 NRPS基因AUG系列引物构建的系统发育进化树Fig.4 Neighbor-Joining phylogenetic tree based on partial NRPS rDNA sequences
经PCR扩增后发现仅引物AUG003/AUG006获得菌株PTLM-1的NRPS 750 bp的目的条带,如图3所示。测序后将核酸序列在NCBI上转化为蛋白序列并进行同源性搜索,从中下载同源性较高的蛋白序列,将PTLM-1对应的蛋白序列与下载的蛋白序列经MEGA 6.0软件进行比对后,以邻接法(Neighbor-Joining)构建基于NRPS功能基因的系统发育进化树,如图4所示。不同种的绿僵菌菌株(Metarhiziumsp.)聚为一枝,都能产生腐败菌素(destruxin);而不同属的炭疽菌(Colletotrichumsp.)和青霉菌(Penicilliumsp.)都能产生金担子素A1(aureobasidin A1)。这说明同属真菌的不同种菌株或不同属真菌都能够产生相同类型的化合物。如菌株PTLM-1(登录号为KX147648)与Tolypocladium inflatum(CAA82227)聚为一枝,自展值为100,表现出很近的亲缘关系。说明菌株PTLM-1可能产生抗菌环肽类化合物——环孢菌素(cyclosporine)。

图3 LM-1 NRPS基因片段扩增图谱Fig.3 PCR amplified products of 18S rDNA and ITS注:A:NRPS基因PCR扩增结果;M:DNA分子质量标准。
2.318S rDNA和ITS rDNA扩增与系统发育学分析

图5 基于菌株PTLM-1 18S rDNA序列构建的系统发育Fig.5 Neighbor-Joining phylogenetic tree based on partial 18S rDNA sequence
经PCR扩增分别获得菌株PTLM-1的18S rDNA和ITS1-5.8S-ITS4 rDNA区域的序列,其中PTLM-1的18S rDNA序列全长为1700 bp,PTLM-1的rDNAITS1-5.8S-ITS4序列全长为500 bp左右。
将菌株PTLM-18S序列在NCBI中进行BLAST比对(GenBank登录号KX147646),发现该序列与

图6 基于菌株PTLM-1 ITS rDNA序列构建的系统发育进化树Fig.6 Neighbor-Joining phylogenetic tree based on partial ITS rDNA sequences

图7 基于菌株PTLM-1 LSU rDNA序列构建的系统发育进化树Fig.7 Neighbor-Joining phylogenetic tree based on partial LSU rDNA sequences
Fusariumsp.的18S rDNA序列同源性最高,相似性达100%。选取同源性较高菌株的18S rDNA基因序列,使用MEGA 6.0软件构建系统发育进化树(图5)。从系统发育进化树中可以看出,菌株PTLM-1与F.solani(EF397944)和F.solani(AB473810)聚为一枝,表现出相当近的亲缘关系,因此可将其初步鉴定为Fusariumsp.。再将该菌株rDNA ITS1-5.8S-ITS4序列与GenBank数据库中收录的真菌rDNA的ITS1-5.8S-ITS4序列进行BLAST比对(GenBank登录号KX147647)。经过比对发现菌株PTLM-1的ITS1-5.8S-ITS4 rDNA序列与F.solani的ITS基因序列同源性最高,相似性可达99%。从系统发育进化树(图6)中可以看出,菌株PTLM-1与F.solani(FJ345352)聚为一枝,自展值为70,表现出较近的亲缘关系。
2.4LSU rDNA和EF-1α的系统发育学分析
将该菌株LSU rDNA序列与GenBank数据库中收录的真菌LSU rDNA序列进行BLAST比对(GenBank登录号KX147649)。经过比对发现菌株PTLM-1的LSU rDNA序列与F.solani的LSU rDNA序列同源性最高,相似性达99%。从系统发育树(图7)中可以看出,菌株PTLM-1与F.solani(KF938479)和F.solani(EU214559)聚为一枝,自展值为94,表现出相当近的亲缘关系。再将该菌株EF-1α序列与GenBank数据库中收录的真菌EF-1α序列进行BLAST比对(GenBank登录号KX147650)。经过比对发现菌株PTLM-1的EF-1α序列与F.solani的EF-1αrDNA序列相似性可达99%。
综合菌落显微形态特征、NRPS、18S rDNA、rDNA ITS1-5.8S-ITS4、LSUrDNA及EF-1α序列分析结果,且与真菌鉴定手册标准菌株对照后,将该菌株PTLM-1鉴定为半知菌类(Fungi Imperfecti)、丛梗孢目(Melanconiales)、瘤座孢科(Tuberculariaceae)、镰刀菌属(Fusariumsp.)、腐皮镰刀菌(F.solani)[16]。
2.5菌株PTLM-1的质谱分析
将菌株PTLM-1发酵7 d后,用丙酮提取发酵产物浓缩蒸干,加适量水溶解后直接进行进样分析。结果如图8所示,菌株PTLM-1的粗提物在m/z 1020.8、1034.8、1035.8、1042.8处产生碎片离子峰,为环孢菌素类化合物的特征分子离子质量,因此发酵产物中确实可能存在环孢菌素类化合物。另外,张梅等[17]从腐皮镰刀菌固体发酵培养物中也分离得到1个次级代谢产物,通过核磁共振、质谱等波谱技术分析得知此化合物为酚类化合物,并且具有抑制酪氨酸酶活性,阻止黑色素的形成,为化妆品行业的发展奠定了理论基础[18]。
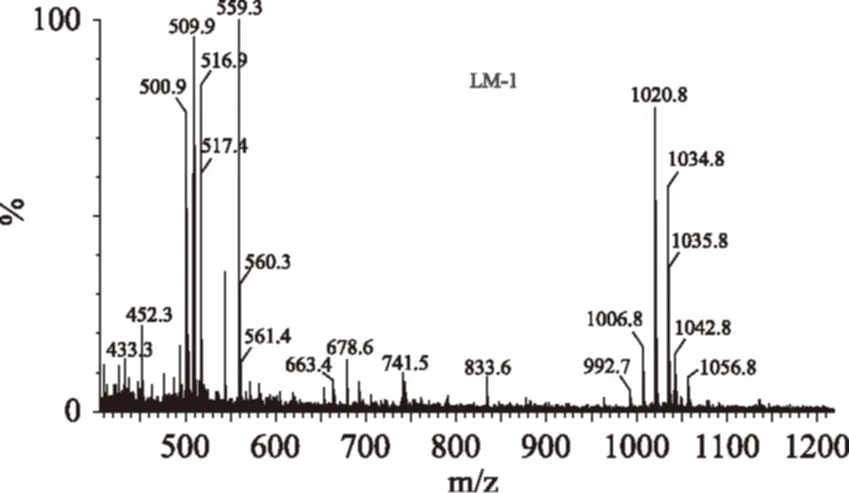
图8 菌株PTLM-1 发酵产物质谱图Fig.8 Mass spectrum diagram for PTLM-1
3 结论与讨论
传统的真菌分类学鉴定主要依据形态学鉴定和生理生化鉴定,然而真菌种类的多样性及环境的不稳定性等不可避免的因素大大限制了这些方法的发展。随着分子生物学的快速发展,通过提取真菌基因组DNA,并对其18S、ITS、LSU及EF-1αrDNA片段进行PCR扩增,进而同源性分析并构建系统发育进化树来确定菌株的种属地位,已成现代真菌鉴定最为有效的方法。
共附生微生物能否产生结构新颖的生物活性物质,基于其是否含有编码相应次级代谢产物合成的基因簇。2005年,Miao课题组通过研究StreptomycesroseosporusNRRL 11379的NRPS合成途径,首次从中克隆出达托霉素合成基因簇,这为从基因到新化合物的预测奠定了理论基础,并且也成为现代天然产物研究与开发的有效手段[19]。本研究基于NRPS基因筛选真菌菌株,目标直接指向新化合物的合成途径,减小已知化合物的发现概率,大大提高了工作效率。
本研究以NRPS功能基因为靶点,从含水量高且种植范围广的巨峰葡萄中分离得到菌株PTLM-1。结合系统发育学分析与传统的形态学观察,将该菌鉴定为腐皮镰刀菌F.solani。根据NRPS系统发育学分析,猜测菌株PTLM-1可能产生环孢菌素类化合物,经发酵后通过质谱分析验证,发现腐皮镰刀菌PTLM-1确实能产生环孢菌素。环孢菌素作为一种微生物次级代谢产物,除了具有抗菌、抗肿瘤等生物活性外,临床上还可将其应用于抑制器官移植时的排斥反应。除此之外,腐皮镰刀菌还能产生其他结构新颖、生理活性独特的化合物,如酚类化合物、大黄素、脑苷脂等。大黄素除了具有抗菌抗炎、抗肿瘤等药理作用外,还可抗氧化清除氧自由基[20];而脑苷脂除了具有抑菌活性、保肝活性及抗溃疡等生物活性外,还对作用于细胞壁的抗生素有增效作用。可见,腐皮镰刀菌的次级代谢产物不仅结构多样,而且生物活性极其显著,具有明显研究价值。
多孔木霉(T.inflatum)产生的环孢菌素H和其它二十多个同系物,是目前为止国内外关于环孢菌素产生菌少有的报道[21]。福建省微生物研究所[22-25]从筛选到的腐皮镰刀菌中也已分离出十余种环孢菌素,并证明它们与多孔木霉产生的环孢菌素组分相同,而且外源氨基酸对多孔木霉和腐皮镰刀菌生物合成环孢菌素的影响不同,这为进一步阐明环孢菌素的生物合成途径,比较不同真菌来源环孢菌素合成酶的特性,指导环孢菌素的工业化生产有重要的意义,但国内外如今对产环孢菌素真菌腐皮镰刀菌遗传特性研究较少,并且在发酵过程中环孢菌素可能会受到菌种种类、发酵条件等多方面影响以至于产量并不稳定,倘若对腐皮镰刀菌进行基因改造,环孢菌素的产量将会大大提高。因此,研究腐皮镰刀菌的NRPS基因片段,为进一步选育高产环孢菌素菌株奠定了良好的基础。
[1]王伟,朱平,程克棣. 药用植物基因组及EST研究[J]. 中国生物工程杂志,2004,5(1):1-5.
[2]许静,徐俊. 海洋共附生微生物天然产物生物合成基因研究进展[J]. 微生物学报,2008,219(7):975-979.
[3]朱鹏.珍珠膜海绵共附生微生物PKS与NRPS的筛选与模块结构研究[D]. 杭州:浙江大学,2008.
[4]Moore B S. Biosynthesis of marine natural products:microorganisms(Part A)[J]. Nat Prod Rep,2005,22(5):580-593.
[5]文艳苹.基于PKS、NRPS基因的抗生素paenimacrolidin和嗜铁素paenibactin研究[D]. 杭州:浙江大学,2011.
[6]朱孟沼,崔晓龙,李铭刚,等. 发现微生物药物的新途径:从基因到新化合物[J]. 天然产物研究与开发,2006,12(5):854-857.
[7]张薇.海绵共附生活性菌PKS和NRPS基因的筛选及多样性研究[D]. 上海:上海交通大学,2008.
[8]符浩.基于放线菌基因组Ⅰ型聚酮合酶和非核糖体肽合成酶保守序列筛选天然活性化合物的研究[D]. 上海:华东理工大学,2012.
[9]Chang Z,Flatt P,Gerwick W H,et al. The barbamide biosynthetic gene cluster:a novel marine cyanobacterial system of mixed polyketide synthase(PKS)-non-ribosomal peptide synthetase(NRPS)origin involving an unusual trichloroleucyl starter unit[J]. Gene,2002,296(1-2):235-247.
[10]肖吉,张光涛,朱义广,等. 海洋微生物次级代谢产物生物合成的研究进展[J]. 中国抗生素杂志,2012,34(4):241-253.
[11]Marahiel M A,Stachelhaus T,Mootz H D. Modular Peptide Synthetases Involved in Nonribosomal Peptide Synthesis[J]. Chem Rev,1997,97(7):2651-2674.
[12]Liang X H,Cai Y J,Liao X R,et al. Isolation and identification of a new hypocrellin A-producing strainShiraiasp. SUPER-H168[J]. Microbiol Res,2009,164(1):9-17.
[13]燕勇,李卫平,高雯洁,等. rDNA-ITS序列分析在真菌鉴定中的应用[J]. 中国卫生检验杂志,2008,56(10):1958-1961.
[14]Kurtzman C P,Robnett C J. Identification and phylogeny of ascomycetous yeasts from analysis of nuclear large subunit(26S)ribosomal DNA partial sequences[J]. Antonie Van Leeuwenhoek,1998,73(4):331-371.
[15]O’Donnell K,Cigelnik E. Two divergent intragenomic rDNA ITS2 types within a monophyletic lineage of the fungus Fusarium are nonorthologous[J]. Mol Phylogenet Evol,1997,7(1):103-116.
[16]魏景超. 真菌分类鉴定手册[M].上海:上海科技出版社,1979:487-533.
[17]张梅,徐良雄,薛璟花,等. 一株茄病镰刀菌的代谢产物研究[J]. 热带亚热带植物学报,2012,36(6):585-590.
[18]Fukai S,Tanimoto S,Maeda A,et al. Pharmacological activity of compounds extracted from persimmon peel(Diospyros kaki THUNB.)[J]. J Oleo Sci,2009,58(4):213-219.[19]刘怡君,潘沐,王明洋,等. 海芦笋内生真菌基于PKS Ⅰ型功能基因的分离与鉴定[J]. 食品科学,2014,72(11):115-119.
[20]于志伟,杨欣,曾宪军,等. 腐皮镰刀菌R13发酵产大黄素的初步研究[J]. 四川大学学报(自然科学版),2015,52(5):1135-1140.
[21]方金瑞,唐信东,任林英,等. 氨基酸对环孢菌素A生物合成的影响[J]. 中国抗生素杂志,1990,12(2):140-141.
[22]方剑英.环孢菌素A衍生物的合成及其生物学活性研究[D]. 福州:福建医科大学,2008.
[23]温耀明.环孢菌素H衍生物的合成及其生物学活性研究[D]. 福州:福建医科大学,2007.
[24]陈振伟.茄病镰刀菌产生的环孢菌素L及其未知组分的研究[D].福州:福建医科大学,2007.
[25]陈秀明.茄病镰刀菌产生的环孢菌素小组分的研究[D]. 福州:福建医科大学,2008.
Isolation and identification based on NRPS functional gene of a fungal epiphytic strain from grapes
LV Man,YANG Ke,WANG Ao,SHI Ya-ning,XIN Zhi-hong*
(College of Food Science and Technology,Nanjing Agricultural University,Nanjing 210095,China)
A fungal epiphytic with nonribosomal peptide synthetase(NRPS)gene was isolated from a root rot of grapes basing on nonribosomal peptide synthetase gene target-screening in order to find a cyclic peptide with antimicrobial activity. The fungal epiphytic with nonribosomal peptide synthetase(NRPS)gene named as PTLM-1 was isolated from grapes by streak plate and slant culture methods. On this basis,constructed a phylogenetic tree,then the species of the strain PTLM-1 was identified asF.solanicombined with morphological observation. It was found that the strain had the potential to produce a cyclic peptide named cyclosporine after NRPS sequence analysising,which was further confirmed by the electrospray ionizsation mass spectrum(ESI-MS)analysis. This study provided new research methods and theoretical basis for target-screening strains which produced cyclic peptide.
grapes;a epiphytic fungi;18S rDNA;ITS rDNA;identification;NRPS
2016-02-22
吕曼(1991-),女,硕士研究生,研究方向:食品营养与化学,E-mail:2014108057@njau.edu.cn。
辛志宏(1974-),男,博士,教授,研究方向:食品营养与化学,E-mail:xzhfood@njau.edu.cn。
农业部农产品质量安全风险评估项目(GJFP2015012)。
TS255.1
A
1002-0306(2016)18-0229-08
10.13386/j.issn1002-0306.2016.18.035

