基于名优谷子品种晋谷21全基因组重测序的分子标记开发
赵庆英 张瑞娟 王瑞良 高建华,3 韩渊怀 杨致荣1,,* 王兴春,,*
基于名优谷子品种晋谷21全基因组重测序的分子标记开发
赵庆英1,**张瑞娟2,**王瑞良2高建华2,3韩渊怀3,4杨致荣1,3,4,*王兴春2,3,4,*
1山西农业大学文理学院, 山西太谷 030801;2山西农业大学生命科学学院, 山西太谷 030801;3山西农业大学农业生物工程研究所, 山西太谷 030801;4杂粮种质资源发掘与遗传改良山西省重点实验室, 山西太谷 030801
小米因其营养丰富日益受到重视, 而小米的品质是民众选择小米时最为关注的指标。晋谷21米质优异, 但由于缺少基因组信息, 严重阻碍了其优异米质形成机制的研究。本研究利用高通量测序技术, 对晋谷21全基因组进行重测序, 获得了14.95 Gb高质量测序数据。进一步将其与豫谷1号参考基因组比较, 发掘了169 037个InDel位点和1 167 555个SNP位点, 其中长度在13~50 bp之间适于琼脂糖凝胶电泳检测的InDel位点为14 578个。选择其中1个SNP位点和68个InDel位点验证, 表明利用二代测序技术开发的InDel和SNP标记真实可靠。基于名优谷子晋谷21重测序数据开发的InDel和SNP分子标记具有通用性, 可用于其他谷子、狗尾草和谷莠子等种质资源的相关研究。同时, 开发了一个晋谷21特异的InDel标记2G5501976, 利用该标记即可快速鉴定待测材料是否为晋谷21及其衍生品种。本研究初步揭示了晋谷21的基因组特征, 不仅为深入解析其优异米质形成的分子机制奠定了基础, 而且为相关分子标记辅助育种、遗传分析和基因克隆提供了分子标记资源。
谷子; 晋谷21; InDel; SNP; 分子标记; 基因组重测序
谷子(L.)起源于中国, 是我国过去几千年来的主栽作物和中华民族的哺育作物。脱壳后的谷子称为小米, 富含蛋白质、脂肪、铁、类胡萝卜素、纤维素、维生素等[1-3]。近年来, 随着人们生活水平的不断提高及食品的多样化和营养化, 优质小米倍受青睐。然而, 长期以来谷子品质相关的研究进展缓慢, 且主要集中在外观品质和简单的营养成分分析方面。中国作物信息网(http://www. cgris.net/)登记在册的谷子种质资源中呈现出16种不同的米色, 其中黄色米占77%。小米中的黄色物质主要是类胡萝卜素, 其含量与外观品质显著相关, 是小米品质育种的重要指标。黄米中类胡萝卜素的含量与绿米相当, 但显著高于白色品种[4]。王润奇等[5]利用三体分析法将白米和青米基因分别定位于第4和第6染色体, 为米色基因的克隆奠定了基础。最近, 我们发现谷子番茄红素β-环化酶(lycopene beta cyclase, LCYB)可能通过影响β-胡萝卜素和总类胡萝卜素在籽粒中的积累, 进而影响小米的米色[6]。蒸煮品质和食用品质是小米品质构成的最重要方面, 主要由直链淀粉和支链淀粉的含量及比例决定。直链淀粉含量较低的小米米饭柔软且口感好, 相反, 直链淀粉含量较高的小米蒸煮后米饭干燥、蓬松、色暗, 有回生现象[7]。遗憾的是, 目前有关淀粉合成和调控机制的认识主要来自拟南芥和水稻等模式植物, 谷子相关研究较少[8-9]。
随着高通量测序的简单化和低成本化, 利用二代测序技术大规模开发谷子分子标记并解析小米品质等复杂农艺性状成为可能。2012年, 由美国国家能源部所属的联合基因组研究所和中国华大基因分别进行的豫谷1号和张谷的全基因组测序工作相继完成[10-11], 从此谷子功能基因组学研究进入了一个新的时代。此后, 一大批基于全基因组测序的分子标记相继被开发[12-16]。Bai等[16]对十里香谷子品种进行了基因组重测序, 通过比对发现十里香和豫谷1号基因组之间存在762 082个SNP, 26 802个1~5 bp InDels和10 109个结构变异; 而十里香和张谷基因组之间存在915 434个SNP、28 546个InDels和12 968个结构变异。最近, 王军等[17]利用简化基因组测序技术构建了一张1648.8 cM的谷子高密度遗传图谱, 并定位了8个农艺性状的11个主效QTL。我国谷子种质资源非常丰富, 在长期的自然进化和人工选择中积累了大量自然变异。2013年, 我国科学家联合完成了916份谷子种质资源全基因组低倍重测序和单倍体型图谱的构建工作, 为谷子基因资源的发掘和遗传改良提供了海量的基础数据信息[18]。
晋谷21原名晋汾7号, 由晋汾52经钴60 γ射线辐射和连续单株选育而成[19]。其米色金黄发亮, 细柔光滑, 品质和产量均超过历史名米沁州黄[19]。晋谷21于1992年荣获中国首届农业博览会银奖, 1994年获山西省科技进步一等奖, 1995年被国家科委列入国家成果重点推广项目。本实验室曾对晋谷21进行了转录组测序, 鉴定出614个新基因, 并优化了7175个已注释基因的结构, 为晋谷21优异米质基因的发掘奠定了基础[20]。尽管如此, 由于缺少晋谷21的基因组信息, 限制了晋谷21优异米质连锁分子标记的开发和相关基因的克隆。为此, 本研究利用Illumina HiSeq 2000高通量测序技术对晋谷21进行了全基因组重测序, 并将其与豫谷1号参考基因组比较, 开发了大量InDel和SNP分子标记, 并将其应用于晋谷21衍生种的分析。本研究不仅为晋谷21优异米质相关基因的发掘和分子育种奠定了基础, 同时也为其他谷子品种的相关研究提供了借鉴。
1 材料与方法
1.1 植物材料及培养条件
全基因组重测序材料为大田种植的健康无病虫害的晋谷21单株植株, 选取15份谷子种质资源(表1)进行引物通用性分析。编号7~15的种质资源由中国农业科学院作物科学研究所的刁现民研究员馈赠, 其余资源为本实验室保存或创制。所有材料均种植于山西省太谷县山西农业大学试验田内。
1.2 基因组DNA的提取、文库构建和高通量测序
采用改良的CTAB法[21]提取用于PCR模板的谷子基因组DNA: 取0.1 g新鲜谷子叶片, 加入0.6 mL CTAB提取液, 利用高通量组织研磨机(宁波新芝SCIENTZ-48)充分破碎; 然后加入500 μL氯仿/异戊醇(24∶1, v/v)进行抽提; 最后取上清液加入等体积的异丙醇以沉淀DNA。重测序用晋谷21基因组的提取采用高效植物基因组DNA提取试剂盒[天根生化科技(北京)有限公司, 货号DP350], 提取操作严格按照试剂盒提供的方法进行。利用配备µCuvette G1.0超微量比色皿的BioSpectrometer分光光度计(PCR用)或者Qubit荧光定量仪(重测序用)测定基因组DNA的浓度和纯度, 并用1% (w/v)的琼脂糖凝胶电泳检测DNA质量。
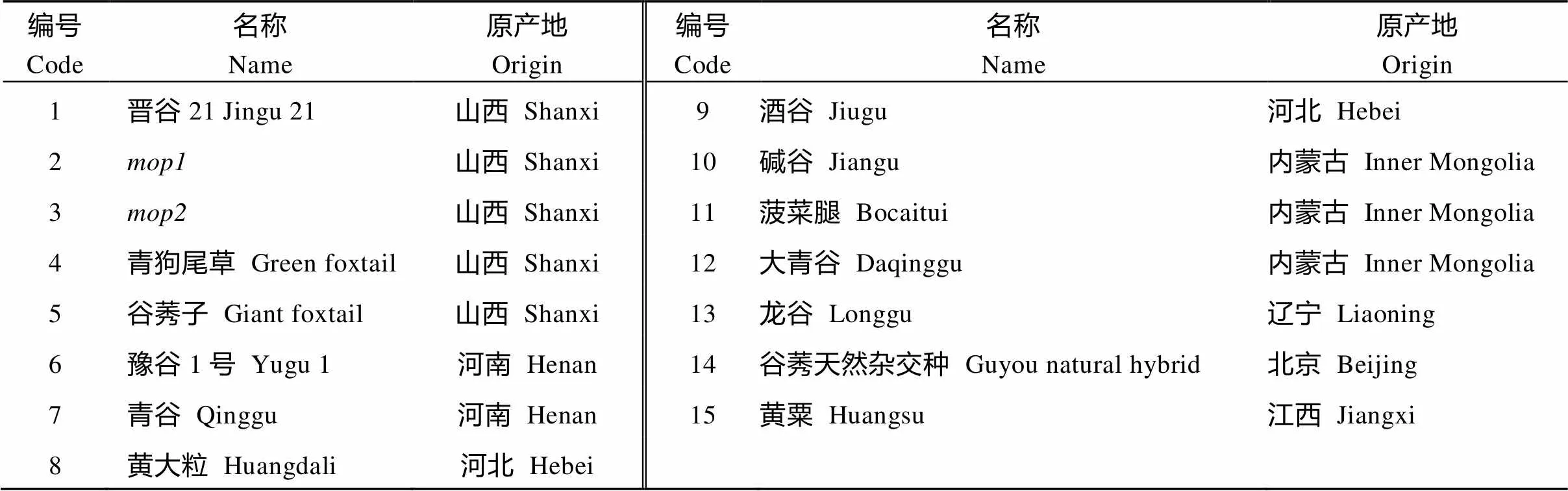
表1 15份谷子种质资源信息
由深圳华大基因科技服务有限公司严格按照Illumina提供的标准方法进行文库的构建和高通量测序: 将5 µg基因组DNA随机打断, 进行片段化; 电泳回收所需长度的DNA片段, 进行末端修复并加上接头制备双端测序文库, 利用Illumina HiSeq 2000测序平台进行高通量测序。
1.3 晋谷21重测序数据分析及InDel和SNP检测
利用Illunima Casava软件对晋谷21经Illumina HiSeq 2000测序平台测序得到的原始图像数据进行碱基识别, 将其转化为原始测序读段(raw reads)。为了保证后续数据分析的质量, 首先将原始测序数据进行过滤, 去除带接头、N所占比例大于5%或质量值≤5的碱基超过50%的低质量测序序列, 从而得到高质量的测序数据。
利用BWA软件[22]将晋谷21重测序数据比对到豫谷1号参考基因组[10], 以定位干净读段(clean reads)在参考基因组上的位置, 统计晋谷21基因组测序深度和基因组覆盖度等信息, 并进行InDel和SNP变异的检测。InDel和SNP的检测利用基因组分析工具包(Genome Analysis Toolkit, GATK) GATK[23]进行, 具体流程为: (1)使用Picard软件包(https://sourceforge.net/projects/picard/)的Mark Duplicate工具去除重复序列, 以消除PCR复制的影响; (2)使用基因组分析工具包(Genome Analysis Toolkit, GATK)[23]对SNP和InDel附近位点进行局部重新比对, 校正由于碱基变异产生的比对结果错误; (3)校正碱基质量值; (4)检测InDel和SNP变异位点。
1.4 基于重测序数据的InDel和CAPS分子标记的开发和引物设计
选取晋谷21和豫谷1号差异碱基数在13~50 bp的InDel位点设计InDel引物, 引物设计原则为扩增片段长度为100~250 bp, 且晋谷21和豫谷1号之间的差异在8%以上。基于晋谷21重测序获得的SNP位点信息, 利用在线软件dCAPS Finder 2.0[24]分析最佳酶切位点和选择限制性内切核酸酶, 并设计酶切扩增多态性(cleaved amplified polymorphic sequence, CAPS)标记引物, 引物设计原则为PCR扩增片段长度500~1000 bp。
1.5 PCR 扩增及标记验证
PCR体系为20 μL, 包括基因组DNA约50 ng、10× PCR 缓冲液 2 μL, 2 mmol L–1dNTPs 2 μL, 10 μmol L–1上下游引物各2 μL和1 UDNA聚合酶。用2.5%琼脂糖凝胶检测PCR或者酶切产物。
2 结果与分析
2.1 晋谷21基因组重测序及测序数据统计分析
利用Illumina paired-end双端测序, 共获得15.45 Gb的晋谷21基因组原始数据(raw data)。经质量评估和过滤, 最终获得83.05 Mb clean reads, 共计14.95 Gb的高质量测序数据, 测序深度达到36.9×(表2)。其中, 质量值大于等于30的碱基达到92.96%, 表明测序质量较好(表2和图1-A)。为保证结果的可靠性, 所有后续分析都基于上述过滤后的高质量数据。
晋谷21基因组重测序数据与豫谷1号参考基因组平均比对效率为96.16%, 其中双端测序序列均定位到参考基因组上, 且距离符合测序片段长度分布的占93.25%, 基因组覆盖度为95.34% (至少覆盖1×) (表2和图1-B)。为了分析插入片段的实际大小, 检测了双端序列在参考基因组上的起止位置。结果表明插入片段长度分布呈正态分布, 且只有一个单峰, 峰值长度约为480 bp, 这表明测序数据文库构建符合要求(图1-C)。

表2 晋谷21全基因组测序数据信息汇总
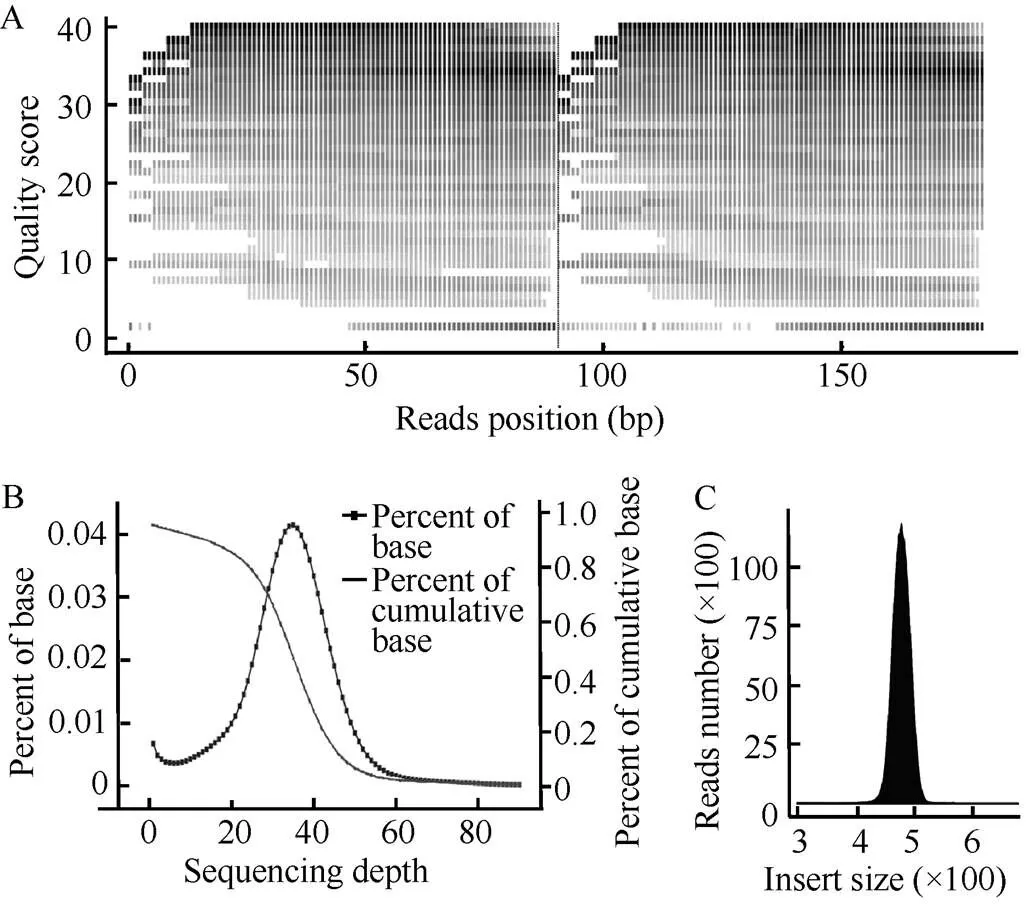
图1 晋谷21基因组测序质量评估
A: 碱基质量分布图; B: 测序深度分布曲线; C: 测序文库插入片段大小分布图。
A: base quality distribution; B: sequencing-depth distribution curve; C: insert size distribution of the sequencing library.
2.2 基于晋谷21重测序数据的InDel分子标记开发
根据样品晋谷21的测序读段在豫谷1号参考基因组上的定位结果, 利用GATK软件包检测晋谷21与豫谷1号之间是否存在InDel。共发现169 037个InDel位点, 平均密度为417.37个 Mb–1, 这些InDel共导致了1752个基因发生变异。其中, 14 578个InDel位点差异片段长度在13~50 bp之间; 34 733个InDel位点差异片段长度在4~12 bp之间; 117 406个InDel位点差异片段长度在1~3 bp之间(图2)。

图2 晋谷21和豫谷1号基因组间的InDel统计
正负数分别表示晋谷21基因组中插入(正数)或者缺失(负数)的碱基数。
The positive and negative numbers indicate the number of insertion (positive) or deletion (negtive) of base in the Jingu 21 genome.
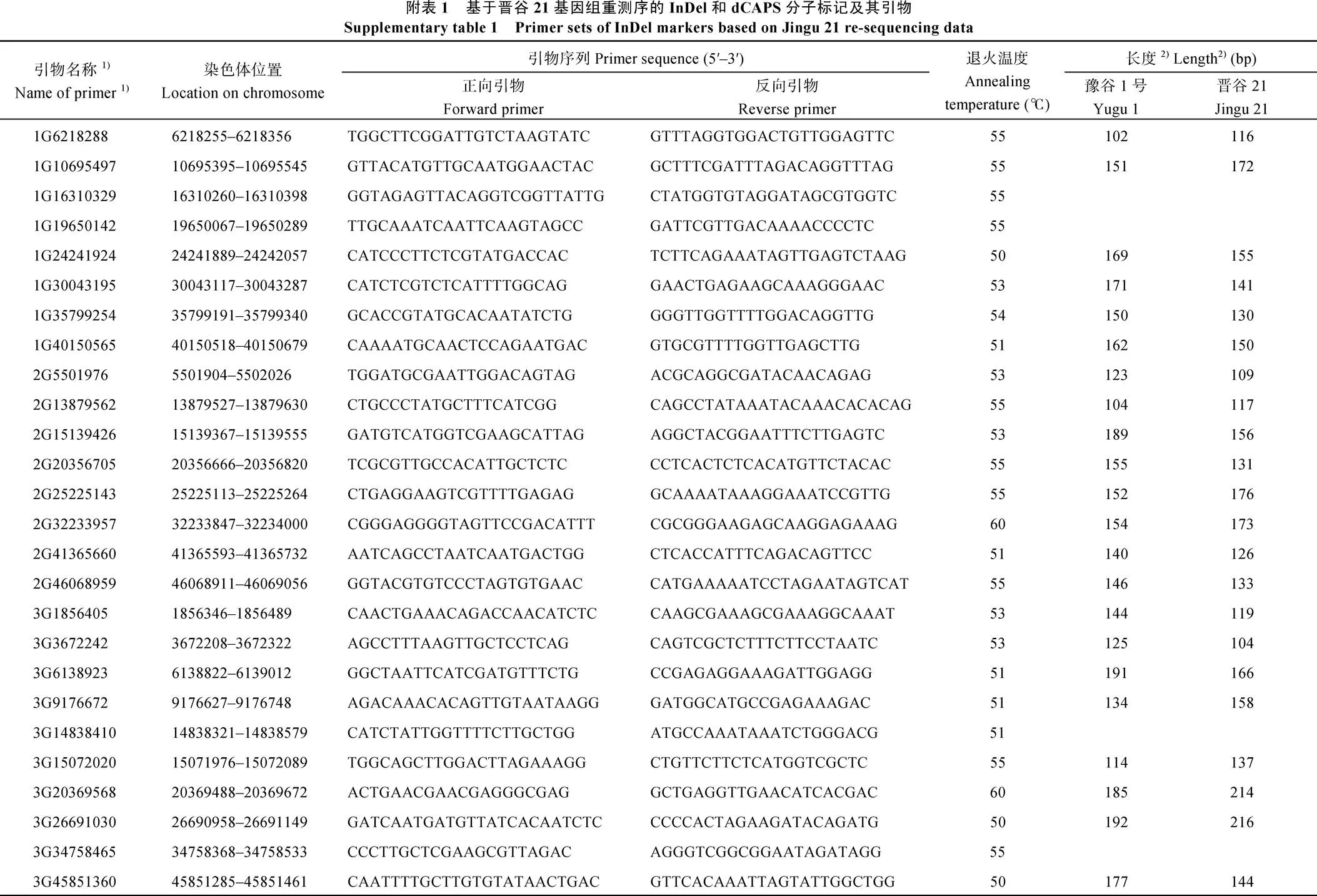
为了进一步验证候选InDel标记的准确性, 根据其在染色体上的分布情况, 筛选并设计了68对代表性InDel标记引物(附表1)。其中, 8对位于第1染色体, 8对位于第2染色体, 11对位于第3染色体, 8对位于第4染色体, 7对位于第5染色体, 6对位于第6染色体, 6对位于第7染色体, 6对位于第8染色体, 8对位于第9染色体(图3和附表1)。为了检测开发的InDel标记的准确性和上述引物的有效性, 以豫谷1号和晋谷21基因组为模板, 进行了PCR扩增和电泳检测。结果表明, 上述68对引物均能扩增出特异条带。进一步分析表明, 61对引物在豫谷1号和晋谷21之间具有多态性, 且扩增片段大小符合预期结果; 7对引物无多态性或者大小与预期不符(图3和附表1)。
2.3 基于晋谷21重测序数据的SNP/CAPS分子标记开发与检测
使用GATK软件工具包对晋谷21基因组DNA进行SNP检测, 共获得1 167 555个SNP, 平均密度为2912.33个 Mb–1。其中, 同类型碱基之间突变的转换型SNP位点851 065个, 不同类型碱基之间的颠换型SNP位点 316 490个; 纯合的SNP位点为1 146 969个, 杂合的SNP位点为20 586个, 纯合率为98.24%。利用SnpEff软件[25]对上述SNP进行了注释, 发生在非编码区的突变为1 117 744个, 发生在编码区的突变为49 811个, 这些SNP共导致7098个基因发生非同义突变(表3)。

图3 InDel标记在晋谷21和豫谷1号的多态性分析
InDel标记详细信息见附表1, 每个分子标记的第1个样品为晋谷21, 第2个样品为豫谷1号。M: D2000 DNA Marker [天根生化科技(北京)有限公司, #MD114]。1~11代表该标记在附表1中的顺序。
The detailed information of the InDel markers are listed in the supplemental table 1. The first and the second lanes of each marker are Jingu 21 and Yugu 1 respectively. M: D2000 DNA Marker [Tiangen Biotech (Beijing) Co. Ltd., #MD114]. 1~11 represent the order of the markers in supplemental Table 1.
为了验证上述SNP位点的准确性, 我们选取了第3染色体位点14 938 250的SNP进行了验证。在该SNP位点参考基因组序列为C, 而晋谷21为碱基T, 从而导致晋谷21产生了一个新的I酶切位点。据此, 设计CAPS标记引物(5′-TTGAGATTTTGC TGGCGATTGG-3′和5′-CGGCATACTTTTGTCACC CTAC-3′), 并利用晋谷21和豫谷1号基因组DNA进行了PCR验证。如图4所示, 晋谷21和豫谷1号都可以扩增出清晰条带, 且二者条带大小相同。利用I酶切后, 晋谷21片段可以切割成424 bp和538 bp两个片段, 而豫谷1号片段不能被切割(图4)。上述结果与预期相符, 表明基于晋谷21重测序数据开发的该SNP位点是正确的。
2.4 基于晋谷21重测序数据开发的分子标记的通用性
选取了1份狗尾草、2份谷莠子和12份谷子, 共计15份种质资源验证基于晋谷21重测序数据开发的InDel和SNP/CAPS分子标记的通用性。结果表明, 9对InDel引物都可以在15个谷子种质资源中进行有效的扩增(表1)。其中标记2G5501976仅在晋谷21背景的种质资源和其他13个种质资源之间有多态性, 而在其他13个种质资源之间无多态性; 其余8个InDel标记在所有种质资源中多态性都较好(图5-A)。类似地, 基于SNP重测序数据开发的CAPS标记3G14938250也可以在其他谷子种质资源中有效扩增, 且利用I酶切后表现出较好的多态性(图5-B)。这表明基于晋谷21全基因组重测序数据开发的InDel和SNP/CAPS分子标记在包括狗尾草和谷莠子在内的其他谷子种质资源中具有通用性, 可用于其他谷子种质资源的相关研究。

表3 SNP注释结果
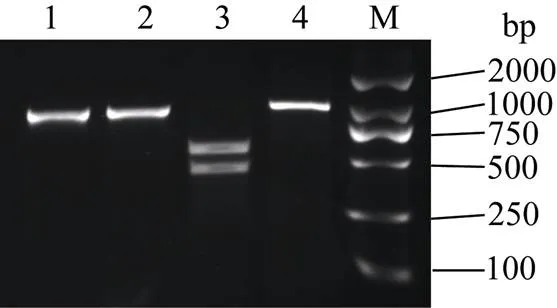
图4 CAPS标记3G14938250对晋谷21和豫谷1号的多态性分析
1: 晋谷21 PCR 扩增产物; 2: 豫谷1号 PCR 扩增产物; 3: 晋谷21 PCR产物的I酶切结果; 4: 豫谷1号PCR 产物I的酶切结果; M: D2000 DNA marker [天根生化科技(北京)有限公司, #MD114]。
1: PCR amplicon of Jingu 21; 2: PCR amplicon of Yugu 1; 3: PCR amplicon of Jingu 21 digested withI; 4: PCR amplicon of Yugu 1 digested withI; M: D2000 DNA marker [Tiangen Biotech (Beijing) Co. Ltd., #MD114].
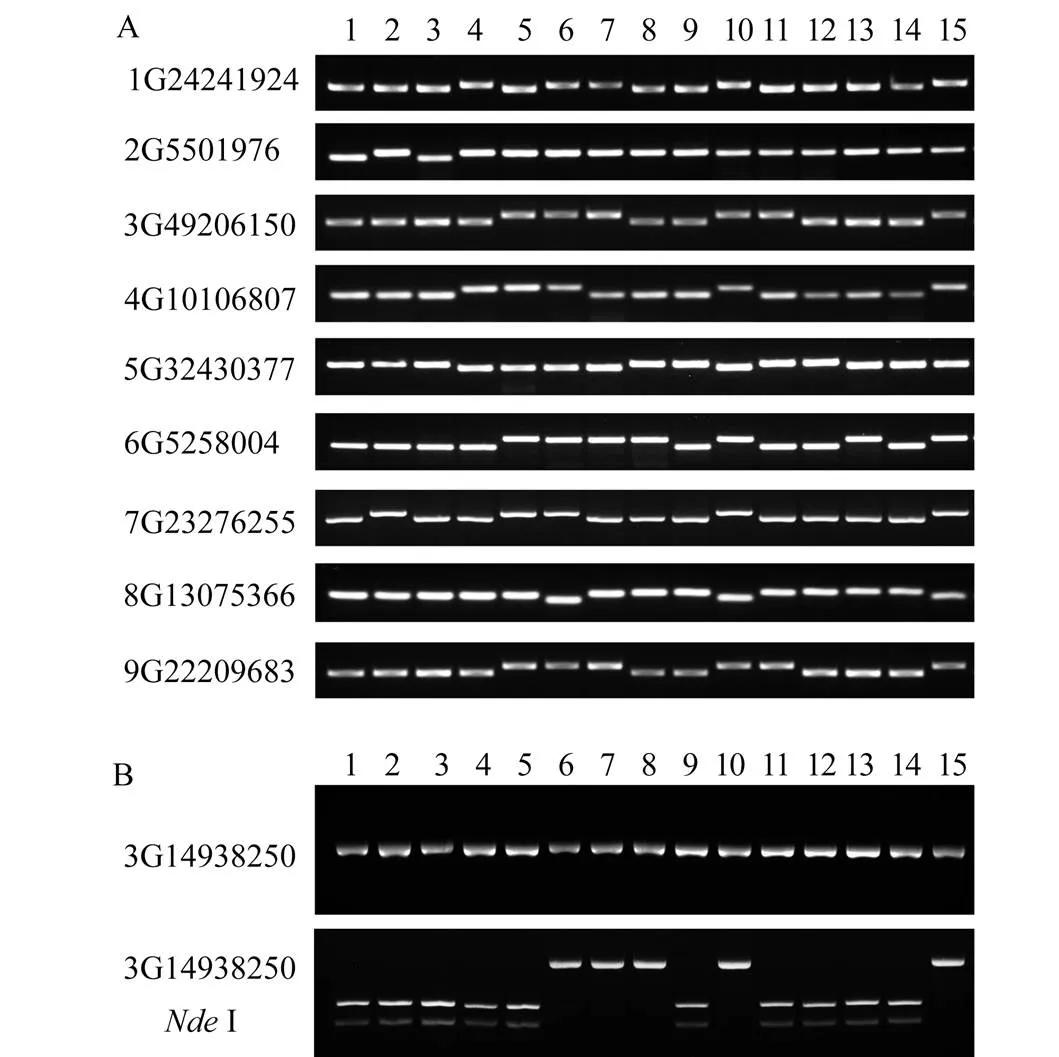
图5 InDel和CAPS标记在15个谷子种质资源的多态性分析
A: InDel标记在15个谷子种质资源的多态性分析; B: CAPS标记在15个谷子种质资源的多态性分析。1~15为谷子种质资源, 详细信息见表1。
A: Polymorphic analysis of InDel markers among 15 foxtail millet germplasm resources; B: Polymorphic analysis of CAPS marker among 15 foxtail millet germplasm resources. The foxtail millet germplasm (1–15) is listed in Table 1.
3 讨论
3.1 基于二代重测序技术开发晋谷21全基因组分子标记
随着高通量测序技术的发展以及豫谷1号和张谷等谷子基因组测序的完成, 利用二代重测序技术在全基因组水平开发分子标记已经成为可能。Jia等[18]对包括晋谷21在内的916份谷子种质资源进行了重测序, 但测序深度较低, 平均仅为0.7×, 导致晋谷21的SNP数据不全, 且无法开发InDel标记。本研究利用Illumina HiSeq 2000高通量测序平台对名优谷子品种晋谷21进行了36.9×深度测序, 并开发了169 037个InDel标记和1 167 555个SNP标记, 对晋谷21优异米质形成分子机制的解析具有重要价值。
与SSR标记一样, InDel标记反映的也是DNA 片段长度的多态性。由于受到测序深度和算法等的限制, 过去往往仅能检测到较小的InDel, 一般小于10 bp[26]。如Bai等[16]利用高通量测序技术开发的谷子InDel标记长度为1~5 bp, 检测这些小的InDel非常困难。为了解决这一问题, 本研究采用了36.9×深度测序, 检测的InDel的长度在1~269 bp之间。其中, 差异片段长度在13~50 bp之间的InDel位点为14 578个, 约占InDel标记总数的8.62% (图2)。这些InDel标记只需要普通琼脂糖凝胶电泳即可检测, 与需要聚丙烯酰胺凝胶电泳检测的SSR标记相比, 可以大大节省人力和物力。
3.2 基于晋谷21重测序数据开发的分子标记的准确性和通用性
设计的68对InDel引物中有61对其扩增片段与预期大小相同, 且呈现出较好的多态性; 而另外7对扩增结果与预期不符, 占所有引物的10.29% (图3)。分别对1G19650142和3G34758465两个标记的豫谷1号和晋谷21扩增片段的测序表明, InDel标记1G19650142 PCR扩增产物为第9染色体23 837 024~ 23 837 239之间216 bp的片段; 而3G34758465 PCR扩增产物为第6染色体10 398 380~10 398 547之间168 bp的片段(王兴春等, 未发表数据)。这表明扩增片段大小与预期不符是非特异扩增而不是高通量测序或者生物信息学分析错误导致的。因此, 建议在设计InDel标记引物时将候选引物序列与豫谷1号参考基因组比对, 选择单一结合位点的引物。
分子标记的通用性可以大大节约开发成本, 提高分子标记利用效率。但以往研究多集中在SSR标记的通用性方面, 而有关InDel和SNP等标记通用性的研究较少[27-28]。本研究表明, 基于晋谷21开发的InDel和SNP/CAPS标记也具有较好的通用性, 可用于其他谷子及其近缘野生种狗尾草和谷莠子的相关研究(图5), 但这些标记是否可用于其他禾谷类作物还有待进一步研究。此外, InDel分子标记2G5501976多态性较差, 在除晋谷21背景之外的13个种质资源之间无多态性。因此, 该InDel标记可以作为晋谷21特异标记, 仅用该标记即可高效区分是否为晋谷21及其衍生品种。
3.3 基于晋谷21重测序数据开发的分子标记在小米品质研究和育种中的应用
小米的品质是一个复杂的数量性状, 受众多基因调控。传统方法开发的分子标记数量有限, 难以对品质等复杂农艺性状相关基因进行精细定位。本研究开发了169 037个InDel位点和1 167 555个SNP位点, 平均密度分别为28.58个 Mb–1和2289.32个 Mb–1, 完全可以满足小米品质等复杂农艺性状基因精细定位的需求。InDel标记2G5501976是晋谷21及其衍生品种的特异分子标记, 是这些品种中基因启动子缺失14 bp导致的。基因编码一个真核生物翻译起始因子, 该基因与晋谷21优异米质的关系还有待进一步研究。此外, 目前水稻中已经克隆了一大批外观品质、营养成分和香味等品质相关的基因[8]。通过序列比对, 筛选谷子中的同源基因, 结合本研究鉴定的InDel和SNP位点, 进一步研究上述品质相关基因在晋谷21中的变异情况, 从而有望揭示晋谷21优异米质形成的分子机制。
4 结论
检测到169 037个InDel位点和1 167 555个SNP位点, 初步揭示了晋谷21的基因组信息。开发了InDel和CAPS分子标记, 为深入解析晋谷21优异米质形成的分子机制和分子标记辅助育种奠定了基础。基于晋谷21重测序数据开发的InDel和SNP标记在狗尾草、谷莠子和谷子等谷子种质资源具有通用性, 可用于谷子种质资源鉴定、遗传分析、基因图位克隆和杂交种鉴定。
致谢:感谢中国农业科学院作物科学研究所的刁现民研究员提供了谷子种质资源(表1编号7~15)。
[1] 张超, 张晖, 李冀新. 小米的营养以及应用研究进展. 中国粮油学报, 2007, 22(1): 51–55 Zhang C, Zhang H, Li J X. Advance of millet research on nutrition and application., 2007, 22(1): 51–55 (in Chinese with English abstract)
[2] 刘敏轩, 陆平. 中国谷子育成品种维生素E 含量分布规律及其与主要农艺性状和类胡萝卜素的相关性分析. 作物学报, 2013, 39: 398–408 Liu M X, Lu P. Distribution of vitamin e content and its correlation with agronomic traits and carotenoids content in foxtail millet varieties in China., 2013, 39: 398–408 (in Chinese with English abstract)
[3] Sharma N, Niranjan K. Foxtail millet: properties, processing, health benefits, and uses., 2017, 34: 329–363
[4] 杨延兵, 管延安, 秦岭, 石慧, 王海莲, 张华文. 不同地区谷子小米黄色素含量与外观品质研究. 中国粮油学报, 2012, 27(1): 14–19 Yang Y B, Guan Y A, Qin L, Shi H, Wang H L, Zhang H W. The studies on yellow pigment content and appearance quality of millet from different regions., 2012, 27(1): 14–19 (in Chinese with English abstract)
[5] 王润奇, 高俊华, 关中波, 毛丽萍. 谷子几种农艺性状基因染色体定位及连锁关系的初步研究. 作物学报, 2007, 33: 9–14 Wang R Q, Gao J H, Guan Z B, Mao L P. Chromosome location and linkage analysis of a few agronomical important traits in foxtail millet., 2007, 33: 9–14 (in Chinese with English abstract)
[6] 张耀元, 路阳, 张彬, 禾璐, 刘龙龙, 王兴春, 李红英, 韩渊怀. 谷子类胡萝卜素生物合成途径基因与米色形成的关系. 分子植物育种, 2016, 14: 1341–1351 Zhang Y Y, Lu Y, Zhang B, He L, Liu L L, Wang X C, Li H Y, Han Y H. The relationship between the generelated to carotenoid synthesis and the colored formation of foxtail millet., 2016, 14: 1341–1351 (in Chinese with English Abstract)
[7] 刘辉, 张敏. 不同品种小米的直链淀粉含量与快速黏度分析仪谱特征值关系研究. 食品科学, 2010, 31: 31–33 Liu H, Zhang M. Relationships between amylose content and gelatinization characteristics of different varieties of millet., 2010, 31: 31–33 (in Chinese with English abstract)
[8] 张昌泉, 赵冬生, 李钱峰, 顾铭洪, 刘巧泉. 稻米品质性状基因的克隆与功能研究进展. 中国农业科学, 2016, 49: 4267–4283 Zhang C Q, Zhao D S, Li Q F, Gu M H, Liu Q Q. Progresses in research on cloning and functional analysis of key genes involving in rice grain quality.. 2016, 49: 4267–4283 (in Chinese with English abstract)
[9] He L, Zhang B, Wang X, Li H Y, Han Y H. Foxtail millet: nutritional and eating quality, and prospects for genetic improvement., 2015, 2: 124–133
[10] Bennetzen J L, Schmutz J, Wang H, Percifield R, Hawkins J, Pontaroli A C, Estep M, Feng L, Vaughn J N, Grimwood J, Jenkins J, Barry K, Lindquist E, Hellsten U, Deshpande S, Wang X, Wu X, Mitros T, Triplett J, Yang X, Ye C Y, Mauro-Herrera M, Wang L, Li P, Sharma M, Sharma R, Ronald P C, Panaud O, Kellogg E A, Brutnell T P, Doust A N, Tuskan G A, Rokhsar D, Devos K M. Reference genome sequence of the model plant., 2012, 30: 555–561
[11] Quan Z W, Cheng S F, Xu X, Pan S K, Xie M, Zeng P, Yue Z, Wang W L, Tao Y, Bian C L, Han C L, Xia Q J, Peng X H, Cao R, Yang X H, Zhan D L, Hu J C, Zhang Y X, Li H N, Li H, Li N, Wang J Y, Wang C C, Wang R Y, Guo T, Cai Y J, Liu C Z, Xiang H T, Shi Q H, Huang P, Chen Q C, Li Y R, Wang J, Zhao Z H, Wang J. Genome sequence of foxtail millet () provides insights into grass evolution and biofuel potential., 2012, 30: 549–554
[12] Zhang S, Tang C, Zhao Q, Li J, Yang L, Qie L, Fan X, Li L, Zhang N, Zhao M, Liu X, Chai Y, Zhang X, Wang H, Li Y, Li W, Zhi H, Jia G, Diao X. Development of highly polymorphic simple sequence repeat markers using genome-wide microsatellite variant analysis in Foxtail millet [(L.) P. Beauv]., 2014, 15: 78
[13] Fang X, Dong K, Wang X, Liu T, He J, Ren R, Zhang L, Liu R, Liu X, Li M, Huang M, Zhang Z, Yang T. A high density genetic map and QTL for agronomic and yield traits in Foxtail millet [(L.) P. Beauv]., 2016, 17: 336
[14] Venkata S B, Muthamilarasan M, Misra G, Prasad M. FmMDb: a versatile database of foxtail millet markers for millets and bioenergy grasses research., 2013, 8: e71418
[15] Yadav C B, Bonthala V S, Muthamilarasan M, Pandey G, Khan Y, Prasad M. Genome-wide development of transposable elements- based markers in foxtail millet and construction of an integrated database., 2015, 22: 79–90
[16] Bai H, Cao Y, Quan J, Dong L, Li Z, Zhu Y, Zhu L, Dong Z, Li D. Identifying the genome-wide sequence variations and developing new molecular markers for genetics research by re-sequencing a Landrace cultivar of foxtail millet., 2013, 8: e73514
[17] Wang J, Wang Z, Du X, Yang H, Han F, Han Y, Yuan F, Zhang L, Peng S, Guo E. A high-density genetic map and QTL analysis of agronomic traits in foxtail millet [(L.) P. Beauv.] using RAD-seq., 2017, 12: e0179717
[18] Jia G Q, Huang X H, Zhi H, Zhao Y, Zhao Q, Li W J, Chai Y, Yang L F, Liu K Y, Lu H Y, Zhu C R, Lu Y Q, Zhou C C, Fan D, Weng Q J, Guo Y L, Huang T, Zhang L, Lu T T, Feng Q, Hao H F, Liu H K, Lu P, Zhang N, Li Y H, Guo E H, Wang S J, Wang S Y, Liu J R, Zhang W F, Chen G Q, Zhang B J, Li W, Wang Y F, Li H Q, Zhao B H, Li J Y, Diao X M, Han B. A haplotype map of genomic variations and genome-wide association studies of agronomic traits in foxtail millet ()., 2013, 45: 957–961
[19] 陈瑛, 卫天业, 景小兰, 张象贤. 晋谷21号的选育及推广前景. 山西农业科学, 1992, (12): 7–8 Chen Y, Wei T Y, Jing X L, Zhang X X. Breeding and popularization prospect of Jingu 21., 1992, (12): 7–8 (in Chinese without English abstract)
[20] 穆彩琴, 张瑞娟, 屈聪玲, 韩渊怀, 王兴春, 杨致荣. 基于RNA-Seq技术的谷子新基因发掘及基因结构优化. 植物生理学报, 2016, 52: 1066–1072 Mu C Q, Zhang R J, Qu C L, Han Y H, Wang X C, Yang Z R. Identifi cation of novel genes and optimization of annotated genes in foxtail millet by RNA-Seq technology., 2016, 52: 1066–1072 (in Chinese with English abstract)
[21] Stewart C N J, Via L E. A rapid CTAB DNA isolation technique useful for RAPD fingerprinting and other PCR applications., 1993, 14: 748–750
[22] Li H, Durbin R. Fast and accurate short read alignment with Burrows-Wheeler transform., 2009, 25: 1754– 1760
[23] McKenna A, Hanna M, Banks E, Sivachenko A, Cibulskis K, Kernytsky A, Garimella K, Altshuler D, Gabriel S, Daly M, DePristo M A. The Genome Analysis Toolkit: a MapReduce framework for analyzing next-generation DNA sequencing data., 2010, 20: 1297–1303
[24] Neff M M, Turk E, Kalishman M. Web-based primer design for single nucleotide polymorphism analysis., 2002, 18: 613–615
[25] Cingolani P, Platts A, Wang L, Coon M, Nguyen T, Wang L, Land S J, Lu X, Ruden D M. A program for annotating and predicting the effects of single nucleotide polymorphisms, SnpEff: SNPs in the genome ofstrain w;;.(Austin), 2012, 6: 80–92
[26] Liu B, Wang Y, Zhai W, Deng J, Wang H, Cui Y, Cheng F, Wang X, Wu J. Development of InDel markers forbased on whole-genome re-sequencing., 2013, 126: 231–239
[27] 张体付, 戚维聪, 顾闽峰, 张晓林, 李坦, 赵涵. 藜麦EST-SSR 的开发及通用性分析. 作物学报, 2016, 42: 492–500 Zhang T F, Qi W C, Gu M F, Zhang X L, Li T, Zhao H. Exploration and transferability evaluation of EST-SSRs in quinoa., 2016, 42: 492–500 (in Chinese with English abstract)
[28] Ul Haq S, Kumar P, Singh R K, Verma K S, Bhatt R, Sharma M, Kachhwaha S, Kothari S L. Assessment of functional EST-SSR markers (sugarcane) in cross-species transferability, genetic diversity among poaceae plants, and bulk segregation analysis., 2016, doi: 10.1155/2016/7052323:7052323
Genome-wide Identification of Molecular Markers Based on Genomic Re-sequencing of Foxtail Millet Elite Cultivar Jingu 21
ZHAO Qing-Ying1,**, ZHANG Rui-Juan2,**, WANG Rui-Liang2, GAO Jian-Hua2,3, HAN Yuan-Huai3,4, YANG Zhi-Rong1,3,4,*, and WANG Xing-Chun2,3,4,*
1College of Arts and Sciences, Shanxi Agricultural University, Taigu 030801, Shanxi, China;2College of Life Sciences, Shanxi Agricultural University, Taigu 030801, Shanxi, China;3Institute of Agricultural Bioengineering, Shanxi Agricultural University, Taigu 030801, Shanxi, China;4Shanxi Key Laboratory of Genetic Resources and Genetic Improvement of Minor Crops, Taigu 030801, Shanxi, China
Foxtail millet becomes more and more popular for its rich nutrients, and the grain quality is the key concern that consumers would consider when selecting millet brand. Jingu 21 is an elite cultivar with high edible quality. However, the lack of genomic information impedes studies on the molecular mechanisms of millet quality formation. Here, we re-sequenced the whole genome of Jingu 21 using high-throughput sequencing technology, and obtained 14.95 Gb high quality data. By comparing sequence of Jingu 21 with the reference genome of Yugu 1, we identified 169 037 InDels and 1 167 555 SNPs. Of these InDels, 14 578 could be detected easily by agarose gel electrophoresis. One SNP and 68 InDel markers were selected to verify the polymorphism between Jingu 21 and Yugu 1, showing that the InDel and SNP markers developed by using next generation sequencing technology were reliable. Although the InDel and SNP markers were generated based on genome re-sequencing data of the elite cultivar Jingu 21, they could also be used for research on other foxtail millet, green foxtail, and giant foxtail. Moreover, a specific InDel marker 2G5501976 for Jingu 21 was developed, which could be used to identify Jingu 21 and its derivative varieties. Taken all together, the genomic characterization of Jingu 21 not only lays a foundation for elucidating the molecular mechanisms of high quality formation, but also provides a large number of molecular markers for marker-assisted selection of high quality millet, genetic analysis and map-based cloning.
foxtail millet; Jingu 21; InDel; SNP; molecular marker; genome re-sequencing
2017-09-11;
2018-01-08;
2018-01-29.
10.3724/SP.J.1006.2018.00686
本研究由国家自然科学基金项目(31471502, 31600289, 31371693, 31771810), 山西省自然科学基金项目(201601D011071), 山西省回国留学人员科研资助项目(2015-067)和山西省留学回国人员科技活动择优资助项目(2014-11)资助。
This study was supported by the National Natural Science Foundation of China (31471502, 31600289, 31371693, 31771810), the Natural Science Foundation of Shanxi Province (201601D011071), the Research Project from Shanxi Scholarship Council of China (2015-067) and the Fund Program for the Scientific Activities of Selected Returned Overseas Professionals in Shanxi Province (2014-11).
杨致荣, E-mail: zryangsx@163.com; 王兴春, E-mail: wxingchun@163.com
**同等贡献(Contributed equally to this work)
E-mail: 192015271@qq.com
http://kns.cnki.net/kcms/detail/11.1809.S.20180128.2016.008.html

