基于SSR标记的黍稷种质资源遗传多样性及亲缘关系研究
薛延桃,陆平,乔治军,刘敏轩,王瑞云1,
基于SSR标记的黍稷种质资源遗传多样性及亲缘关系研究
薛延桃1,2,陆平2,乔治军3,刘敏轩2,王瑞云1,3
(1山西农业大学农学院,山西太谷 030801;2中国农业科学院作物科学研究所,北京 100081;3山西省农业科学院农作物品种资源研究所/农业部黄土高原作物基因资源与种质创制重点实验室/杂粮种质资源发掘与遗传改良山西省重点实验室,太原 030031)
【目的】利用SSR标记,分析黍稷种质资源(野生材料和地方品种)的遗传多样性水平,揭示不同来源黍稷种质资源的亲缘关系和遗传群体结构差异,为黍稷起源进化研究奠定基础。【方法】用6份地理差异显著的黍稷种质资源对137对小宗作物课题组开发的具有多态性的SSR引物进行初步筛选,最终筛选103对条带清晰、扩增良好且多态性稳定的SSR引物,利用这103对多态性SSR标记对146份黍稷材料进行PCR扩增,通过遗传参数、聚类、遗传结构等分析,评估不同个体间及不同群体间的遗传多样性,探讨遗传结构差异。【结果】103对SSR标记共检测出308个等位基因(Na),平均值为2.99,平均Shannon-Weaver指数(I)为0.8478,平均期望杂合度为0.3642,平均多态性信息含量指数()为0.5544。103对SSR标记的分布区间为0—1、1—2、2—3、3—4和4—5,分辨率范围为0.334—4.002,77.67%的标记分布于区间1—4,具有适度分辨力。国内资源的观测等位基因数(2.9126)、多样性指数(0.8302)、期望杂合度(0.5023)、多态性信息含量指数(0.5278)均高于国外资源,遗传多样性更丰富。12个群体的遗传距离的变化范围为0.0783—0.5762,均值为0.2938;遗传一致度变化范围为0.5620—0.9247,均值为0.75,遗传相似性与地理分布具有一定相关性,地理分布越近,遗传距离越小,遗传一致度越高。聚类分析在遗传距离为0.15处可以把12个群体分为4个组群,其中南美洲和山西资源各自独立分为一支,与其他资源亲缘关系较远。个体间聚类中,国内外资源划分非常显著,在遗传距离为0.63处,146份黍稷资源可分为3大组群,组群Ⅰ和组群Ⅱ为国外资源,组群Ⅲ为国内资源。组群Ⅱ在遗传距离为0.39处又分为3个亚群,组群Ⅲ在遗传距离为0.45处分为5个亚群,其中亚洲与欧洲资源、中国河北与中国山西、中国内蒙古资源的遗传关系较近。遗传结构分析结果显示国内外群体间存在明显的遗传分化,其中5个组群(组群2、组群5、组群6、组群7和组群9)为国内野生资源特有基因型,分布较为分散;2个组群(组群1和组群4)为国外资源特有基因型,分布较为集中。中国宁夏、南美洲资源的群体结构趋向单一化,中国河北、中国黑龙江、亚洲资源的群体结构趋向多元化。UPGMA聚类结果与遗传结构分析结果一致,且不同地区黍稷资源群体间遗传关系远近均与其地理分布相关。【结论】野生资源的遗传多样性高于国外资源,其中中国河北群体的遗传多样性最丰富,中国河北可能是黍稷的起源中心。
黍稷;野生糜子;国外品种;SSR标记;遗传多样性;群体结构
0 引言
【研究意义】作物种质资源是开展作物品种选育的物质基础,稀有特效种质对推动育种进程具有决定性作用,新的育种目标能否实现决定于育种单位所拥有的种质资源,此外,种质资源也是开展生物学理论研究和分子遗传的重要基础材料,因此,开展种质资源的收集、评估、保存和利用对中国农业研究和发展具有十分重要的意义。黍稷(L.),属于禾本科黍属(L.)[1],一年生草本植物[2]。它是最古老的农作物之一,有10 000多年的栽培历史,在世界范围内都有种植[3-4]。黍稷生长周期短,抗旱耐贫瘠,是干旱半干旱地区的重要作物[5-6]。中国目前保存黍稷资源8 800多份,准确评估黍稷资源在形态学和分子水平的遗传多样性,有利于其合理开发与利用,并促进其育种工作和基础理论方面的研究[7]。【前人研究进展】遗传多样性是生物多样性的重要组成部分,通过遗传多样性分析可以为各稀有物种的收集、育种应用以及起源进化研究提供有利数据。早期关于黍稷遗传多样性的研究多集中于形态学特征。胡兴雨等[8]和董孔军等[9]分别对国家种质库的8 016份黍稷种质资源和甘肃省600份黍稷地方资源的11个农艺性状进行主成分分析和聚类分析,评估了参试材料在形态学水平上的遗传多样性并以此为基础构建了黍稷初级核心种质。近年来,随着分子生物技术的快速发展,分子标记在黍稷遗传多样性研究方面也得到了较广泛的应用。在不同类型的分子标记中,SSR标记具有多等位基因、共显性和高度多态性等优点[10],广泛应用于作物的遗传多样性评价中,是评估遗传多样性的最佳选择[11-13]。Cho等[14]、王银月等[15]和Rajput等[16]采用SSR-PCR技术分别开发了25对、116对、254对多态性较好的黍稷特异性SSR标记。王瑞云等[17]开发了85对高基元SSR标记,其分辨能力强,扩增多态性好;朱建楚[18]、董俊丽等[19]将分子水平和表型结合对黍稷资源进行了遗传多样性分析;Hu等[20]、Hu等[21]、连帅等[22],Liu等[23]和王瑞云等[24]分别利用SSR标记评估了不同地理来源的黍稷资源的遗传多样性,研究表明聚类结果与地理分布密切相关,王瑞云等[17]和Hu等[21]的研究结果表明黄土高原地区的遗传多样性最丰富,推测黄土高原可能是黍稷的起源地。连帅等[25]利用63对SSR多态性引物对192份黍稷资源进行遗传多样性分析,结果表明,国内外群体遗传分化不明显,群体间亲缘关系较近,个体间存在相互渗透。【本研究切入点】目前,黍稷遗传多样性研究多数集中于栽培品种与地方品种的研究,有关黍稷野生资源和国外地方品种的遗传多样性,特别是分子水平群体遗传结构的研究鲜有报道。【拟解决的关键问题】本研究利用本课题组开发的黍稷基因组SSR标记对近年来新引进的国内主栽区的黍稷野生资源以及国外资源共计146份材料进行SSR标记水平的遗传多样性分析,旨在揭示不同来源黍稷种质资源的遗传多样性和群体遗传结构差异水平,为进一步开展黍稷起源地、传播途径以及性状进化等研究奠定基础。
1 材料与方法
1.1 材料
所用的146份黍稷资源(12个群体)均由中国农业科学院作物科学研究所小宗作物课题组近几年新收集和引进,包括85份国内野生资源和新引进的61份国外地方品种(电子附表1)。
1.2 DNA的提取与检测
选取50 mg室内花盆培养出苗20 d左右的幼叶,用DNA提取试剂盒(北京鼎国昌盛生物有限公司)提取基因组DNA。采用1%的琼脂糖凝胶电泳检测DNA的质量,用超微量分光光度计(P360)测定其浓度。调试其终浓度至30 ng·μL-1,-20℃保存备用。
1.3 SSR分析
选用6份地理差异显著的黍稷种质资源对137对小宗作物课题组开发的具有多态性的SSR引物进行初步筛选,最终筛选103对条带清晰、扩增良好且多态性稳定的SSR引物,用于后续遗传多样性分析。
PCR扩增反应在MY-CYCLER PCR仪上进行。PCR反应体系包括1.6 μL 10×PCR buffer、0.8 μL 2.5 mmol·L-1dNTP、0.1 μL Taq酶(5.0 U·μL-1)、0.5 μL引物、0.1 μL DNA模版和5.5 μL ddH2O。PCR扩增程序为94℃ 5 min;94℃ 45 s,55℃ 50 s,72℃ 1 min,39个循环;72℃ 10 min;4℃保存。用8%的聚丙烯酰胺凝胶电泳分离PCR产物,硝酸银染色显影。
1.4 数据分析
分析SSR标记扩增条带,如果在至少2份资源中观察到不同大小的2条DNA条带,则SSR标记被认为是多态性的,有条带记为“1”,无条带记为“0”。利用以下公式计算每个SSR标记物的分辨率(resolving power,Rp值):Rp=∑,=1-(2×︱0.5-︱),其中为某等位基因信息量,为某等位基因在6份材料中出现的频率。分别按照地理来源与国家来源,将146份黍稷资源分为12个群体和30个群体,利用PowerMarker3.25和MEGA7计算不同群体间和群体内各位点的值及遗传距离,并绘制聚类图。利用PopGen1.32计算不同群体间和群体内各位点的遗传多样性指标,包括观测等位基因数(observed number of alleles,Na)、有效等位基因数(effective number of alleles,Ne)、观测杂合度(observed heterozygosity,Ho)、期望杂合度(expected heterozygosity,He)及Shannon-Weaver指数(I)。利用Structure2.3.4对黍稷资源进行遗传结构分析。
2 结果
2.1 SSR标记分析
使用103对SSR标记对146份糜子资源进行检测(表1),共检测出308个等位基因(Na),平均值为2.9903,其中有效等位基因(Ne)为236.55个,平均值为2.2966,所占比重为76.8%。检测的等位基因数的范围为1(BM994、BM5191和LMX635)—6(BM5038),其中,62对标记检测到多个等位基因(3—6),38对标记检测到2个等位基因,3对标记检测到单个等位基因。
Shannon-Weaver指数范围(I)为0(BM994、BM5191和LMX635)—1.5964(BM5181),平均值为0.8478.。观测杂合度(Ho)范围为0(BM114、BM136、BM994、BM5191和LMX635)—0.9920(BM4851),平均值为0.4871。期望杂合度(He)范围为0(BM994、BM5191和LMX635)—0.7992(BM5181),平均值为0.3642。Nei's期望杂合度范围为0(BM994、BM5191和LMX635)—0.7951(BM5181)。值范围为0.0640(BM994)—0.8035(BM4956),平均值为0.5544。一些SSR标记(BM114、BM136、BM994、BM5191和LMX635)的Shannon-Weaver指数、观测杂合度、期望杂合度、Nei's期望杂合度为0,表明个体之间存在普遍异交。
引物分辨率(Rp值)指不同标记对不同基因型的辨别能力,引物分辨率越高,其对不同基因的分辨能力越强,因此从各标记的高分辨能力也可以观察到这些标记的高多态性。103对SSR标记的Rp值范围为0.334(BM1533、BM4739、BM4741、BM4947、LMX635和LMX1429)—4.002(LMX1761)。Rp值的分布区间为0—1、1—2、2—3、3—4和4—5,分布频率分别为21.36%、45.63%、26.21%、5.83%和0.97%(图1),由以上数据可知,77.67%的标记分布区间1—4,具有适度分辨力,选择有效的标记利于后续遗传多样性的分析。
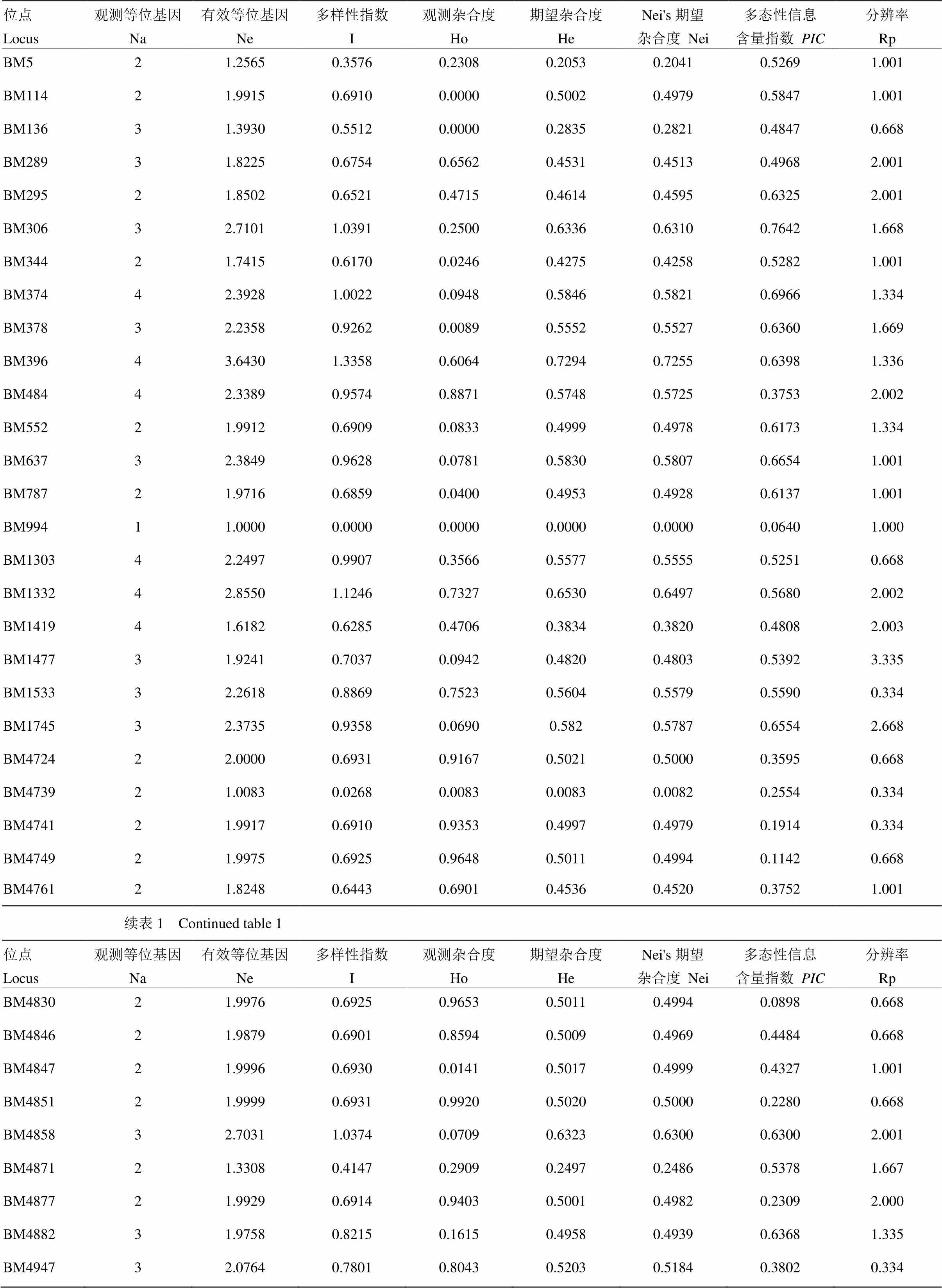
表1 103对SSR标记的遗传参数

图1 103个SSR标记的分辨率(Rp值)
2.2 不同群体间的遗传多样性分析
利用PopGen1.3.2软件分析12个不同群体间黍稷资源的遗传多样性(表2),结果表明,各群体间等位基因(Na)变化范围为1.4947—2.6602,最大的为中国河北群体,最小的为南美洲群体。有效等位基因(Ne)变化范围为1.4510—2.0961,均值为1.8037。就国内外两大群体而言(表3),国内各群体的有效等位基因数(2.2620±0.7862)大于国外群体(2.0429±0.6601),国内群体间的有效等位基因范围为1.6302—2.0961(河北>黑龙江>辽宁>宁夏>内蒙古>山西),均值为1.8740,比全部参试群体的平均有效等位基因高0.0703。国外群体间的有效等位基因范围为1.4510—1.9860(欧洲>亚洲>北美洲>大洋洲>非洲>南美洲),均值为1.7334,比全部参试群体的平均有效等位基因低0.0703。由以上数据可知国内外群体间遗传丰富度差异不大,国内野生糜子遗传丰富度略大于国外地方品种。
Shannon-Weaver指数(I)范围为0.3246—0.7608,最大的为中国河北群体,最小的为南美洲群体。国内群体Shannon-Weaver指数(0.8302)大于国外群体(0.7434),其不同群体的变化趋势与Ne一致。从Nei’s基因杂合度分析,观测杂合度(Ho)最大的是中国辽宁群体(0.3443),最小的是中国黑龙江群体(0.2727),均值为0.3123。期望杂合度(He)最大的是中国河北群体(0.4789),最小的是南美洲群体(0.3123),均值为0.4143。国内外不同群体的观测杂合度和期望杂合度的变化趋势不一致,国内群体的Ho值(0.2972±0.3030)小于国外群体(0.3172±0.3405),He值(0.5023±0.1909)大于国外群体(0.4581±0.1967)。各参试群体的值变化范围为0.1383(南美洲)—0.4898(中国黑龙江),国内群体的值大于国外群体。
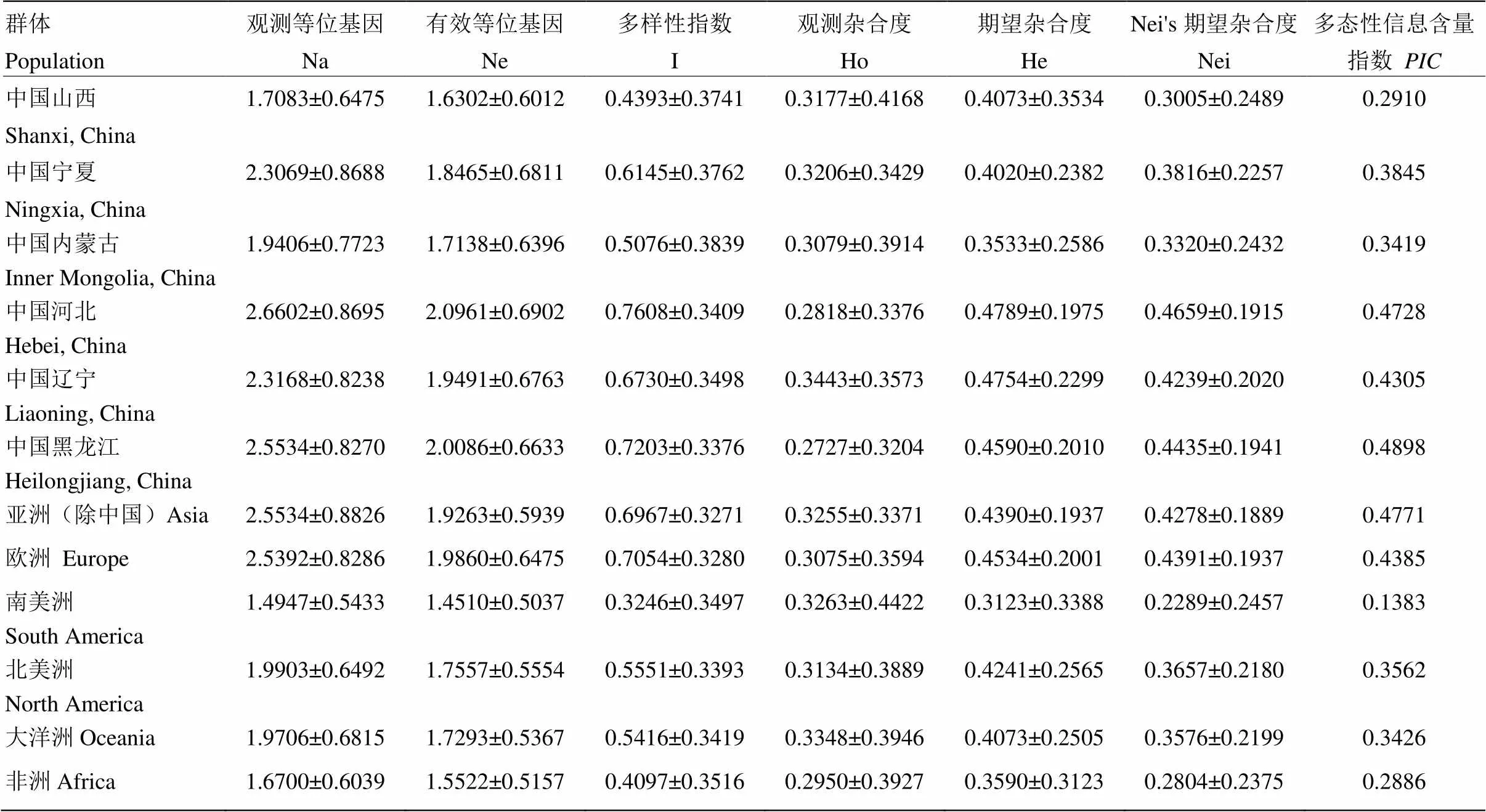
表2 12个群体的遗传多样性分析
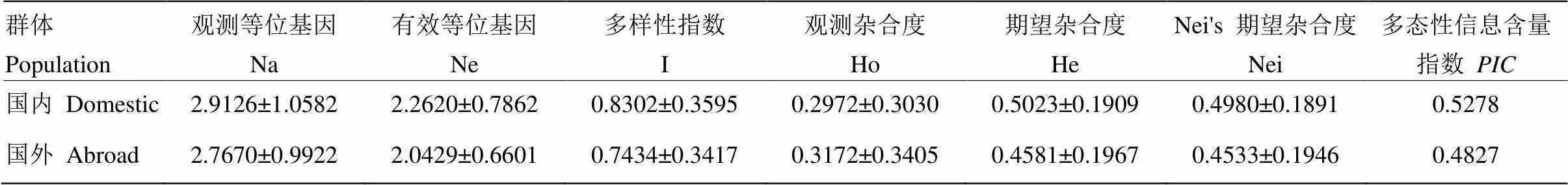
表3 国内外群体的遗传多样性分析
综合以上遗传参数可知,国内资源的遗传多样性高于国外资源。国内群体中河北、黑龙江的遗传多样性较高,国外群体中亚洲、欧洲的遗传多样性较高。
2.3 不同群体间遗传相似性分析
利用PopGen1.3.2软件分析12个群体的遗传相似性(表4),结果表明,遗传距离的变化范围为0.0783—0.5762,均值为0.2938。遗传一致度变化范围为0.5620—0.9247,均值为0.7500。欧洲群体和亚洲群体的遗传距离最小(0.0783),遗传一致度最大(0.9247),说明这两个群体的亲缘关系较近。而南美洲群体和中国山西群体的遗传距离最大(0.5762),遗传一致度最小(0.5620),两者的亲缘关系较远。由以上分析可知,遗传相似性与地理分布具有一定相关性,地理分布越近,遗传距离越小,遗传一致度越高,亲缘关系越近。
2.4 UPMGA聚类分析
基于UPMGA对12个群体进行聚类(图2),遗传距离0.15处,12个群体分为4个组群,分别为组群Ⅰ、组群Ⅱ、组群Ⅲ、组群Ⅳ。组群Ⅰ和组群Ⅱ为国外群体,组群Ⅲ和组群Ⅳ为国内群体。遗传距离为0.18处南美洲群体独立分为一支,与其余国内外群体遗传关系均较远,从地理上看,它与别的群体相距均较远,各种环境因素、气候条件的不同可能是造成该结果的主要原因。遗传距离为0.15处,中国山西群体独立分为一支,与其他国内群体相差较远。国内群体中,中国河北群体、黑龙江群体、辽宁群体遗传关系较近,值得注意的是,河北群体和黑龙江群体尽管地理分布相距较远,但两者的遗传距离最小,且聚在同一个亚群。国外群体中,亚洲群体、欧洲群体和北美洲群体遗传关系较近,大洋洲群体次之。

表4 12个群体遗传距离和遗传一致度
将国外群体按不同的国家分为24个群体,然后与国内6个群体进行聚类(图3),结果显示,遗传距离为0.20处,国内群体与国外群体有明显的分离,与以上结果一致。但是,从图中可以看出,遗传距离为0.10处,国外群体被分为若干个小群,几乎没有分为大的聚类,总体而言,亚洲与欧洲各群体的遗传关系较近,与12个群体间聚类的结果吻合。
基于UPGMA对146份个体进行聚类(图4),结果显示国内外资源划分非常显著。在遗传距离为0.63处,146份黍稷资源分为3大组群,组群Ⅰ和组群Ⅱ为国外资源,组群Ⅲ为国内资源。组群Ⅰ包括2份资源,均为亚洲资源。在遗传距离为0.39处,组群Ⅱ可分为3个亚群,分别为亚群A、亚群B和亚群C。亚群A包括29份资源,分别为亚洲6份、欧洲15份、大洋洲3份、北美洲3份和非洲2份。亚群B包括24份资源,分别为亚洲13份、欧洲5份、南美洲2份、大洋洲2份、非洲1份和北美洲1份。亚群C包括6份资源,分别为亚洲4份、欧洲1份和北美洲1份。在遗传距离为0.45处,组群Ⅲ可分为5个亚群,分别为亚群D、亚群E、亚群F、亚群G和亚群H。亚群D包括4份资源,中国山西1份和中国河北3份。亚群E包括20份资源,分别为中国辽宁2份和中国黑龙江18份。亚群F包括10份资源,分别为中国河北2份、中国辽宁2份和中国黑龙江5份。亚群G包括21份资源,分别为中国宁夏15份、中国内蒙古5份和中国河北1份。亚群H包括30份资源,分别为中国河北20份、中国山西2份、中国内蒙古6份和中国辽宁2份。从各组群和亚群分布可以看出,亚洲与欧洲资源、中国河北与中国山西、中国内蒙古资源的遗传关系较近,各组群分布与地理来源有一定关联。
2.5 遗传结构分析
利用Structure2.3.4软件对146份黍稷资源进行群体结构分析(图5),并根据对数似然方差Var[Lnp](D)]对值绘制折线图,最佳分组应为9组。国内外群体间存在明显的遗传分化(表5),组群2、组群5、组群6、组群7和组群9是国内野生资源特有基因型,各省份群体分布较为分散,其中中国河北、中国黑龙江、中国辽宁资源集中分布在组群5、组群7和组群9,中国宁夏、中国内蒙古资源集中分布在组群2和组群6,各组群分布与地理来源基本相符。组群1和组群4为国外材料特有基因型,各群体分布更为集中。组群3中除2份亚洲资源外,其余均为国内野生资源(80%)组群8中除1份中国山西资源外,其余均为国外资源(97%),中国山西野生糜子和亚洲其他国家地方品种存在不同程度的遗传混杂,遗传关系相对较近。此外,中国宁夏资源全部分布在组群2,南美洲资源全部聚集在组群8,表明这两个地区的黍稷资源的群体结构趋向单一化。相反,中国河北资源的群体结构最复杂,趋向多元化,分别被聚到6个组群中;其次是中国黑龙江和亚洲资源,分别被聚到4个组群中。这些结果很好地印证了UPGMA聚类结果,均于各群体的地理分布有一定的相关性。
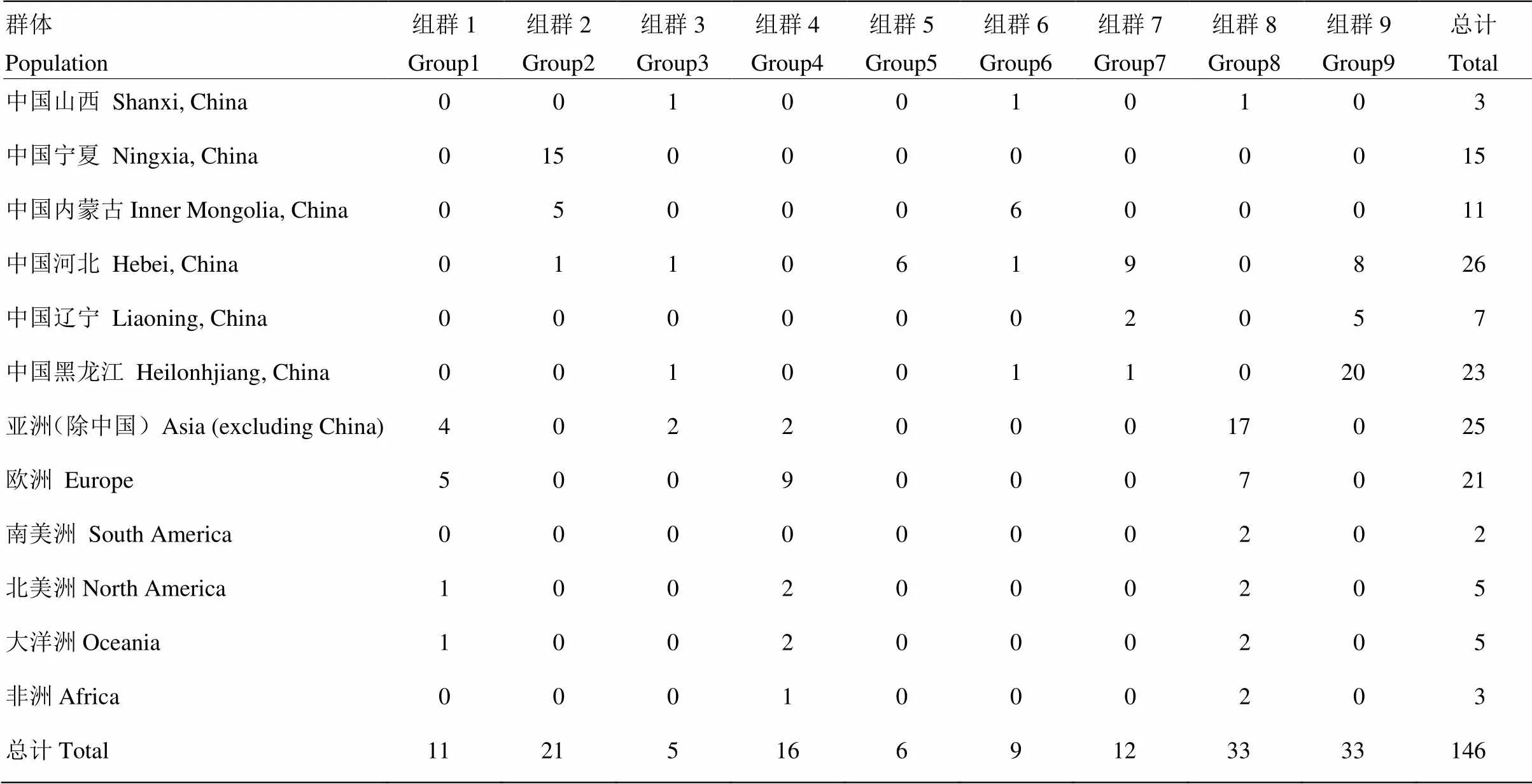
表5 12个群体的遗传结构分析

图2 基于遗传距离的12个群体黍稷资源聚类图
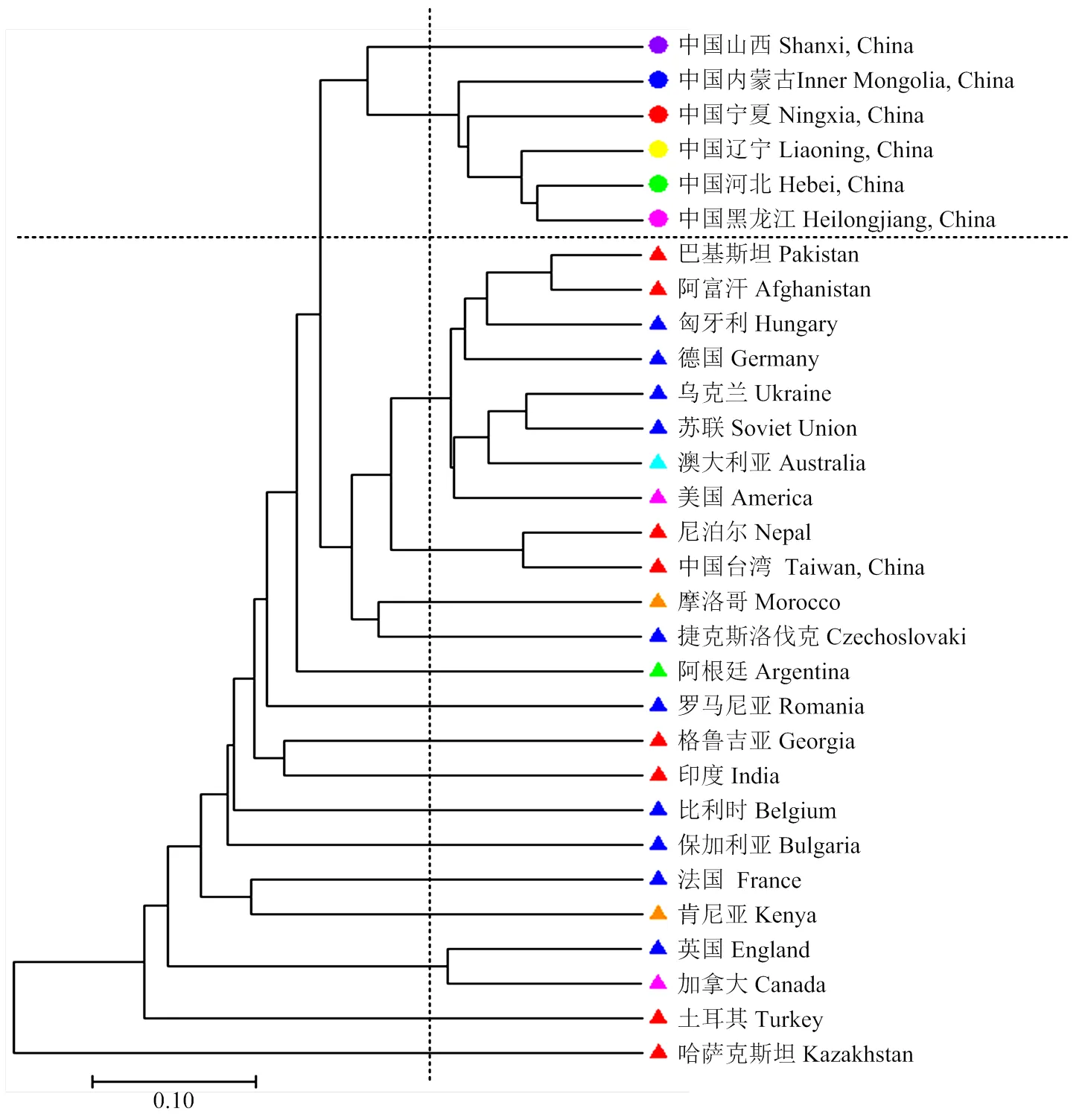
图3 基于遗传距离的30个群体黍稷资源聚类图

中国山西 Shanxi, China; 中国宁夏Ningxia, China; 中国内蒙古,Inner Mongolia, China; 中国河北 Hebei, China; 中国辽宁 Liaoning, China; 中国黑龙江 Heilongjiang, China; 亚洲Asia; 欧洲Europe; 南美洲South America; 北美洲North America; 大洋洲 Oceania; 非洲 Africa
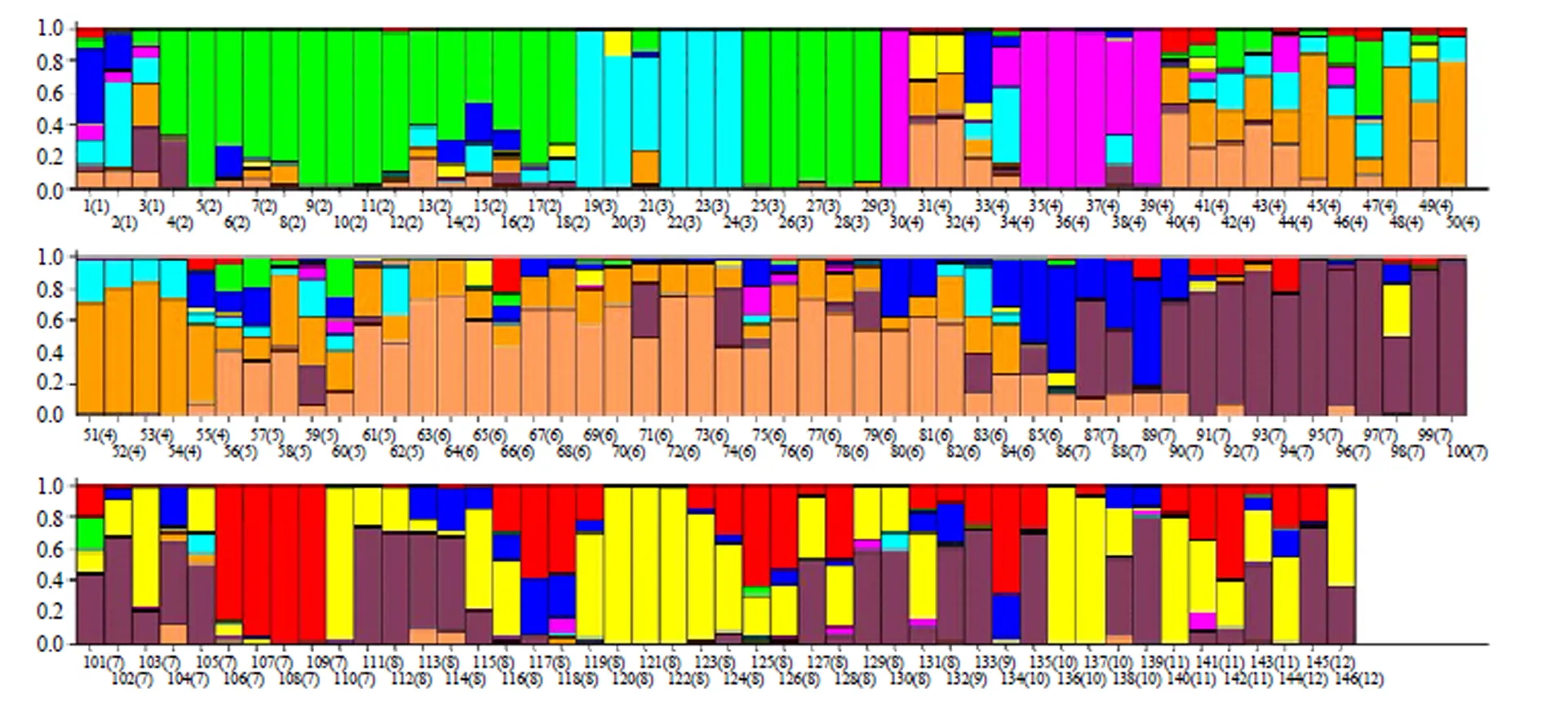
图5 146份黍稷种质资源群体遗传结构图
3 讨论
3.1 黍稷SSR标记的遗传多样性
SSR标记是分析遗传多样的有效标记,已被广泛应用于多种作物的遗传多样性研究,如玉米[11]、小麦[12]、燕麦[13]、水稻[26]等。近年来,不少报道中开发了黍稷特异性SSR标记,并分析了不同黍稷资源的遗传多样性。Cho等[14]开发了25对多态性SSR标记,共检测到110个等位基因,平均每个位点4.4个。连帅等[25]采用63对SSR标记对192份黍稷地方品种和野生品种进行遗传多样性分析,共扩增出161个等位基因,平均每个位点2.56个,值为0.0877—0.8020,平均0.4855。Hou等[27]采用14个多态性SSR标记检测到43个等位基因,平均每个位点3个。王瑞云等[24]利用15个糜子特异性荧光SSR标记分析了132份国内黍稷资源的遗传多样性,检测到107个等位变异,平均每个位点变异数为7个,值范围为0.0893—0.8538,平均0.4864。本研究中利用103对SSR标记对146份黍稷资源进行遗传多样性分析,扩增出308个等位基因,平均每个位点2.99个,值为0.064—0.8035,平均0.5544,Rp值为0.334—4.002,77.67%的标记具有适度分辨力。等位基因数低于王瑞云等[24]的7个,Cho等[14]的4.4个,高于连帅等[25]的2.56个,与Hou等[27]的3个相当,这可能与引物筛选标准不同有关。平均值大于0.5,高于连帅等[25]和王瑞云等[24]的报道,表明所选引物为高度多态性信息引物,可用于黍稷遗传多样性和起源进化的进一步研究。
3.2 参试材料的遗传多样性以及与地理分布的关系
本研究中MEGA结果显示,个体间聚类和群体间聚类均将国内外群体分为两大组群,国内群体中,河北、黑龙江、辽宁资源亲缘关系较近,各遗传参数也表明这些地区黍稷资源的遗传多样性丰富。国外群体中亚洲、欧洲遗传距离最小,遗传一致度最大,亲缘关系最近。中国山西群体与南美洲群体的遗传距离最大,两者均独立分为一支,与其他材料遗传差异较大,且两者地理分布相距远,环境气候有很大差异,表明聚类分析与地理分布相关。STRUCTURE分析结果显示国内外群体间存在明显的基因型分型,国内外群体间地理位置相距较远,地理距离的隔离对各群体的基因流具有一定阻碍。此外还发现中国山西资源与国外亚洲资源存在部分遗传混杂,表明两者亲缘关系较近,且中国山西与亚洲其他国家地理相距较近,暗示中国山西与亚洲其他国家的黍稷资源间可能存在基因流。黍稷群体间可能存在遗传距离与地理距离的相关性,该研究结果与前人[10,21,23-24]报道相似。本研究聚类分析和遗传结构分析结果均表明国外群体遗传分化不明显,没有大的聚类,个体间存在相互渗透,这与连帅等[25]研究结果相似,这可能与国外黍稷资源遗传背景比较狭窄有关,因此,国外黍稷在育种上需加强种质资源的引进与创新。
3.3 黍稷起源进化研究现状
黍稷是干旱半干旱地区的主要作物之一,具有重要的经济价值。研究黍稷野生资源是开展黍稷起源进化研究的重要根据之一。自1753年,提出黍稷原生于印度起[29],国内外学者对黍稷的分类和进化进行了一系列的研究。目前,中国是世界上黍稷考古发现最早、资料最丰富的国家,以黄河中下游为中心,西至新疆,东至黑龙江,多处发现黍稷的遗迹,是公认的黍稷起源中心[28-30]。高俊山等[31]对中国620份栽培黍稷品种酯酶同工酶谱类型地理分布的研究表明中国的栽培黍稷起源于黄土高原。Hu等[21]和王星玉等[32]也得出了相同的结论。中国黍稷专家魏仰浩将野黍定位为黍稷的亚种[29-30]。通过从酯酶同工酶谱和生态性状分析栽培黍稷与野生黍稷的关系,表明栽培黍稷是以具有各种原始性状的野生黍稷及其近缘种进化而来[29-31]。本研究利用SSR标记,从DNA分子水平分析了黍稷野生资源的遗传多样性,结果显示黍稷野生资源中中国河北群体的遗传多样性最丰富,其群体结构趋向多元化,与黑龙江、辽宁群体遗传关系较近,与内蒙古、山西资源个体间存在相互渗透。与栽培黍稷相比较,野生黍稷的遗传多样性更为丰富,在很大程度上保持着原先的遗传状态。一方面可能是因为来自于河北的参试材料较多;另一方面,河北一带野生糜子分布广泛,目前栽培黍稷种植区域也较大,因此,推测河北也有可能是栽培黍稷的初级或次级起源中心,关于黍稷起源中心尚需丰富野生资源进一步研究确定。
4 结论
国内材料野生资源的遗传多样性高于国外材料,其中河北、黑龙江的遗传多样性最丰富,河北可能是黍稷的起源中心。聚类结果与遗传结构分析结果相似,均与地理分布有一定的相关性。
附表1 参试材料
Table S1 Broomcorn millet varieties and landraces used in this study

序号Number群体编号与名称Group number and name材料编号Material number材料来源Material source 11中国山西 Shanxi, China大同-1中国山西大同 Datong Shanxi, China 2中国山西 Shanxi, China大同-2中国山西大同 Datong Shanxi, China 3中国山西 Shanxi, China山西岗县野糜子中国山西岗县 Gangxian Shanxi, China 42中国宁夏 Ningxia, China宁夏-1中国宁夏 Ningxia, China 5中国宁夏 Ningxia, China宁夏-2中国宁夏 Ningxia, China 6中国宁夏 Ningxia, China宁夏-3中国宁夏 Ningxia, China 7中国宁夏 Ningxia, China宁夏-4中国宁夏 Ningxia, China 8中国宁夏 Ningxia, China宁夏-5中国宁夏 Ningxia, China 9中国宁夏 Ningxia, China宁夏-6中国宁夏 Ningxia, China 10中国宁夏 Ningxia, China宁夏-7中国宁夏 Ningxia, China 11中国宁夏 Ningxia, China宁夏-8中国宁夏 Ningxia, China 12中国宁夏 Ningxia, China宁夏-9中国宁夏 Ningxia, China 13中国宁夏 Ningxia, China宁夏-10中国宁夏 Ningxia, China 14中国宁夏 Ningxia, China宁夏-11中国宁夏 Ningxia, China 15中国宁夏 Ningxia, China宁夏-12中国宁夏 Ningxia, China 16中国宁夏 Ningxia, China宁夏-13中国宁夏 Ningxia, China 17中国宁夏 Ningxia, China宁夏-14中国宁夏 Ningxia, China 18中国宁夏 Ningxia, China宁夏-15中国宁夏 Ningxia, China 193 中国内蒙古 Inner Mongolia, China赤峰-2中国内蒙古赤峰 Chifeng Inner Mongolia, China 20中国内蒙古 Inner Mongolia, China赤峰-4中国内蒙古赤峰 Chifeng Inner Mongolia, China 21中国内蒙古 Inner Mongolia, China赤峰-5中国内蒙古赤峰 Chifeng Inner Mongolia, China 22中国内蒙古 Inner Mongolia, China赤峰-6中国内蒙古赤峰 Chifeng Inner Mongolia, China 23中国内蒙古 Inner Mongolia, China赤峰-7中国内蒙古赤峰 Chifeng Inner Mongolia, China 24中国内蒙古 Inner Mongolia, China赤峰-8中国内蒙古赤峰 Chifeng Inner Mongolia, China 25中国内蒙古 Inner Mongolia, China内蒙-2中国内蒙古 Inner Mongolia, China 26中国内蒙古 Inner Mongolia, China内蒙-3中国内蒙古 Inner Mongolia, China 27中国内蒙古 Inner Mongolia, China内蒙-4中国内蒙古 Inner Mongolia, China 28中国内蒙古 Inner Mongolia, China内蒙-5中国内蒙古 Inner Mongolia, China 29中国内蒙古 Inner Mongolia, China内蒙-6中国内蒙古 Inner Mongolia, China 304 中国河北 Hebei, China承德-2中国河北承德 Chengde Hebei, China 31中国河北 Hebei, China承德-3中国河北承德 Chengde Hebei, China 32中国河北 Hebei, China承德-5中国河北承德 Chengde Hebei, China 33中国河北 Hebei, China承德-A中国河北承德 Chengde Hebei, China 34中国河北 Hebei, China承德-6中国河北承德 Chengde Hebei, China 35中国河北 Hebei, China承德-7中国河北承德 Chengde Hebei, China 36中国河北 Hebei, China承德-8中国河北承德 Chengde Hebei, China 37中国河北 Hebei, China承德-9中国河北承德 Chengde Hebei, China 38中国河北 Hebei, China承德B中国河北承德 Chengde Hebei, China 39中国河北 Hebei, China承德-10中国河北承德 Chengde Hebei, China 40中国河北 Hebei, China承德-11中国河北承德 Chengde Hebei, China 41中国河北 Hebei, China承德C中国河北承德 Chengde Hebei, China 42中国河北 Hebei, China承德-12中国河北承德 Chengde Hebei, China 43中国河北 Hebei, China承德D中国河北承德 Chengde Hebei, China 44中国河北 Hebei, China河北-4中国河北 Hebei, China 45中国河北 Hebei, China河北-5中国河北 Hebei, China 46中国河北 Hebei, China河北-6中国河北 Hebei, China 47中国河北 Hebei, China河北-9中国河北 Hebei, China 48中国河北 Hebei, China河北-10中国河北 Hebei, China 49中国河北 Hebei, China河北-14中国河北 Hebei, China 50中国河北 Hebei, China河北-15中国河北 Hebei, China 51中国河北 Hebei, China河北-16中国河北 Hebei, China 52中国河北 Hebei, China河北-17中国河北 Hebei, China 53中国河北 Hebei, China河北-19中国河北 Hebei, China 54中国河北 Hebei, China河北-20中国河北 Hebei, China 55中国河北 Hebei, China河北-7中国河北 Hebei, China 565 中国辽宁 Liaoning, China辽宁-1中国辽宁 Liaoning, China 57中国辽宁 Liaoning, China辽宁-2中国辽宁 Liaoning, China 58中国辽宁 Liaoning, China辽宁-3中国辽宁 Liaoning, China 59中国辽宁 Liaoning, China辽宁-6中国辽宁 Liaoning, China 60中国辽宁 Liaoning, China辽宁-7中国辽宁 Liaoning, China 61中国辽宁 Liaoning, China辽宁-8中国辽宁 Liaoning, China 62中国辽宁 Liaoning, China辽宁-9中国辽宁 Liaoning, China 636 中国黑龙江 Heilongjiang, China齐齐哈尔野-1中国黑龙江齐齐哈尔 Qiqihar Heilongjiang, China 64中国黑龙江 Heilongjiang, China齐齐哈尔野-2中国黑龙江齐齐哈尔 Qiqihar Heilongjiang, China 65中国黑龙江 Heilongjiang, China齐齐哈尔野-3中国黑龙江齐齐哈尔 Qiqihar Heilongjiang, China 66中国黑龙江 Heilongjiang, China齐齐哈尔野-4中国黑龙江齐齐哈尔 Qiqihar Heilongjiang, China 67中国黑龙江 Heilongjiang, China齐齐哈尔野-5中国黑龙江齐齐哈尔 Qiqihar Heilongjiang, China 68中国黑龙江 Heilongjiang, China齐齐哈尔野-6中国黑龙江齐齐哈尔 Qiqihar Heilongjiang, China 69中国黑龙江 Heilongjiang, China齐齐哈尔野-7中国黑龙江齐齐哈尔 Qiqihar Heilongjiang, China 70中国黑龙江 Heilongjiang, China黑龙江245-1中国黑龙江 Heilongjiang, China 71中国黑龙江 Heilongjiang, China黑龙江245-2中国黑龙江 Heilongjiang, China 72中国黑龙江 Heilongjiang, China黑龙江245-3中国黑龙江 Heilongjiang, China 73中国黑龙江 Heilongjiang, China黑龙江245-4中国黑龙江 Heilongjiang, China 74中国黑龙江 Heilongjiang, China黑龙江245-5中国黑龙江 Heilongjiang, China 75中国黑龙江 Heilongjiang, China黑龙江245-6中国黑龙江 Heilongjiang, China 76中国黑龙江 Heilongjiang, China黑龙江245-7中国黑龙江 Heilongjiang, China 77中国黑龙江 Heilongjiang, China黑龙江246-2中国黑龙江 Heilongjiang, China 78中国黑龙江 Heilongjiang, China黑龙江246-3中国黑龙江 Heilongjiang, China 79中国黑龙江 Heilongjiang, China黑龙江246-4中国黑龙江 Heilongjiang, China 80中国黑龙江 Heilongjiang, China黑龙江246-5中国黑龙江 Heilongjiang, China 81中国黑龙江 Heilongjiang, China黑龙江246-6中国黑龙江 Heilongjiang, China 82中国黑龙江 Heilongjiang, China黑龙江246-8中国黑龙江 Heilongjiang, China 83中国黑龙江 Heilongjiang, China黑龙江247-1中国黑龙江 Heilongjiang, China 84中国黑龙江 Heilongjiang, China黑龙江247-2中国黑龙江 Heilongjiang, China 85中国黑龙江 Heilongjiang, China黑龙江248-1中国黑龙江 Heilongjiang, China 867 亚洲 AsiaAMES32315格鲁吉亚 Georgia 87亚洲 AsiaAMES32316格鲁吉亚 Georgia 88亚洲 AsiaPI163298印度 India 89亚洲 AsiaPI163300印度 India 90亚洲 AsiaPI170592土耳其 Turkey 91亚洲 AsiaPI207501阿富汗 Afghanistan 92亚洲 AsiaPI211058阿富汗 Afghanistan 93亚洲 AsiaPI211059阿富汗 Afghanistan 94亚洲 AsiaPI211060阿富汗 Afghanistan 95亚洲 AsiaPI212108阿富汗 Afghanistan 96亚洲 AsiaPI202317阿富汗 Afghanistan 97亚洲 AsiaPI253953阿富汗 Afghanistan 98亚洲 AsiaPI253955阿富汗 Afghanistan 99亚洲 AsiaPI649381阿富汗 Afghanistan 100亚洲 AsiaPI269955巴基斯坦 Pakistan 101亚洲 AsiaPI269957巴基斯坦 Pakistan 102亚洲 AsiaPI269958巴基斯坦 Pakistan 103亚洲 AsiaPI269959巴基斯坦 Pakistan 104亚洲 AsiaPI346939哈萨克斯坦 Kazakhstan 105亚洲 AsiaPI427247尼泊尔 Nepal 106亚洲 AsiaPI427247尼泊尔 Nepal 107亚洲 AsiaPI427248尼泊尔 Nepal 108亚洲 AsiaPI427249尼泊尔 Nepal 109亚洲 AsiaPI427250尼泊尔 Nepal 110亚洲 AsiaPI433381中国台湾 Taiwan, China 1118 欧洲 EuropePI649372法国 France 112欧洲 EuropePI477123德国 Germany 113欧洲 EuropePI209790德国 Germany 114欧洲 EuropePI531407德国 Germany 115欧洲 EuropePI232929匈牙利 Hungary 116欧洲 EuropePI289320匈牙利 Hungary 117欧洲 EuropePI289321匈牙利 Hungary 118欧洲 EuropePI289324匈牙利 Hungary 119欧洲 EuropePI290726英国 England 120欧洲 EuropePI346934乌克兰 Ukraine 121欧洲 EuropePI346941乌克兰 Ukraine 122欧洲 EuropePI346942乌克兰 Ukraine 123欧洲 EuropePI346933苏联 Soviet Union 124欧洲 EuropePI346935苏联 Soviet Union 125欧洲 EuropePI346944苏联 Soviet Union 126欧洲 EuropePI346945苏联 Soviet Union 127欧洲 EuropePI442533比利时 Belgium 128欧洲 EuropePI516181罗马尼亚 Romania 129欧洲 EuropePI531399保加利亚 Bulgaria 130欧洲 EuropePI531402捷克斯洛伐克 Czechoslovakia 131欧洲 EuropePI531406捷克斯洛伐克 Czechoslovakia 1329 南美洲 South AmericaPI202294阿根廷 Argentina 133南美洲 South AmericaPI202295阿根廷 Argentina 13410 北美洲 North AmericaPI296376加拿大 Canada 135北美洲 North AmericaPI677103美国 America 136北美洲 North AmericaPI649385美国 America 137北美洲 North AmericaPI649384美国 America 138北美洲 North AmericaPI649383美国 America 13911 大洋洲 OceaniaPI365040澳大利亚 Australia 140大洋洲 OceaniaPI365842澳大利亚 Australia 141大洋洲 OceaniaPI365845澳大利亚 Australia 142大洋洲 OceaniaPI367683澳大利亚 Australia 143大洋洲 OceaniaPI367684澳大利亚 Australia 14412 非洲 AfricaPI517016摩洛哥 Morocco 145非洲 AfricaPI517017摩洛哥 Morocco 146非洲 AfricaPI531419肯尼亚 Kenya
附表2 所用SSR引物的序列及退火温度
Supplement table 2 Sequence of SSR primers and annealing temperature

位点 Locus正向引物 Forward primer (5'-3')反向引物 Reverse primer (5'-3')退火温度 Tm (℃) BM5CCCTTCTTCCCTACTTTGCCGTGTGCGTGCATGGGTGT55 BM114ATCGTAGAAACCATTGGCCCTGACCCATGGACACTTTTCA55 BM136AATGTCACAGGTTTCCCTCGGCGAGAAAGAGGAGAGGGTT55 BM289TGGGACAATATGGCAAGGTTACAAATGCCTGATGGTAGGC55 BM295CACACAGATATTTGGCACCGTGAGGATCCGAAAAGATTGG55 BM306ATTTTCTGGGCAATTCAACGGTCCTCATCCCTTCCCTCTC55 BM344AGCACTGAGGCACAATTCCTGTGCTGGGGTTTGTGACTTT55 BM374CTACCGCTTCAAAACGAAGGTGTCCCACTCTCCTACCTACTACC55 BM378ATGGGATGCACAGGTACACATCCTTAGGTCATCGTCCTATTTG55 BM396TTGATTATGCTTTGGAGGGGCCTCTCCTTACACGGGGATT55 BM484GGACAGGATGAGGAGGATCACGGTTACCATCGCCTTCTTA55 BM552GCAGCAATAGATTTGCCTCCTCTAGCCTACCCGAACTTGC55 BM637CACACTTGTGCTGTTGGGTCTCTAGCCTACCCCAACTTGC55 BM787TCCGTGACATGATGCAGACTGCACTATTTCTGCAGGAGCC55 BM994AACAGAACCGCACATCAGTGTCAGGTGAGCGATTCTTCCT55 BM1303TTGACGCGTACAACAAGAGCACCGAAGAGAAGGTTGCTGA55 BM1332CGGTTCAGTATTCAGGGCATCACTACACGTACCCGGTCCT55 BM1419CGGTTCAGTATTCAGGGCATCACAAGGATCAGGCCAAAGT55 BM1477GCGAAAGATGAACTGCCAATGCTGCGACGGATACTGATCT55 BM1533CTCTTGTCGTCTTGGTCCGTCGTGCGTGTGTCGAGAGAG55 BM1745CAGCTGATCATTTGAAAAGTATGGTAGTTGTCGCGATCATGAGG55 BM4724TGCATGAAGAAGTGCAGGTGATGATGCCCAAGAGATCGAC55 BM4739GGATGAGAGGAAGGGGTTGTCTTCCCTCCAAACTTCCTCC55 BM4741GTCTGCGTACCTCGGAAGTCTTCCCTCCAAACTTCCTCCT55 BM4749TCTGCGTACCTCGGAAGTCTTTCCCTCCAAACTTCCTCCT55 BM4761GATCACAAAGGGGAACAGGAACATGAGCCAGCTCTCCACT55 BM4830GTTGAAGGCAGACACCTTCCCTCCTCCCTCTCCCTCCC55 BM4846TCTAGCCTACCCCAACTTGCACAAAACCGCTGATCTGCTT55 BM4847GGACGAGTCGGTGAAGAGAGAATTTCTGTAGCGGTGTCGC55 BM4851TTCAATCCAATCCAAGGAGCAACCTTGAAGACATGAGCTGG55 BM4858CCCAACTTGCTTGGGATAAAACCTGATGGCATATTGCTCC55 BM4871CCAGTCGGTGAAGAGAGACCCTCTGTACTCGAGGGCGGT55 BM4877CGCTCCTTTCCTTTCTGTTGAGGAGCCTACTGCTGGAACA55 BM4882TGGGTGTCAGCTTGACTCAGTGCTTACATCCACTGCTTGC55 BM4947GATTTGGGACGAGTCGGTTAAAAGAATTTTCGGGCGACTT55 BM4954GGCGGATCGATGATTGTAGTTAAACGAGGGCAGAAAGGAA55 BM4956GGCTGTAAATCCGCTCGTAGTCCCTCTAATGCAGCCAGTT55 BM4958TCCATAGCTTCCATAGCATTTTCCCCAAAGGATATGGTTCCAA55 BM4962TCCATTGCTAGCGTTCTCCTTAGCGCCAACATGGTGTAGA55 BM4965CCTCCATCGAGAAACCAACTTGCAGAGACATGCATCACAA55 BM4967CTTCACCCACCACACCTTCTAGAATCTGACACCTGCACCA55 BM4969CTTCACCCACCACACCTTCTGAATCTGACACCTGCACCAA55 BM4973TTATCTTCCTCCGCCTCATCTGCATCACAACTGAAGACGA55 BM4997TTCTATCGCTGTGGTGGTCACAACTGCTTCTACAACGGCA55 BM5037ATTCCACCCTTGACCTCCTTTGCATTGACTGCCTTGAAAC55 BM5038GGTCTCTTAAGGAGGGCTGGTGCATTGACTGCCTTGAAAC55 BM5043TGACGAGATCATCTGGCTTGGAAAAGACAGGAGCGCAAAC55 BM5181CCTCACGGTGCATCAGAGTCCAGAGAGCTAAGATATAGGGAATC55 BM5190ACGATCACGAATAAATCCGCTTGAGATGATTCCTGTGTGTGTC55 BM5191GCCCGATAGCCAACGTAATAATGTTTTGAAGGCCCTATGC55 BM5197CTCTGCAACACTGCCACCTATTTACACCGTCGAATTTTATGA55 BM5198CGGAAATAAAGCAACCATGCATTGTGTGCTAGGCCACCTC55 BM5199TCACGTCCCTACACTTCACGGAGGTCGACAGGAAATGCTC55 BM5200CGGAAATAAAGCAACCATGCGACTGACGTAGGGCGTTCTC55 BM5222TGGAGTGTGTGTGTATGTGTGTGCTTGTAGGGCTTGTGGCAAT55 BM5233CTGCTAGAAAAGGACGGGTGCTAGCTAGCCAGGCCCATC55 BM5236CATGCCTCATCCCCTTATTCCGAAACGGCAAACACAAATA55 BM5255GTAAATTCAGATGCTTGCCGCCATCCGAGTTCAAGTTGGT55 BM5259CATACTTCCACACGTGCCTGGGACTATTCTTTGCTACTCCGGT55 BM5260GCGTGCGATAATCATTCGTGCCTACAGAAATATCAGCTGCG55 BM5278CGCCGTTTCATTTTTCTTTCTCACACACACACTACACTCACACTC55 BM5279CGCCGTTTCATTTTCTTTCTCTCGCTCACACACACACTCA55 BM5292GCATGCATGTGTGTGTGTGTAACATATGTGGACGGTTGGC55 LMX258GTGTCTCTTTCGTCTTGCCCGGGACACTTCCACCATCATC55 LMX503GTGGTACAGCTGCTCGTTCAAGGAGGAACCAGGAAGCAAT55 LMX515CGTTTTCTCGCTACACACGATGGACAACGGAAAACGTACA55 LMX621ATGAATCACCCGATCCACATACGCCAACATCAGCATATCA55 LMX632GCTGTCGGTCAGTCCTGTTTACGCCAACATCAGCATATCA55 LMX635GCACACGCATCATCACAAGTGCTCATTCAACGACAGATGC55 LMX645GCTAGGTGTCGTTTTCTCGCGGGGCGTCTAAGCTTGATATT55 LMX653CGATGAACGAAAATTCACCCGTTCATTCGTCCAAATGCCT55 LMX684GGCCTCCTGTGGTCTTCTCTGACCCTCTCCTCCTACCACC55 LMX786CCTGGACACACACACACACATCTTGTCACTGTCGGCGTAG55 LMX691ACTCATGGTTACGGCAACTGGCGCGAGAGAGAGAGAGAGA55 LMX836GCGCAGTAATATATTTCAGTAATTCAGCATCATCGTCAAGACCTCA55 LMX1067ATCGACGACTAGGCCCTGTATGCGGAGTGTCTTTGTTCTG55 LMX1080ATCGACGACTAGGCCCTGTAGGCCGTCACTATATCTGTCACC55 LMX1220AGTTACTTTTCCACACCCGCTTTATTGGGAGTTCGGATGG55 LMX1251GCAGTTTTGTTTCGATCGGTTGGTGTCAAGACATGTGGATG55 LMX1380GCCTCCTGTCTTGTAGCGTCAGGGTAGGCTGAGAGCCTGT55 LMX1387TTTCAGGGACTGGACTGGACGTAGGGGGTAGCTGAGAGCC55 LMX1400AGCAACGGAGGTGAGAGAGATCGACACACACGACACACAC55 LMX1429AATATCCCTTTTGTCGCACGATGCATTGATGGGCTTGATT55 LMX1625ACCCAACCGTATATCCAACGTGTCACAGTTGTCCTGGCAT55 LMX1672ATTTGACCTGTGACCTCGCTCCTTTCTGTTTCTGCAAGCC55 LMX1760TGTGGGAGAGAAGTGGGCCAAGGAAGGAATAAACCGCA55 LMX1749CTCAACACCACACCTTCCCTCTTTTGATCCATGGGCGTAG55 LMX1761GAGATGGTGCGGATTCTGAGTCATTTCCACTGTCACTGCC55 LMX1959GTGGTGAGAAGAAGCCTTGCGATGGGTCGATCAGCAGAAT55 LMX2175GTGACCGACGACTACCGTTTCACCCCTTTCCTCTCGCTAT55 LMX2410CTCAACACCACACCTTCCCTCTTTTGATCCATGGGCGTAG55 LMX1940GCAGTGGGTCAGCTTATGGTTCTCTCTGTGTGTGTGCGTG55 LMX2019CCTCTCCTTACACGGGGATTTTGATTATGCTTTGGAGGGG55 LMX2068TGAGATTGGCATCAAGCAAGTTTCTGGTCAGTTCGGTCAG55 LMX2074GCCACACTAAATAAGCTTTGTGTCTGGTCGTCACTGATTACGGA55 LMX2185GAGTTAGAGGACAGCGTGGCTGCAGCAGAGAATGTGCTACT55 LMX2281TTCTCCGTCAGCTCACATTGTCCATTGTTCATTTAGTAGAAACCT55 LMX2305CGCACTAGCCCTTGTCTTTCCGCCCTACGAACAAATCACT55 LMX2370CCAAGTCCCAAGGTTTTTCAGAGAGAGAGAGGCGTGTGCT55 LMX2382CAACAAGGTTGGTTGGCTTTATGCTGCTGCAGATGTTTTG55 LMX2540TATTCGAGCCCCATTTCTTGGCGTTATCCGGATGATGAAG55 LMX2734CACACAGATATTTGGCACCGTGAGGATCCGAAAAGATTGG55 LMX2782GCCGGAGTATAGATCCGACAGTCAGGCCGTGAACGTTATT55
[1] WANG R Y, WANG H G, LIU X Y, LIAN S, CHEN L, QIAO Z J, MCINERNEY C E, WANG L. Drought-induced transcription of resistant and sensitive common millet varieties.2017, 27(4): 1303-1314.
[2] 王瑞云, 杨阳, 王海岗, 陈凌, 王纶, 陆平, 刘敏轩, 乔治军. 糜子PmNCED1的克隆及其对 PEG 胁迫的响应. 核农学报, 2018, 32(2): 244-256.
WANG R Y, YANG Y, WANG H G, CHEN L, WANG L, LU P, LIU M X, QIAO Z J. Cloning of PmNCED1 and its response to PEG stress., 2018, 32(2): 244-256. (in Chinese)
[3] 王星玉, 王纶. 黍稷种质资源描述规范和黍稷标准. 北京: 中国农业出版社, 2006: 5-15.
WANG X Y, WANG L.. Beijing: China Agriculture Press, 2006: 5-15. (in Chinese)
[4] 王璐琳, 王瑞云, 何杰丽, 薛延桃, 陈凌, 王海岗, 乔治军. 糜子特异性SSR标记的开发. 山西农业科学, 2018, 46(1):1-4,86
WANG L L, WANG R Y, HE J L, XUE Y T, CHEN L, WANG H G, QIAO Z J. Development of broomcorn millet specific SSR marker., 2018, 2018, 46(1):1-4,86. ( in Chinese)
[5] WANG R Y, HUNT H V, QIAO Z J, Wang L, HAN Y H. Diversity and cultivation of broomcorn millet (L.) in China: A review., 2016, 70(3): 332-342.
[6] WANG R Y, WANG H G, LIU X H, JI X, CHEN L, LU P, LIU M X, TENG B, QIAO Z J. Waxy allelic diversity in common millet (L.) in China. The Crop Journal, 2018, https://doi.org/10.1016/j.cj.2018.02.004.
[7] 王瑞云. 糜子遗传多样性及进化研究进展. 北京: 中国农业出版社, 2017.
WANG R Y.(L.). Beijing: China Agriculture Press, 2017 (in Chinese)
[8] 胡兴雨, 陆平, 贺建波, 王纶, 王星玉, 张红生, 张宗文, 吴斌. 黍稷农艺性状的主成分分析与聚类分析. 植物遗传资源学报, 2008, 9(4): 492-496.
HU X Y, LU P, HE J B, WANG L, WANG X Y, ZHANG H S, ZHANG Z W, WU B. Principal component analysis and cluster analysis of agronomic traits of broomcorn millet., 2008, 9(4): 492-496. (in Chinese)
[9] 董孔军, 杨天育, 何继红. 甘肃省糜子地方资源农艺性状遗传多样性分析. 河北农业科学, 2012, 16(2): 1-3.
DONG K J, YANG T Y, HE J H. Genetic diversity of agronomic traits in broomcorn millet local resources in Gansu Province., 2012, 16(2): 1-3. (in Chinese)
[10] RAJPUT S G, SANTRA D K. Evaluation of genetic diversity of proso millet germplasm available in the United States using simple-sequence repeat markers., 2016, 56(5): 2401-2409.
[11] LI L J, YANG K C, PAN G T, RONG T Z. Genetic diversity of maize populations developed by two kinds of recurrent selection methods investigated with SSR markers., 2008, 7(9): 1037-1045.
[12] MANIFESTO M M, SCHLATTER A R, HOPP H E, Suárez E Y, Dubcovsky J. Quantitative evaluation of genetic diversity in wheat germplasm using molecular markers., 2001, 41(3): 682-690.
[13] He X Y, Bjørnstad Å. Diversity of North European oat analyzed by SSR, AFLP and DArT markers., 2012, 125(1): 57-70.
[14] CHo Y I, Chung J W, Lee G A, Ma K H, Dixit A, Gwag J G, Park Y J. Development and characterization of twenty-five new polymorphic microsatellite markers in proso millet (L.)., 2010, 32(3): 267-273.
[15] 王银月, 刘敏轩, 陆平, 乔治军, 杨天育, 李海, 崔喜艳. 构建黍稷分子遗传图谱SSR引物的筛选. 作物杂志, 2014(4): 32-38.
Wang Y Y, Liu M X, Lu P, QIAO Z J, Yang T y, Li H, Cui X y. Construction of molecular genetic map of broomcorn millet for SSR primer screening., 2014(4): 32-38. (in Chinese)
[16] Rajput S G, Plyler-Harveson T, Santra D K. Development and characterization of SSR markers in proso millet based on switchgrass genomics., 2014, 5(1): 175.
[17] 王瑞云, 刘笑瑜, 王海岗, 陆平, 刘敏轩, 陈凌, 乔治军. 用高基元微卫星标记分析中国糜子遗传多样性. 中国农业科学, 2017, 50(20): 3848-3859.
Wang R y, Liu X y, Wang H g, Lu P, Liu M x, Chen L, qiao z j. Analysis of genetic diversity of Chinese broomcorn millet by using high elementary microsatellite markers., 2017, 50(20): 3848-3859. (in Chinese)
[18] 朱建楚. 基于PCR标记的糜子遗传多样性分析[D]. 杨凌: 西北农林科技大学, 2005.
Zhu J C. Analysis of genetic diversity of broomcorn millet based on PCR markers[D]. Yangling:Agriculture and Forestry , 2005. (in Chinese)
[19] 董俊丽, 王海岗, 陈凌, 王君杰, 曹晓宁, 王纶, 乔治军. 糜子骨干种质遗传多样性和遗传结构分析. 中国农业科学, 2015, 48(16): 3121-3131.
Dong J l, Wang H g, Chen L, Wang J j, Cao X n, Wang L, qiao z j. Analysis of genetic diversity and genetic structure of broomcorn millet of the Chinese., 2015, 48(16): 3121-3131. (in Chinese)
[20] Hu Y G, Zhu J, Liu F, Zhang Z, Chai Y, Weining S. Genetic diversity among Chinese landraces and cultivars of broomcorn millet () revealed by the polymerase chain reaction., 2008, 153(3): 357-364.
[21] Hu X, Wang J, Lu P, Zhang H. Assessment of genetic diversity in broomcorn millet (L.) using SSR markers., 2009, 36(8): 491-500.
[22] 连帅, 王瑞云, 马跃敏, 刘笑瑜, 季煦. 不同生态区糜子种质资源的遗传多样性分析. 山西农业大学学报: 自然科学版, 2015, 35(3): 225-231.
Lian S, Wang R Y, Ma Y M, Liu X Y, Ji X. Analysis of genetic diversity of broomcorn millet germplasms in different ecotype zone.,2015, 35(3): 225-231. (in Chinese)
[23] Liu M x, Xu Y, He J h, Zhang S, Wang Y y, Lu P. Genetic diversity and population structure of broomcorn millet (L.) cultivars and landraces in china based on microsatellite markers., 2016, 17(3): 370.
[24] 王瑞云, 季煦, 陆平, 刘敏轩, 许月, 王纶, 王海岗, 乔治军. 利用荧光SSR分析中国糜子遗传多样性. 作物学报, 2017, 43(4): 530-548.
Wang R y, Ji x, Lu P, Liu M x, Xu Y, Wang L, Wang H g, qiao z j. Analysis of genetic diversity of Chinese broomcorn millet by fluorescence SSR., 2017, 43(4): 530-548. (in Chinese)
[25] 连帅, 陆平, 乔治军, 张琦, 张茜, 刘敏轩, 王瑞云. 利用SSR分子标记研究国内外黍稷地方品种和野生资源的遗传多样性. 中国农业科学, 2016, 49(17): 3264-3275.
LIAN S, LU P, QIAO Z J, ZHANG Q, ZHANG Q, LIU M X, WANG R Y. Genetic diversity in broomcorn millet (L.) from China and abroad by using SSR markers., 2016, 49(17): 3264-3275. (in Chinese)
[26] THOMSON M J, POLATO N R, PRASETIYONO J,TRIJATMIKO K R, SILITONGA T S, MCCOUCH S R. Genetic diversity of isolated populations of Indonesian landraces of rice (L.) collected in east Kalimantan on the island of Borneo, 2009, 2(1): 80-92.
[27] Hou S Y, Sun Z X, Li Y S, Wang Y, Ling H B, Xing G F, HAN Y H, Li H Y. Transcriptomic analysis, genic SSR development, and genetic diversity of proso millet (; Poaceae)., 2017, 5(7):1-11
[28] 王星玉. 黍稷史话. 种子通讯, 1985, 4: 21.
Wang X y. History of broomcorn millet., 1985, 4: 21. (in Chinese)
[29] 魏仰浩. 试论黍的起源. 农业考古, 1986, 2: 248-251.
Wei Y h. On the Origin of broomcorn millet., 1986, 2: 248-251. (in Chinese)
[30] 董玉琛, 郑殿升. 中国作物及其野生近缘植物: 粮食作物卷. 北京:中国农业出版社, 2006.
Dong Y C, Zheng D s. Crops and Their Wild Relatives in China: Food Crops Rolls. Beijing: China Agriculture Press, 2006. (in Chinese)
[31] 高俊山, 徐新宇, 胡荣海, 魏仰浩. 糜黍酯酶同工酶的研究. 作物品种资源, 1990, 2: 2.
Gao J s, Xu X y, Hu R h, Wei Y h. Studies on esterase isoenzymes of broomcorn millet.s, 1990, 2: 2. (in Chinese)
[32] 王星玉, 王纶, 温琪汾, 师颖. 山西是黍稷的起源和遗传多样性中心. 植物遗传资源学报, 2009, 10(3): 465-470.
Wang X y, Wang L, Wen Q F, Shi Y. Shanxi is the origin and genetic diversity center of broomcorn millet., 2009, 10(3): 465-470. (in Chinese)
(责任编辑 李莉)
Genetic Diversity and genetic relationship of Broomcorn Millet (L.) germplasm based on SSR markers
XUE YanTao1,2, LU Ping2, QIAO ZhiJun3, LIU MinXuan2, WANG RuiYun1,3
(1College of Agriculture, Shanxi Agricultural University, Taigu 030801, Shanxi;2Institute of Crop Science, Chinese Academy of Agricultural Sciences, Beijing 100081;3Institute of Crop Germplasm Resources, Shanxi Academy of Agricultural Sciences/Key Laboratory of Crop Gene Resources and Germplasm Enhancement on Loess Plateau, Ministry of Agriculture/Shanxi Key Laboratory of Genetic Resources and Genetic Improvement of Minor Crops, Taiyuan 030031)
【Objective】The objective of this study is to analyze the genetic diversity and relationship of broomcorn millet landraces and wild materials by SSR markers, to provide available data for further evolutionary study of broomcorn millet.【Method】137 SSR primers are used to identify polymorphisms in six representatives which selected randomly from the total of accessions. A total of 103 primers produce clear and reproducible polymorphic fragments among the six accessions and then are used to amplify 146 broomcorn millet accessions. Genetic diversity and relationship between different individuals and populations is evaluated by analyzing genetic parameter, clustering, and genetic structure.【Result】 103 SSR markers detect a total of 308 alleles (Na) with an average of 2.99 for each SSR and the mean values of Shannon-Weaver index (I), Nei andwere 0.8478, 0.3642 and 0.5544, respectively. Their resolution range was 0.334-4.002 and more than 60% distribution at intervals of 1-4, indicated the moderate resolving power of these SSR. The observed number of alleles (2.9126), Shannon-Weaver index (0.8302), expected heterozygosity (0.5023), andvalue (0.5278) of broomcorn millet accessions in China were all higher than those in abroad, indicated more abundant genetic diversity in Chinese samples. The genetic distance of the 12 populations ranged from 0.0783 to 0.5762 with a mean of 0.2938. The genetic identity ranged from 0.5620 to 0.9247 with a mean of 0.75. We found that the genetic similarity had a certain correlation with geographical distribution. The closer geographical distribution, the smaller genetic distance, the higher genetic identity. Cluster analysis divided 12 populations into 4 groups at a genetic distance of 0.15. Among them, resources in South America and Shanxi were each independently divided into one group, which had a far-distance relationship with other resources. In the inter-individual clustering, the division of resources at home and abroad was very significant. At a genetic distance of 0.63, 146 broomcorn millet accessions could be divided into three groups. Group Ⅰand group Ⅱwere foreign accessions, and group Ⅲwas domestic accessions. Further, groupⅡwas divided into three subpopulations at a genetic distance of 0.39, and group Ⅲwas divided into five subpopulations at a genetic distance of 0.45. There had closer genetic relationship between Asia and European resources, as well as Hebei, Shanxi and Inner Mongolia in China resources than other populations. The result of genetic structure analysis showed that there is obvious genetic differentiation between the domestic populations and foreign populations. Five groups (Group 2, Group 5, Group 6, Group 7 and Group 9) were unique genotypes which owned by Chinese wild resources and distributed more widely, 2 groups (Group 1 and Group 4) were unique genotypes of foreign resources and have a relative narrow distribution. The population structures of Ningxia and South America tend to be independent, and the population structures of Hebei, Heilongjiang and Asia tend to be diversified. The UPGMA clustering results were consistent with the results of genetic structure analysis, and the genetic relationships were related to their geographical distribution.【Conclusion】The genetic diversity of wild accessions is higher than that of landraces, of which Hebei population has the most abundant genetic diversity, so we suppose Hebei province may be the sub-origin center of broomcorn millet.
L; wild broomcorn millet; foreign germplasm; SSR markers; genetic diversity; population structure
2018-05-07;
2018-06-05
国家自然科学基金(31271791)、山西省回国留学人员科研资助项目(2016-066)、国家现代农业产业技术体系建设专项(CARS-06-13.5-A16)、农业部谷子高粱产业体系(CARS-07-12[1].5-A1)、农业部作物种质资源保护项目(NB2012-2130135-25-06-1)
薛延桃,Tel:18234839761;E-mail:yantaoxue305@163.com。通信作者刘敏轩,Tel:010-62159962;E-mail:liuminxuan@caas.cn。通信作者王瑞云,Tel:15234420135;E-mail:wry925@126.com
10.3864/j.issn.0578-1752.2018.15.002

