玉米SRO基因家族的鉴定及表达分析
赵秋芳,马海洋,贾利强,陈曙,陈宏良
玉米SRO基因家族的鉴定及表达分析
赵秋芳,马海洋,贾利强,陈曙,陈宏良
(中国热带农业科学院南亚热带作物研究所/农业部热带果树生物学重点实验室,广东湛江 524091)
【目的】SRO(similar to rcd one)是植物所特有的一类小蛋白家族,其在植物的生长发育,及应对非生物胁迫中发挥着重要功能。基于玉米全基因组数据库,鉴定玉米SRO家族基因,分析其序列、基因定位、蛋白结构及其系统进化关系,同时解析在玉米组织表达特异性及其在高盐和干旱胁迫下的表达变化,为阐明SRO基因在玉米生长和逆境响应中的功能研究奠定基础。【方法】利用拟南芥SRO家族基因为探针,在玉米全基因组查找并下载玉米SRO基因序列,并从MaizeGDB中获取玉米SRO基因相关信息,包括CDS、氨基酸序列及染色体位置等。通过生物信息学工具(GSDS2.0、Expasy-protparam、SOPMA、Plant-mPLoc、EMBL-EBI、MEME)对获得序列的基因结构、蛋白质分子量、等电点、二级结构、亚细胞定位、保守结构域、保守基序原件等进行预测和分析。同时利用Clustalx(1.83)和MEGA 6.0软件进行同源序列比对并构建系统进化树。运用实时荧光定量PCR技术分析玉米组织表达特异性及其在高盐和干旱胁迫下的表达变化情况。【结果】从玉米全基因组共鉴定6个SRO家族基因,分别命名为。分布于第1、4、5和9染色体,包含25个内含子。序列分析发现CDS序列长度在1 2151 791 bp;编码氨基酸数目为404596 aa;分子量为45.2366.78 kD;等电点为7.019.17。亚细胞定位分析发现ZmSRO1a/ZmSRO1b/ZmSRO1c/ZmSRO1d定位于叶绿体,ZmSRO1e则定位于过氧化氢酶体,ZmSRO1f定位于细胞核。系统进化树分析发现ZmSROs分为3个亚类,Ⅰa亚类包括ZmSRO1a/ZmSRO1b/ZmSRO1c,Ⅰb亚类包括ZmSRO1f,Ⅰc亚类包括ZmSRO1d/ZmSRO1e。保守结构域分析结果显示ZmSRO1a/ZmSRO1b/ZmSRO1c/ZmSRO1d/ZmSRO1e包含PARP和RST结构域,缺少WWE结构域,ZmSRO1f包含WWE和PARP催化中心,RST结构域缺失。ZmSROs蛋白共找到5个保守基序,命名为基序15。ZmSRO1a/ZmSRO1b/ZmSRO1c包含所有保守基序,ZmSRO1d/ZmSRO1e缺少保守基序3,ZmSRO1f缺少保守基序5。组织表达分析发现在根系特异性表达。高盐胁迫下,玉米根系中在1 h时显著上调表达,地上部中均下调表达,而在处理6 h显著上调表达。干旱胁迫下,玉米根系在1 h显著上调表达,在24 h显著上调表达;地上部中均下调表达。【结论】玉米SRO家族基因包含6个成员,被划分为3个亚类,6个在玉米根系中特异性表达,且可以不同程度地响应干旱和高盐胁迫。
玉米;SRO基因;非生物胁迫;基因表达
0 引言
【研究意义】玉米(L.)是世界重要的粮食作物之一,在世界粮食总产量中处于第一位,同时也是重要的饲料和工业原料[1]。但实际生产中玉米常常会因受到诸多不利环境因素的影响而大面积减产,比如干旱、盐胁迫和低温等。SRO(similar to rcd one)是植物所特有的一类小蛋白家族,其在植物的生长发育及应对盐、干旱、重金属等非生物胁迫中发挥着重要作用。对玉米SRO家族基因进行全面的生物信息学、组织特异性表达及盐、干旱胁迫条件下表达进行分析,有利于更深入了解SRO家族基因的功能,并对玉米抗逆基因挖掘和抗逆育种具有重要意义。【前人研究进展】SRO是植物所特有的类RCD-ONE蛋白家族,它们通常包含高度保守的PARP(poly (ADP ribose) polymerase catalytic;PS51059)催化中心和C末端的RST(RCD1-SRO-TAF4;PF12174)保守结构域[2]。目前对SRO蛋白家族的研究主要集中在拟南芥中。据报道,在拟南芥中共鉴定出6个SRO同源基因,分别为、[2-3]。AtSRO1与AtRCD1(radical-induced cell death 1)同源性最高,除具有PARR和RST保守结构域外,还具有N端的WWE(PS50918)结构域,属于亚家族A[4],AtSRO2-5仅包含PARP和RST结构域,缺少WWE结构域,属于亚家族B[5]。以往研究表明,PARP-RST结构域是植物所特有的,而WWE-PARP结构域广泛存在于生物体中,且高度保守[6-7]。是植物中首个被鉴定的SRO家族基因,拟南芥中突变体表现出对盐、寒害、紫外线、MV、以及CO毒害和激素(ABA/JA/ETH)的耐受性的增加,同时影响拟南芥的生长,表现为株高减少、早花、连坐叶片、限制根系生长和生殖发育等[1,5,8-14]。同样参与到非生物胁迫的应激反应,但是与之间有些不同,突变体增强盐胁迫下的拟南芥的渗透调节能力,而突变体则表现出对盐胁迫敏感表型[13]。此外,有研究表明可以响应盐胁迫[15],和的转录子也受到O3引起的氧化胁迫诱导[5]。目前SRO家族基因在水稻、小麦、番茄、苹果、陆地棉等作物中有部分研究,其作用和功能也已日益清楚。水稻OsSRO1c蛋白可以与水稻干旱转录因子SNAC1和DST直接作用,通过调节气孔关闭和过氧化氢累积来响应干旱和氧化胁迫[16-17]。小麦在干旱、高盐和H2O2胁迫条件下均上调表达,可以通过调节植物体内的氧化还原平衡来提高植物生长和抵抗逆境胁迫的能力[18]。番茄与序列相似,且在盐胁迫条件下上调表达,特别是在根系中[19]。苹果中鉴定出6个SRO家族基因,其中被认为是重要的非生物胁迫调节因子,可以增强其耐受盐、干旱、及氧化胁迫的能力[20]。吕有军等[21]在陆地棉中鉴定出12个SRO基因,它们的表达存在组织特异性,且明显受冷处理和盐处理的诱导,、、也受到不同程度胁迫处理的诱导。【本研究切入点】随着研究的深入,植物SRO家族基因功能被越来越多的人所认识,其在植物生长,特别是应答各种非生物胁迫中的作用被日益重视,但是该家族基因在玉米中鲜见报道。【拟解决的关键问题】本研究以玉米全基因组数据为基础,鉴定SRO家族成员,分析基因结构、亚细胞定位、蛋白保守结构域及系统进化关系等信息,明确玉米SRO家族基因在各个组织中的表达特异性和胁迫条件下的表达模式,为解析SRO蛋白功能和抗逆调控作用奠定基础,同时也为玉米抗逆基因挖掘和抗逆育种提供理论参考。
1 材料与方法
1.1 试验材料及处理方法
供试玉米材料为B73,玉米不同组织器官(根、茎、叶、雄穗和雌穗)取自B73自交系,采样时期为玉米抽穗期,采样时间为2017年5月23日。取样完成后,液氮速冻,-80℃保存,备用。非生物胁迫于2017年6月8日开始,具体试验如下:选取B73生长饱满的种子,表面消毒杀菌后,28℃催芽2 d,转入1/2 Hoagland培养液中培养7 d后,正常营养液培养至玉米3叶1心,选取生长一致玉米幼苗各12株进行干旱和高盐胁迫处理,每个处理4株,3次重复。干旱胁迫:幼苗置于20% PEG-6000溶液中培养;高盐胁迫:幼苗置于200 mmol·L-1NaCl溶液中培养,并于处理后0、1、6和24 h采集玉米幼苗和根系样品,液氮速冻,-80℃冰箱保存备用。
1.2 玉米SRO基因家族的鉴定及序列分析
以拟南芥全基因组数据库(TAIP)中6个拟南芥SRO蛋白序列为探针,在玉米基因组数据库MaizeGDB(https://www.maizegdb.org/)和Phytozome12.0(https:// phytozome.jgi.doe.gov/pz/portal.html)搜索玉米SRO家族候选成员,并利用Pfam和SMART对候选基因进行进一步确认玉米SRO家族基因,同时获得其CDS、氨基酸序列及染色体位置信息。
1.3 玉米SRO基因的生物信息学分析
运用Expasy protparam tool(http://web.expasy.org/ protparam/)在线工具分析玉米SRO基因家族成员的分子量和等电点;分别运用在线工具plant-mploc(http://www.csbio.sjtu.edu.cn/bioinf/plant-multi/)和SOPMA(https://npsa-prabi.ibcp.fr/cgi-bin/npsa_automat. pl?page=npsa_sopma.html)对SRO进行亚细胞定位和蛋白的二级结构预测。利用GSDS2.0(http://gsds.cbi.pku. edu.cn/)在线工具绘制玉米SRO基因结构图,分析其外显子和内含子结构。利用EMBL-EBI(http://www. ebi.ac.uk/interpro/)分析氨基酸序列的蛋白质保守区,并用IBS1.0软件绘制蛋白保守结构域图。利用MEME(http://meme-suite.org/tools/meme)在线工具对玉米SRO蛋白的保守基序进行预测,参数中预测数目设置为5,长度设置为6—50,其他参数均为默认设置。从水稻基因组获取已知的水稻SRO家族蛋白序列,利用Clustalx(1.83)进行同源序列比对,并利用MEGA6.0软件,采用相邻连接法(Neighbor-Joining,NJ)构建拟南芥、水稻、玉米SRO蛋白家族的系统进化树,校验参数BootStrap重复为1 000次,其他参数均为默认值。
1.4 玉米SRO基因家族表达
采用华越洋植物RNA快速提取试剂盒提取玉米样品总RNA,使用Roche第一链cDNA合成试剂盒将提取的总RNA反转录cDNA。根据玉米SRO基因序列,采用Oligo 7软件设计定量引物,上海生工生物工程有限公司合成,引物序列详见表1。实时荧光定量PCR反应在Roche lightCycler480中进行,试剂盒为Thermo Fisher scientific,反应体系10 μL包括SYBR Green Master MIX(2×),5 μL、上下游引物(10 μmol·L-1)各1 μL、cDNA 1 μL和ddH2O 2 μL。反应条件为50℃ 2 min,95℃ 3 min;95℃ 15 s,58℃ 15 s,72 ℃ 30 s,45个循环,3次重复。采用2-ΔΔCt方法进行数据计算,基因的相对表达量使用(平均值±标准差)表示。

表1 ZmSROs实时荧光定量PCR分析所用引物
2 结果
2.1 玉米SRO基因的鉴定及序列分析
从玉米基因组数据库中共鉴定到6个SRO家族基因,并按照You等[17]方法进行命名,其基因序列信息见表2。6个分别分布于第1、4、5和9染色体,其中和位于第1染色体,位于第4染色体,和位于第5染色体,位于第9染色体。玉米SRO家族基因编码区长度为1 215—1 791 bp;编码氨基酸数目为404—596 aa;分子量为45.23—66.78 kD;等电点为7.01—9.17,均大于7,均为碱性蛋白。亚细胞预测定位4个ZmSROs定位于叶绿体,ZmSRO1f定位于细胞核,ZmSRO1e则定位于过氧化氢酶体中(表2)。
通过对6个ZmSROs蛋白的二级结构预测(表3),结果表明,ZmSROs蛋白的二级结构以α-螺旋和无规则卷曲结构为主,扩展链结构和β-转角结构所占比例较小。其中ZmSRO1a、ZmSRO1b、ZmSRO1c和ZmSRO1f的无规则卷曲结构所占比例最大,为38.76%—46.55%,其次为α-螺旋结构,为28.22%—30.54%;ZmSRO1d和ZmSRO1e的α-螺旋结构所占比例最大,分别为47.10%和43.62%,其次为无规则卷曲结构分别为30.36%和32.25%。
2.2 玉米SRO基因结构、蛋白保守结构域及进化树分析
6个s的基因结构图见图1。均含有5个内含子和6个外显子,和均包含3个内含子和4个外显子,而仅包含2个内含子和3个外显子。
利用EBL-EBI在线分析发现,根据SRO蛋白序列中所包含的保守功能区域,可以将ZmSROs蛋白分为2类,A类为ZmSRO1a/ZmSRO1b/ZmSRO1c/ ZmSRO1d/ZmSRO1e共5个蛋白,均包含PARP催化中心和C-末端的RST结构域,缺少WWE结构域,但是由于蛋白质氨基酸序列长度不同,其保守结构域出现的位置也不相同。ZmSRO1a/ZmSRO1b/ZmSRO1c的PARP和RST结构域分别位于第235—466 aa和第486—572 aa,而ZmSRO1d和ZmSRO1e蛋白的PARP和RST结构域分别位于第147—384 aa和第373—448 aa;B类为ZmSRO1f包含WWE和PARP催化中心,C-末端的RST结构域缺失,其中WWE位于第75—129 aa,PARP催化中心位于第189—404 aa(图2)。

表2 玉米SRO基因家族的序列信息
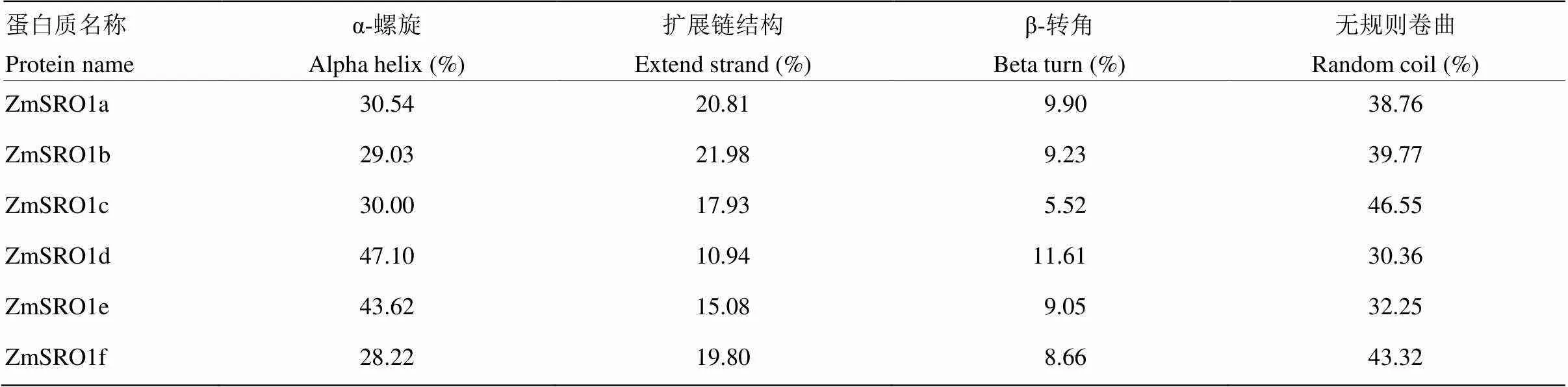
表3 玉米SRO蛋白的二级结构
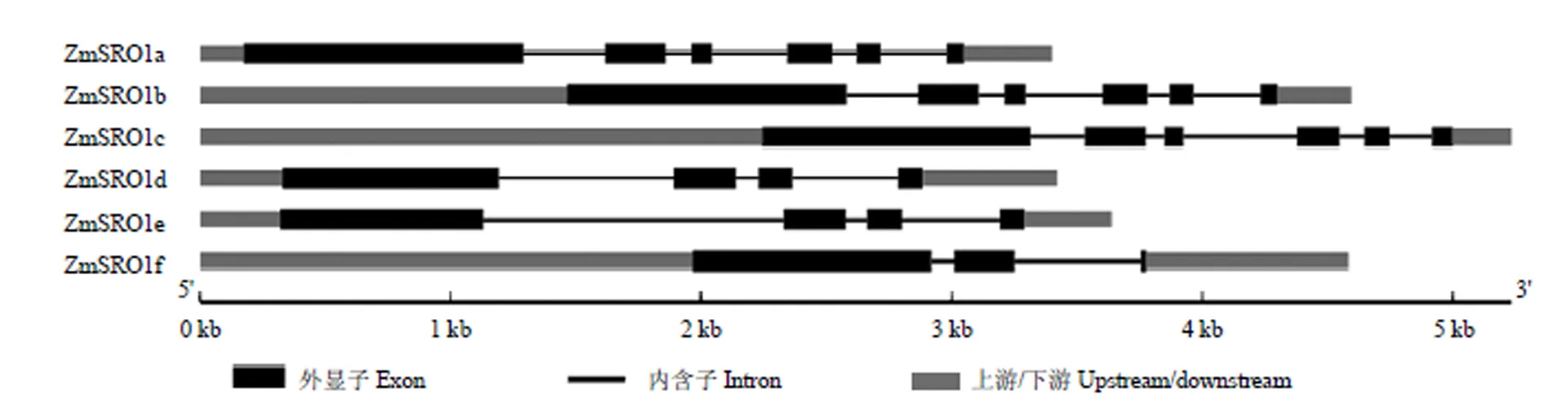
图1 ZmSROs基因结构图
利用MEME在线工具预测ZmSROs蛋白的保守基序,由图3可知,ZmSROs蛋白共找到5个保守基序,ZmSRO1a/ZmSRO1b/ZmSRO1c包含保守基序1—5,ZmSRO1d/ZmSRO1e缺少保守基序3,ZmSRO1f缺少保守基序5。结合ZmSROs蛋白结构域分析可以看出,保守基序1位于PARP催化中心,保守基序5位于RST保守结构域中。
利用MAGA6.0构建玉米、拟南芥、水稻SRO蛋白家族的系统进化树(图4),SRO家族蛋白可以被分为Ⅰ和Ⅱ两类,其中Ⅰ类由可以被分为Ⅰa、Ⅰb、Ⅰc 3个亚类,Ⅱ类可以被分为Ⅱa和Ⅱb 2个亚类。在玉米和水稻中仅存在第Ⅰ亚类,缺少第Ⅱ亚类。按照水稻的亚家族分类,可以将ZmSROs分为3个亚类,ZmSRO1a/ZmSRO1b/ZmSRO1c同源性最高,均属于Ⅰa亚类,ZmSRO1d/e属于Ⅰc亚类,ZmSRO1f属于Ⅰb亚类,玉米和水稻均为禾本科植物亲源关系较近,其SRO蛋白序列同源性非常高,进化关系非常接近。
2.3 玉米SRO基因在不同组织的表达分析
利用qRT-PCR技术分析了6个在玉米根、茎、叶、雄穗和雌穗中的表达(图5),6个在玉米根系相对表达量显著高于其他组织部位,说明6个均在根系中有特异性表达,而在雌穗和雄穗中的表达量较低,几乎不表达。结果表明,可能在玉米根系发育过程中起重要作用。
2.4 玉米SRO基因在逆境胁迫的表达分析
为了明确玉米SRO基因对逆境胁迫下的应答机制,分析了SRO基因在盐胁迫和干旱胁迫下根系和地上部的表达模式(图6和图7)。在根系中,200 mmol·L-1NaCl处理下,除外,其余5个处理1 h时显著上调表达,1 h相对表达量分别为0 h的13.5、17.1、5.4、31.1和27.9倍,随后6和24 h后表达量下降,表明玉米根系中的5个基因均可以迅速响应盐胁迫信号,而对盐胁迫信号没有响应。20%PEG处理下,在1 h表达量为0 h处理的3.6倍,显著上调表达;在24 h的表达量为0 h的2.7倍,显著上调表达,表明玉米根系中和可能响应干旱胁迫。盐胁迫条件下玉米地上部的表达均下调,表达差异不大而在处理6 h显著上调表达,为0 h的3.06倍,说明玉米地上部可能对盐胁迫有响应。干旱胁迫处理下,均下调表达,而和表达差异不显著,变化趋势与盐胁迫处理类似。
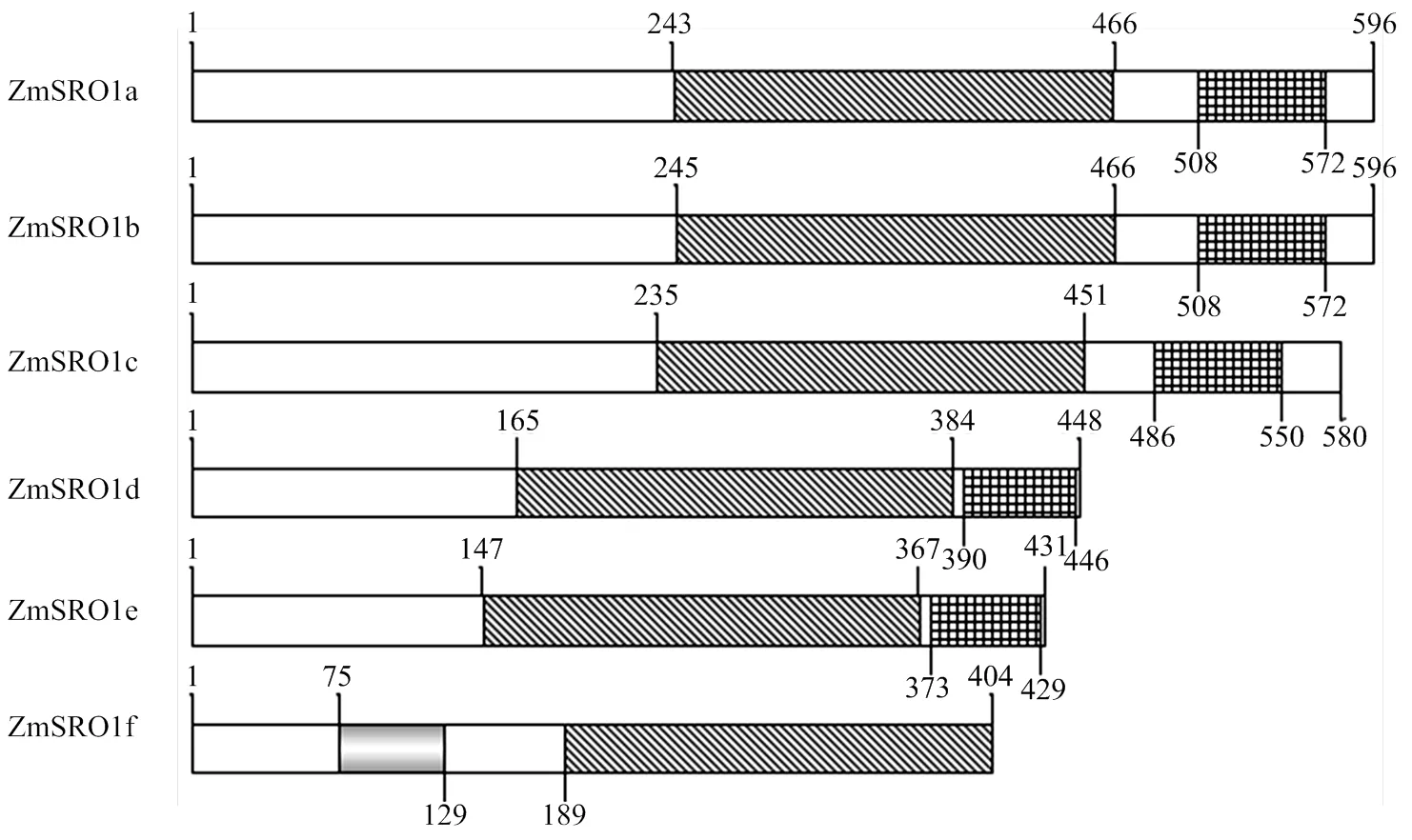
灰色区域、黑色斜纹网格区域和黑色竖纹区域分别表示ZmSROs蛋白的WWE、PARP和RST结构域
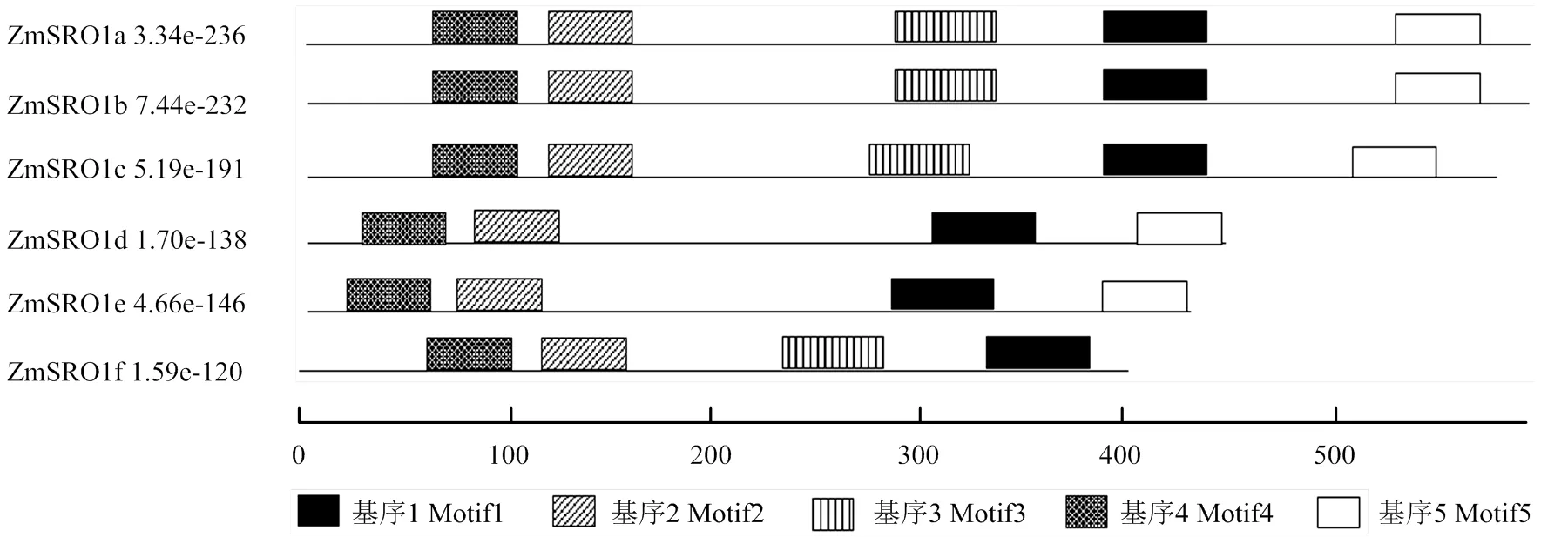
图3 ZmSROs蛋白保守基序分布
3 讨论
由于全球气候变化,极端气候条件频发,以及土壤质量下降和盐渍化的加剧,使得干旱和盐害等非生物胁迫成为影响植物生长和产量的主要限制因子。玉米作为世界重要粮食作物,玉米产业发展也时刻面临着非生物胁迫等逆境环境的危害,因而挖掘重要的抗逆基因,选育抗逆玉米新品种,以提高玉米自身的抗逆能力显得尤为重要。研究发现许多植物特有的转录因子可以影响植物生长发育,并响应外界环境变化[22],如NAC、AP2/ERF、WRKY、Dof和ARF等[23-27]。SRO是最近被发现的陆生植物特有蛋白家族,其在植物生长发育以及响应非生物胁迫方面具有重要作用。目前,拟南芥[2-3]、水稻[16-17]、小麦[18]、番茄[19]、苹果[20]等植物中的SRO家族已被分离鉴定,并对其功能进行了初步研究,但SRO基因家族在玉米中的研究仍鲜见报道。本研究从玉米全基因组中共鉴定出6个,依次命名为,系统进化树分析发现,玉米和水稻SRO蛋白相似性非常高,亲缘关系相近。根据植物SRO蛋白可以被分为2类(Ⅰ和Ⅱ),Ⅰ类被进一步分为Ⅰa、Ⅰb、Ⅰc 3个亚类,系统进化树分析表明AtSRO1、AtRCD1、OsSRO1a/ OsSRO1b和ZmSRO1a/ZmSRO1b/ZmSRO1c属于Ⅰa亚类,OsSRO1d/OsSRO1e和ZmSRO1f属于Ⅰb亚类,OsSRO1c和ZmSRO1d/ZmSRO1e属于Ⅰc亚类,Ⅱ类蛋白也可以被分为Ⅱa和Ⅱb 2个亚类,其中AtSRO2/AtSRO3属于Ⅱa,AtSRO4/AtSRO5属于Ⅱb亚类(图4)。Ⅱ类蛋白是双子叶植物所特有的[17],玉米和水稻中均不存在Ⅱ类的SRO蛋白。根据其包含蛋白家族结构域可将ZmSROs分为2类,A类包括ZmSRO1a/ZmSRO1b/ZmSRO1c/ZmSRO1d/ZmSRO1e,均包含PARP催化中心和C-末端的RST结构域,缺少WWE结构域,而B类仅包括ZmSRO1f,包含PARP催化中心和WWE结构域,缺少C-末端的RST结构域(图2),以往研究表明水稻OsSRO1d中也缺少RST结构域,其原因可能是OsSRO1d的RST结构域中被额外插入18个氨基酸序列,使得在结构域分析时RST结构域未被分析出来[17]。蛋白保守基序预测显示ZmSRO1f缺少保守基序5,结合蛋白保守结构域分析保守基序5恰好位于RST保守结构域内(图4和图5),而ZmSRO1f仅含404个氨基酸,其氨基酸数目较少可能是RST保守结构域缺失的原因。
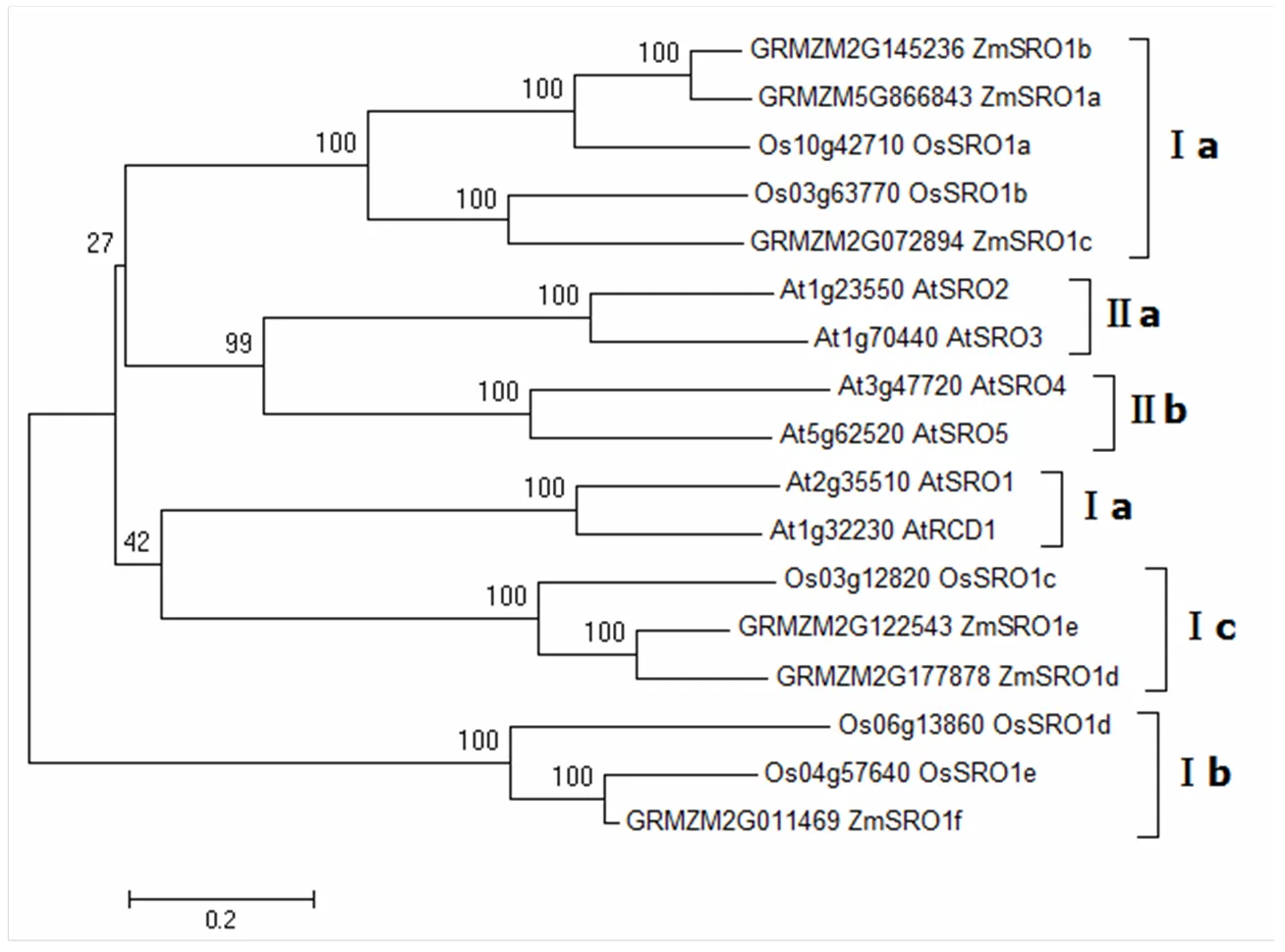
图4 玉米、拟南芥、水稻的SRO蛋白家族的系统进化树
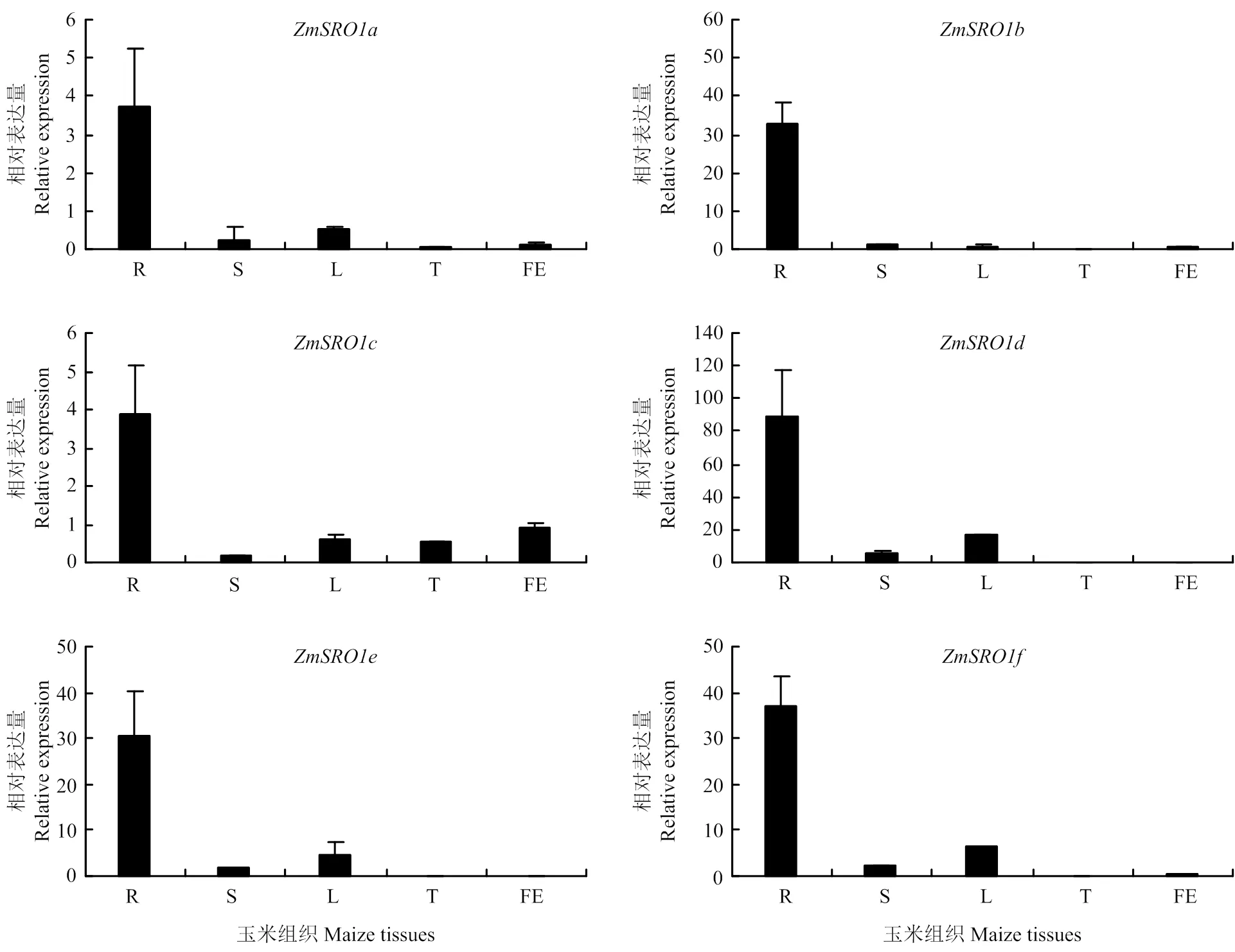
R:玉米根;S:茎;L:叶片;T:雄穗;FE:雌穗 R: root; S: stem; L: leaf; T: tassel; FE: female ear
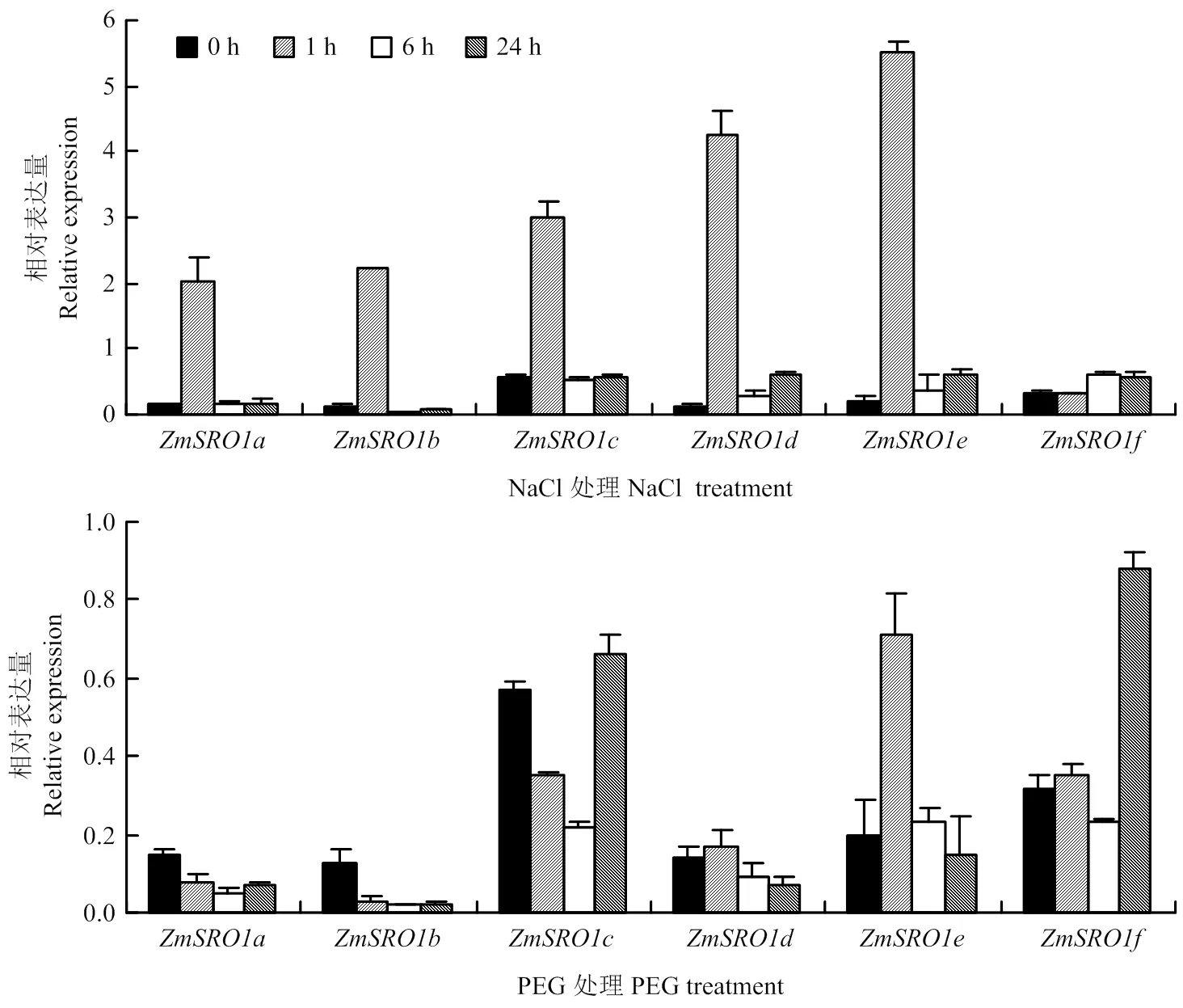
图6 胁迫条件下ZmSROs在玉米根系的表达
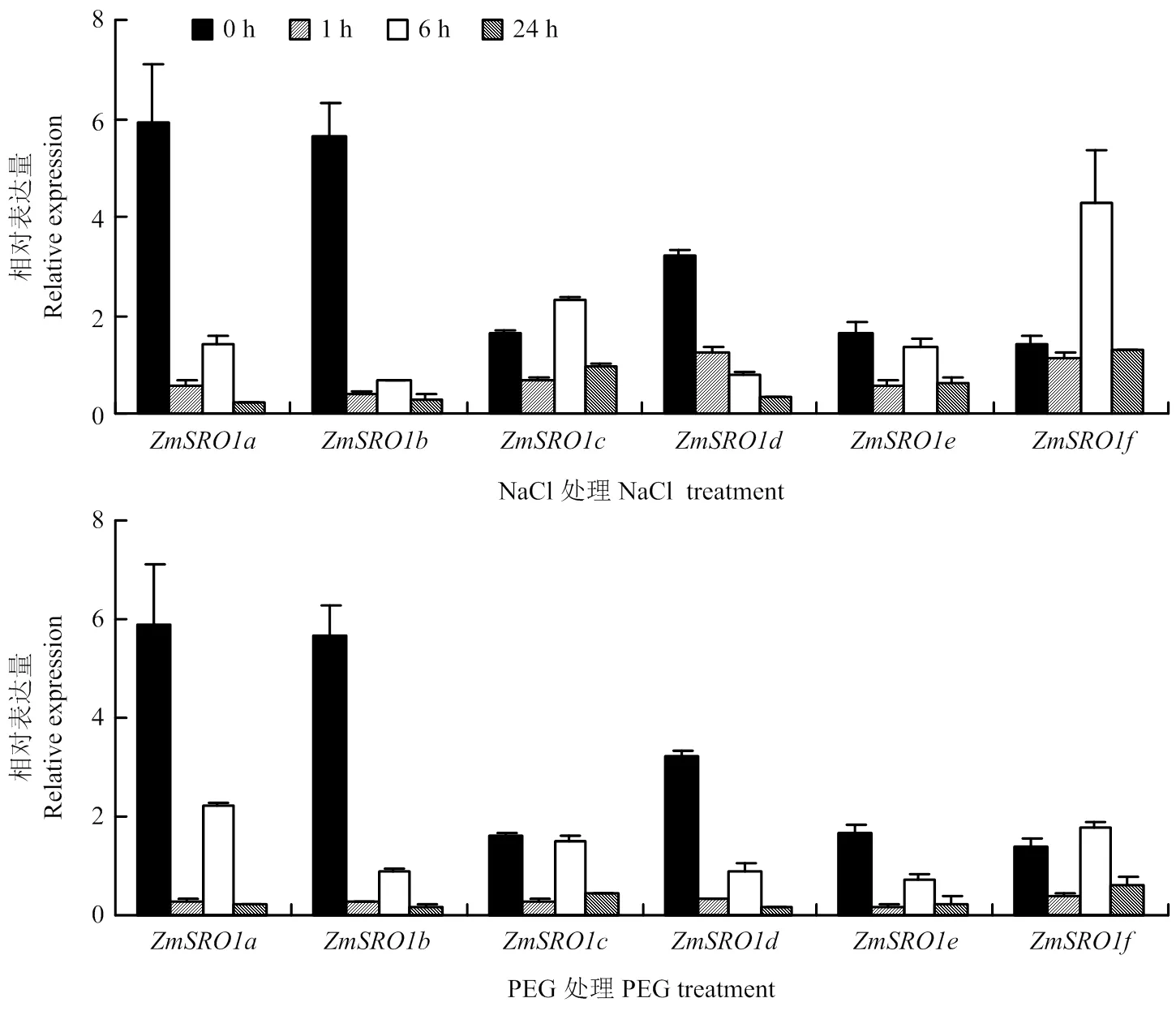
图7 胁迫条件下ZmSROs在玉米地上部的表达
SRO基因可以调控植物生长发育,因此其在植物不同组织部位的表达情况也备受关注。有研究表明苹果中SRO家族基因存在组织特异性表达,在果实中的表达显著高于其他组织,而其余在根、茎和花中的表达量较高,而在叶片和果实中的表达量低,同时在花中表达量最高,说明可能在苹果花发育过程中发挥重要作用[20]。水稻中和呈现组成性表达,和呈现组织特异性表达,在水稻三叶苗期和分蘖期表达量高,在叶片、鞘和4—5 cm幼穗中表达量低,在子房发育和胚乳成熟阶段表达量高,而在胚胎发育早期的表达量较低[17]。本研究结果显示在根系中表达量显著高于其他组织,暗示在玉米根系发育中发挥作用。另外在雄穗和雌穗中的表达量均较低,几乎不表达,表明其对玉米的生殖生长阶段作用较小(图6)。
SRO家族不仅影响植物的生长进程,而且在许多植物中均证实SRO家族可以响应植物的非生物胁迫。如在ABA和JA处理下显著上调表达[17],小麦中可以通过调节植物体内的氧化还原平衡来提高其耐干旱、高盐和H2O2胁迫的能力[18]。番茄在番茄根系中,盐胁迫条件下显著上调表达[19]。苹果表达量在100 μmol·L-1ABA和4℃处理下分别上调14和37倍,而在20%PEG处理下,、和分别上调18、17和14倍,说明可以响应ABA和寒害胁迫,、和响应干旱胁迫[20]。本研究中,玉米根系中5个基因均在200 mmol·L-1NaCl处理1 h显著上调表达,表明根系中这些基因可以迅速响应盐胁迫信号,另外和在干旱条件下显著上调表达,说明和响应干旱胁迫(图6)。相较根系SRO基因的表达量变化,地上部SRO基因表达量普遍呈现下调现象(图7),综上所述,可能在玉米根系响应干旱和高盐胁迫中发挥作用。
4 结论
从玉米全基因组中鉴定到6个SRO家族基因,均含有SRO蛋白特有的PARR催化中心结构域。系统进化树分析ZmSROs被分为3个亚类,其中ZmSRO1a/b/c属于Ⅰa亚类,ZmSRO1f属于Ⅰb亚类,ZmSRO1d/e属于Ⅰc亚类。在玉米根系特异性表达。玉米根系中的受盐胁迫诱导上调表达,和受干旱胁迫诱导上调表达,同时地上部受盐胁迫诱导上调表达,其余基因在干旱和盐胁迫均下调表达。
[1] 齐涛. 中国玉米国际竞争力研究[D]. 杨凌: 西北农林科技大学, 2011.
Qi T. The international competitiveness of cron in china [D]. Yangling: Northwest agriculture and forestry university, 2011. (in Chinese)
[2] Jaspers P, Blomster T, Brosché M, Salojärvi J, Ahlfors R, Vainonen J P, Reddy R A, Immink R, Angenent G, Turck F, Overmyer K, Kangasjärvi J. Unequally redundant RCD1 and SRO1 mediate stress and developmental responses and interact with transcription factors., 2009, 60(2): 268-279.
[3] 李保珠, 赵翔, 赵孝亮, 彭雷. 拟南芥SRO蛋白家族的结构及功能分析. 遗传, 2013, 35(10): 1189-1197.
LI B Z, ZHAO X, ZHAO X L, PENG L. Structure and function analysis ofSRO protein family., 2013, 35(10): 1189-1197. (in Chinese)
[4] Aravind L. The WWE domain: a common interaction module in protein ubiquitination and ADP ribosylation., 2001: 26: 273-275.
[5] Jaspers P, Overmyer K, Wrzaczek M, Vainonen J P, Blomster T, Salojärvi J, Reddy R O, Kangasjärvi J. The RST and PARP-like domain containing SRO protein family: analysis of protein structure, function and conservation in land plants., 2010, 11: 170.
[6] Otto H, Reche P A, Bazan F, Dittmar K, Haag F, Koch-Nolte F. In silico characterization of the family of PARP-like poly (ADPribosyl) transferases (pARTs)., 2005, 6: 139.
[7] Hassa P O, Haenni S S, Elser M, Hottiger M O. Nuclear ADPribosylation reactions in mammalian cells: where are we today and where are we going?, 2006, 70(3): 789-829.
[8] Ahlfors R, Lang S, Overmyer K, Jaspers P, Brosche M, Tauriainen A, Kollist H, Tuominen H, Belles-Boix E, Piippo M, Inze D, Palva T, Kangasjarvi J.RADICAL-INDUCED CELL DEATH1 belongs to the WWE protein-protein interaction domain protein family and modulates abscisic acid, ethylene, and methyl jasmonate responses., 2004, 16: 1925-1937.
[9] Ahlfors R, Brosché M, Kollist H, Kangasjärvi J. Nitric oxide modulates ozone‐induced cell death, hormone biosynthesis and gene expression in., 2009, 58(1): 1-12.
[10] Katiyar-Agarwal S, Zhu J, Kim K, Agarwal M, Fu X, Huang A, Zhu J K. The plasma membrane Na+/H+antiporter SOS1 interacts with RCD1 and functions in oxidative stress tolerance in., 2006, 103: 18816-18821.
[11] Fujibe T, Saji H, Arakawa K, Yabe N, Takeuchi Y, Yamamoto K T. A methyl viologen-resistant mutant of Arabidopsis, which is allelic to ozone-sensitive rcd1, is tolerant to supplemental ultraviolet-Birradiation., 2004, 134: 275-285.
[12] Overmyer K, Tuominen H, Kettunen R, Betz C, Langebartels C, Sandermann H, Kangasjärvi J. Ozone-sensitivemutant reveals opposite roles for ethylene and jasmonate signaling pathways in regulating superoxide- dependent cell death., 2000, 12(10): 1849-1862.
[13] Teotia S, Lamb R S. The paralogous genes RADICALINDUCED CELL DEATH 1 and SIMILAR TO RCD ONE 1 have partially redundant functions duringdevelopment., 2009, 151(1): 180-198.
[14] Teotia S, Lamb R S. RCD1 and SRO1 are necessary to maintain meristematic fate in., 2011, 62: 1271-1284.
[15] Borsani O, Zhu J H, Verslues P E, Sunkar R, Zhu J K. Endogenous siRNAs derived from a pair of natural cis-antisense transcripts regulate salt tolerance in., 2005, 123: 1279-1291.
[16] You J, Zong W, Li X, Ning J, Hu H, Li X, Xiao J, Xiong L. The SNAC1-targeted gene OsSRO1c modulates stomatal closure and oxidative stress tolerance by regulating hydrogen peroxide in rice., 2012, 64(2): 569-583.
[17] You J, Zong W, Du H, Hu H, Xiong L. A special member of the rice SRO family, OsSRO1c, mediates responses to multiple abiotic stresses through interaction with various transcription factors., 2014, 84(6): 693-705.
[18] Liu S, Liu S, Wang M, Wei T, Meng C, Wang M, Xia G. A wheat SIMILAR TO RCD-ONE gene enhances seedling growth and abiotic stress resistance by modulating redox homeostasis and maintaining genomic integrity., 2014, 26(1): 164-180.
[19] Babajani G, Effendy J, Plant A L. Sl-SROl1 increases salt tolerance and is a member of the radical-induced cell death 1-similar to RCD1 gene family of tomato., 2009, 176(2): 214-222.
[20] Li H, Li R, Qu F, Yao J, Hao Y. Identification of the SRO gene family in apples (Malus×domestica) with a functional characterization of MdRCD1., 2017, 13(5): 94.
[21] 吕有军, 杨卫军, 赵兰杰, 姚金波, 陈伟,李燕,张永山. 陆地棉SRO基因家族的鉴定及表达分析. 作物学报, 2017, 43(10): 1468-1479.
Lü Y J, YANG W J, ZHAO L J, YAO J B, CHEN W, LI Y, ZHANG Y S. Genome-wide identification and expression analysis of SRO genes family inL.., 2017, 43(10): 1468-1479. (in Chinese)
[22] Riechmann JL, Heard J, Martin G, Reuber L, Jiang C, Keddie J,Adam L, Pineda O, Ratcliffe OJ, Samaha RR, Creelman R, Pilgrim M, Broun P, Zhang JZ, Ghandehari D, Sherman BK, Yu G.transcription factors: genome-wide comparative analysis among eukaryotes., 2000, 290:2105-2110.
[23] Olsen AN, Ernst HA, Leggio LL, Skriver K. NAC transcriptionfactors: structurally distinct, functionally diverse., 2005, 10:79-87.
[24] Du H, Huang M, Zhang Z, CHENG S. Genome-wide analysis of the AP2/ERF gene family in maize waterlogging stress response., 2014, 198(1): 115-126.
[25] Chen L, Song Y, Li S, Zhang L, Zou C, Yu D. The role of WRKY transcription factors in plant abiotic stresses., 2012, 1819(2): 120-128.
[26] NOGUERO M, ATIF R M, OCHATT S, THOMPSON R D. The role of the DNA-binding One Zinc Finger (DOF) transcription factor family in plants., 2013,, 32-45.
[27] Wang Y, Deng D, Shi Y, MIAO N, BIAN Y, YIN Z. Diversification, phylogeny and evolution of auxin response factor (ARF) family: insights gained from analyzing maize ARF genes., 2012, 39(3): 2401-2415.
(责任编辑 李莉)
Genome-wideIdentification and Expression Analysis ofGenes Family in Maize
ZHAOQiufang, MAHaiyang, JIA Liqiang,CHENShu, CHENHongliang
(South Subtropical Crops Research Institute, Chinese Academy of Tropical Agricultural Science/Key Laboratory of Tropical Fruit Biology, Ministry of Agriculture, Zhanjiang 524091, Guangdong)
【Objective】The SRO (similar to rcd one) is a group of plant-specific proteins which have important functions in plant growth and development, and response to abiotic stress. Based on maize genome information, the SRO family genes were identified, and the sequences, gene localization, protein structure and their evolutionary relationships were analyzed. Meanwhile, the SRO family genes tissue-specific expression profiles and expression pattern under salt and drought stresses ofwere analyzed. This research can lay a foundation for researching the function of SRO family genes in the growth and response mechanisms of stress in maize. 【Method】The Arabidopsis SRO protein sequences were used as queries to identify SRO in maize genome and download all the SRO gene sequences, which information regarding the maize SRO genes, including CDS sequences , amino acid sequences and chromosome location were obtained from MaizeGDB. The gene structure, protein molecular weight, isoelectric point, secondary structure, subcellular localization, conserved domain, and conserved element were also prospected and analyzed by GSDS2.0, Expasy-protparam, SOPMA, Plant-mPLoc, EMBL-EBI, MEME bioinformatics tools. Meanwhile the homologous sequence alignment and phylogeny were analyzed by Clustalx (1.83) and MEGA6.0 software. The expression levels of ZmSROs genes in different maize tissues and under high salt and drought stresses were analyzed by real-time fluorescence quantitative PCR. 【Result】Six SRO family genes were identified from Maize genome, and designatedto.were mapped on 1, 4, 5 and 9 chromosome, and contained two to five introns. The CDS of 6varied in length from 1,215 to 1,791 bp, and their predicted protein products ranged from 404 to 596 aa, with corresponding molecular masses of 45.23 kD and 66.78 kD, and with predicted isoelectric points in the range of 7.01 to 9.17. Subcellular localization analysis showed that ZmSRO1a/ZmSRO1b/ZmSRO1c/ZmSRO1d were located in the chloroplast, ZmSRO1e was located in the peroxisome, and ZmSRO1f was located in the nucleus. Phylogentic analysis revealed that ZmSROs can be divided into three subgroups. Subgroup Ⅰa contains ZmSRO1a/b/c, subgroups Ⅰb contains ZmSRO1f, and subgroup Ⅰc contains ZmSRO1d and ZmSRO1e. Conservative structure domain analysis showed that ZmSRO1a/ZmSRO1b/ZmSRO1c/ZmSRO1d/ZmSRO1e contain the PARP and RST domain, lack the WWE domain. ZmSRO1f contains the PARP and WWE domain, lacks the RST domains. A total 5 motif were identified from ZmSROs proteins,named motif 1—motif 5. ZmSRO1a/ZmSRO1b/ZmSRO1c contain all of motif,ZmSRO1d/ZmSRO1e lack the motif 3,ZmSRO1f lacks the motif 5. Different tissues expression assay showed that allhad specific expressions in root. The expression ofin root were significantly up-regulated under high salinity treatment for 1 h.were down-regulated in shoots, However, the expression ofin shoot was significantly up-regulated under high salinity treatment for 6 h.in root was up-regulated under drought stress for 1 h and that ofwas up-regulated for 24 h. Meanwhile, the expressions ofin shoot were down-regulated under drought stress and that ofandhad no significant changes in shoot. 【Conclusion】Maize SRO family contains six genes, and ZmSROs genes could be classified into threes subgroups. Six ofgenes had specific expressions in root and could respond to high salt and drought stresses in varying degrees.
; SRO gene; abiotic stress; gene expression
2018-02-28;
2018-05-09
国家自然科学基金(31501818)、中国热带农业科学院基本科研业务费专项资金(1630062017006、1630062017015和1630062015003)
赵秋芳,E-mail:qiufangzhao@163.com。通信作者贾利强,E-mail:lqjia@mail.zaas.ac.cn
10.3864/j.issn.0578-1752.2018.15.0016

