基于简化基因组技术的啤酒花栽培种和野生种SNP位点开发及遗传结构分析
赵亚琴,樊丛照,张际昭,邱远金,辛海量,李晓瑾,张本刚,王果平*
1.新疆中药民族药研究所,新疆 乌鲁木齐 830002
2.第二军医大学,上海 200433
3.中国医学科学院药用植物研究所,北京 100193
啤酒花Humulus lupulusL.隶属桑科(Moraceae)葎草属HumulusL.,是新疆特色的药食同源植物,其体内含有的树脂类、黄酮类、黄腐酚等化学成分具有抗病毒、抗氧化等药用价值,同时啤酒花也是生产啤酒的基本原料,在保持啤酒风味及延长啤酒保质期等方面发挥着重要的经济价值[1-2]。野生啤酒花主要分布在新疆阿勒泰地区的额尔齐斯河及其分支流域,如桦树森林公园、哈巴河、布尔津、塔城地区和伊犁地区[3]。目前,在全国各地均有商业化种植。新疆不仅是啤酒花的原产地之一,也是我国啤酒花的主要产区,但据调查,目前新疆栽培种植的啤酒花主要是美国和日本引进的品种,在生产上存在品种单一、病虫害严重等问题[2]。为培育本土品种,实现本地资源利用的最大化,探究新疆野生资源的遗传多样性及其与外来品种之间的遗传差异性,有助于了解新疆本地野生资源的遗传潜力,为其遗传育种提供理论指导。目前对啤酒花遗传特性的研究相对较少[5-6],特别是对其遗传背景、新疆野生种与栽培种的关系等方面的研究,野生资源遗传信息的缺乏也成为制约其遗传育种的瓶颈。有研究表明,野生个体和栽培个体的遗传多样性在化学成分上存在明显差异。所以探究新疆野生啤酒花植物的遗传特征,明晰栽培个体与野生个体之间的遗传背景及其遗传关系,对培育地方品种、加强野生资源的利用具有重要意义。
随着高通量测序技术的快速发展,基于第二代测序技术的简化基因组测序specific-locus amplified fragment sequencing(SLAF-seq)、restriction-site associated DNA sequencing(RAD-seq)、genotyping by sequencing(GBS)等推动了进化生物学的又一步快速发展[7]。其中,SLAF-seq是一种高通量、高分辨率的SNPs位点识别与分型技术,是简化基因组测序的一次革命[8-10]。SLAF-seq技术利用生物信息学方法,对目标物种的参考基因组进行系统分析,设计一个合适的酶切方案,构建SLAF-seq文库,筛选出特异性长度片段,再应用高通量测序技术获得高通量标签序列,然后对数据分析,获取满足要求的SLAF片段。这些片段可以充分代表全基因组的序列特征信息,依据这些片段可以开发出大量的分子标记特别是单核苷酸多态(SNP)[11]。SLAF测序技术具高通量、高精度、短周期等优点,已经被运用于遗传定位、高密度遗传连锁图谱构建及不同个体间的多态性分析、系统进化和种质资源鉴定等领域[12-13]。本研究基于SLAF-seq测序技术以及获取在新疆同域分布的20个啤酒花的野生种及18个栽培个体的大量多态性SLAF标签,进而开发特异性强、稳定性高的的群体SNP位点。基于这些SNP位点从基因组水平明晰其野生种与栽培种之间的亲缘关系及遗传结构。旨在分析啤酒花不同个体间的遗传分化,探讨啤酒花野生种与栽培种之间的亲缘关系,为其野生资源育种提供科学依据。
1 材料
本研究在野生啤酒花集中分布区采集20个野生个体,其中7份来源与阿勒泰地区,5份来源于塔城地区,1份来源于伊犁地区,3份来源于新源县,4份来源于昌吉地区;18个栽培个体主要来源于阿勒泰地区、昌吉地区、哈密市及南疆的焉耆县、沙雅县及喀什地区。经中国医学科学院药用植物研究所张本刚研究员鉴定为啤酒花H.lupulusL.。采样过程中选择生长健壮、无病虫的植株,采集其幼嫩的叶片并记录经纬度,采集的新鲜样品迅速用硅胶迅速干燥,存放于-80 ℃冰箱备用。
2 方法
2.1 基因组DNA制备
本实验采用3×CTAB法提取分布在38个地点啤酒花的总DNA。采用1%琼脂糖凝胶电泳进行电泳检测DNA的产量和质量,确保所提取基因组DNA质量达到建库要求,利用Thermo核酸检测仪(Nanodrop 2000/2000c)测定DNA的浓度[14]。
2.2 酶切方案设计
根据已发布的啤酒花基因组信息(ftp://ftp.ncbi.nlm.nih.gov/genomes/all/GCA/000/831/365/GCA_000831365.1_hl_SW_version_1.0.fasta/GCA_000831 365.1_hl_SW_version_1.0.fasta_genomic.fna.gz).(基因组大小2.05 Gb,GC含量38.57%)作为参考基因组,组装出的基因组大小为2.05 Gb,GC含量为38.57%。利用北京百迈客生物技术公司自主研发的酶切预测软件对参考基因组进行酶切预测,最终选择Rsa I和HaeIII酶对基因组DNA酶切,酶切片段长度在364~414 bp的序列定义为SLAF标签,SLAF标签在基因组上基本分布均匀。最终获得酶切片段(SLAF标签)数满足预期标签数[15]。
2.3 测序及产出数据的质量分析
基于位于重复序列的酶切片段比例尽可能低,酶切片段在基因组上尽量均匀分布,酶切片段长度与实验体系吻合程度等原则设计酶切方案,对检测合格的各个地点的啤酒花个体基因组DNA分别进行酶切。对得到的酶切片段(SLAF标签)进行3’端加A处理、连接Dual-index测序接头、PCR扩增、纯化、混样、切胶选取目的片段,文库质检合格后用Illumina测序平台进行测序。为评估酶切实验的准确性,选用水稻日本晴Oryza sativaL.ssp.japonica作为对照进行测序。对测序得到的原始数据进行识别、过滤、质检、评估等分析,获取各个个体的序列(reads)。
2.4 SLAF标签和SNP标记的开发
利用Dual-index[16]对测序得到的原始数据进行识别,得到不同个体的reads分离的等位基因。过滤测序reads的接头后,进行测序质量和数据量的评估。通过Control数据评估RsaI和HaeIII酶的酶切效率,以此判断实验过程的准确性和有效性。本试验测序产生reads来源于都是不同地点的啤酒花在同一限制性内切酶的作用下产生的长度相同或相近的酶切片段,根据各个序列的相似度将38个个体的reads进行聚类,聚类到一起的reads来源于同一个SLAF标签[17-18]。同一SLAF标签在不同个体间的序列相似度远高于不同SLAF标签间的相似度;在同一个SLAF标签中存在不同个体间序列的差异(即有多态性),即可定义为多态性SLAF标签。以每个SLAF标签中深度最高的序列类型作为参考序列开发全基因组范围的SNP标记,对开发出的SNP根据完整度>0.5,MAF>0.05的标准进行筛选,最后利用筛选出的具有代表性的高质量SNP进行遗传进化树分析[19]、遗传结构分析[20]和主成分分析(principal component analysis,PCA)[21]。
利用北京百迈客生物技术公司开发的软件计算遗传多样性,利用AMOVA分析种群内、中群建和居群间的分子差异[22]。
3 结果与分析
3.1 建库评估
SLAF-seq测序reads为基因组DNA的酶切片段,其碱基分布会受到酶切位点和PCR扩增的影响,测序reads的前2个碱基会呈现与酶切位点一致的碱基分离,后续碱基分布会呈现不同程度的波动(图1)。

图1 啤酒花测序碱基分布Fig.1 Sequence distribution of H.lupulus
结果表明本研究双端比对效率在97.71%,比对效率基本正常。酶切效率是评价简化基因组实验是否成功的一个关键指标。基因组上的复杂结构区域(如环状结构域、连续酶切位点等)、基因组DNA样品纯度较低、酶切时间不足等因素都可能影响限制性内切酶的活性,导致部分酶切位点未被切开。通过统计测序reads插入片段中残留酶切位点的比例,统计比例越高,酶切效率越好。本实验中水稻日本晴数据的酶切效率为89.92%,双端比对效率为97.71%,酶切效率为89.92%,表明酶切反应及SLAF建库均为正常。
3.2 测序数据统计与评估
本研究采用读长126 bp×2作为后续的数据评估和分析数据。测序质量值(Q)是评估高通量测序单碱基错误率的重要指标,测序质量值越高对应的碱基测序错误率越低。如果某碱基测序出错的概率为0.001,则该碱基的质量值Q应该为30(Q30)。对38个地点个体的测序数据进行统计,包括reads数量、Q30和GC含量。测序平均Q30为93.42%,平均GC含量为42.53%。由于所测序列的Q30数据较高,表明碱基出错率很低,测序结果可靠(表1)。
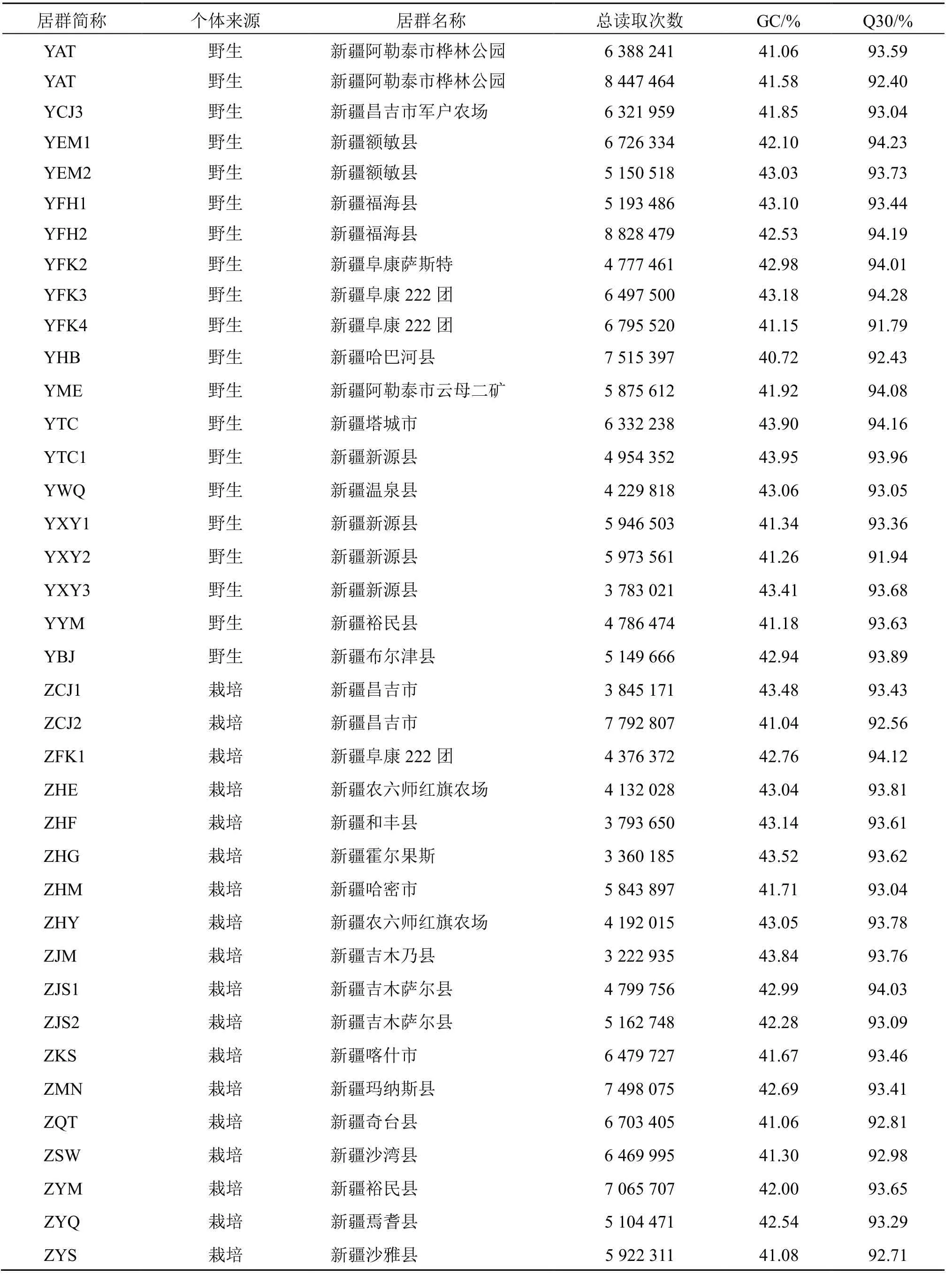
表1 啤酒花测序质量(Q30)及GC含量Table 1 Content of Q30 and GC of H.lupulus
3.3 SLAF标签与SNP标记的鉴定
通过序列分析,从38个地点的啤酒花基因组中共获得了863 228个SLAF标签。标签的平均测序深度为13.40 X,其中,多态性SLAF标签有443 922个,共获得2 867 140个高质量的群体SNP标记(表2)。
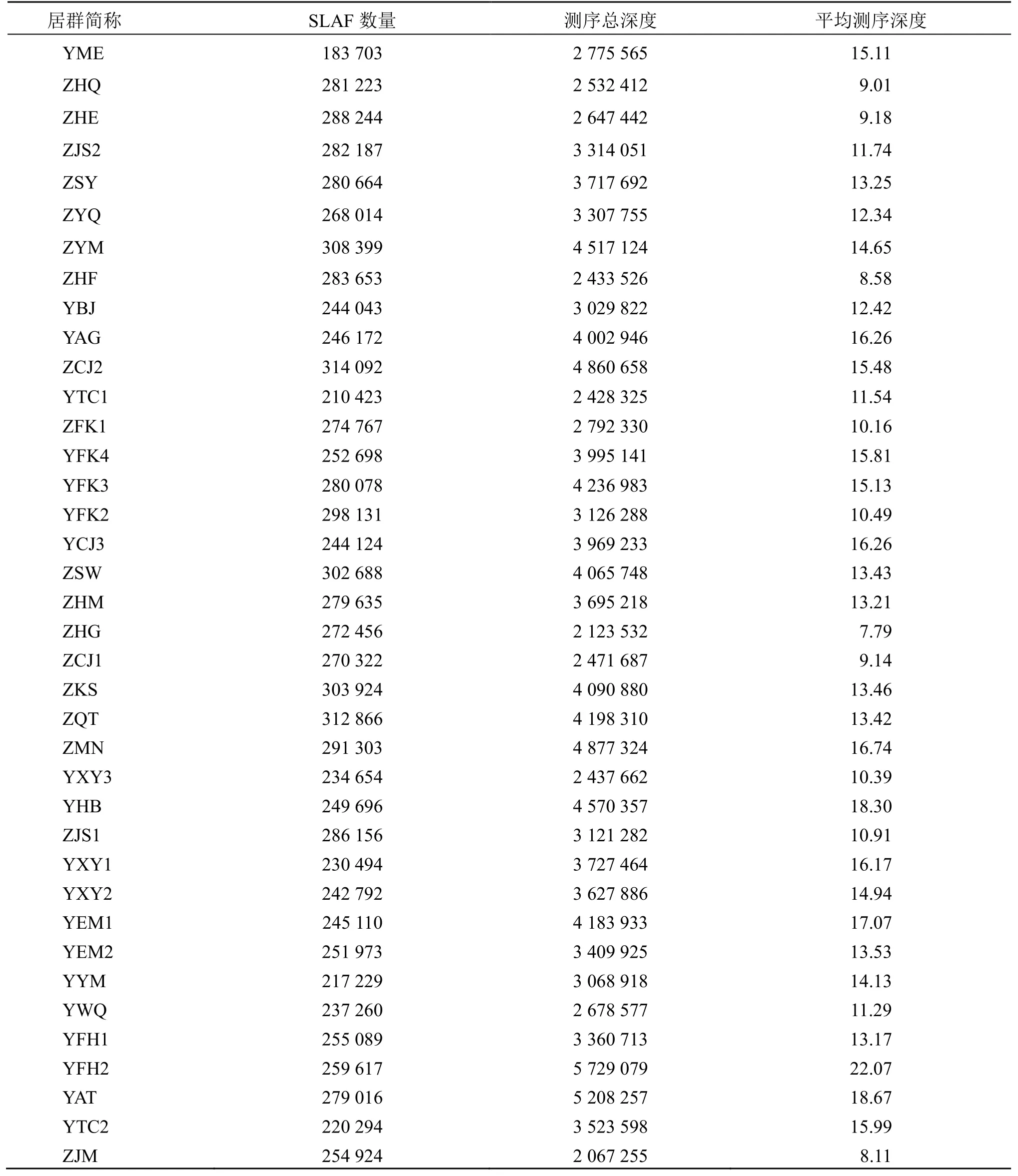
表2 啤酒花SLAF标签Table 2 Label of SLAF of H.lupulus
3.4 系统发育分析
基于2 867 140个SNP位点对38个不同地点啤酒花的野生种与栽培种进行系统发育分析,结果表明38个啤酒花个体大致形成2个主要类群,类群I包括阿勒泰地区、塔城地区、伊犁地区的16个野生个体,类群II主要包括所有的栽培个体及阜康及昌吉地区的4个野生种(YFK2、YFK3、YCJ3、YFK4,图2)。聚类分析显示栽培个体与多数野生个体各自单独聚成一类,表明栽培个体与野生个体之间的亲缘关系较远。

图2 基于邻接法的啤酒花的进化树Fig.2 Evolutionary trees of H.lupulus based on NJ method
3.5 遗传结构及PCA分析
基于开发出的2 867 140个SNP位点分析38个地区啤酒花的遗传结构。交叉验证聚类结果表明,当K=2时,交叉验证错误率最低,可将38份啤酒花可划分成2个不同的类群(图3),结果与系统发育结果一致。S1类群(绿色)包括16个地区的野生个体,S2类群(黄色)包括18个地区的栽培个体及4个野生个体。结果表明野生个体与栽培个体之间存在较大的遗传差异,形成了较明显的遗传分化。而在阜康及昌吉地区的栽培啤酒花品种来源多样,遗传组成比较复杂。

图3 交叉各个K值对应的个体聚类图及不同K值所对应的的交叉验证错误率Fig.3 Admixture individual cluster values corresponding to each K value and admixture validation error rate corresponding to different K values
基于开发的SNP位点对20个野生个体及18个栽培个体进行PCA分析(PC1和PC2的累积方差贡献率为35.89%),如图4所示,20个野生个体和18个栽培个体在空间上表现出明显的分离趋势,说明野生个体与栽培个体之间具有明显的遗传差异性。其中野生个体分布在主成分坐标轴的右侧,且彼此紧密地混聚在一起,说明采集的野生资源遗传基础总体较为狭窄,而栽培个体由于其品种来源多样,遗传背景复杂,表现出比野生个体更广泛分布的特征,并且不同地区间的栽培个体之间也表现出较大的差异性。

图4 PCA分析Fig.4 Analysis of principal components
3.6 遗传多样性与遗传分化
38个啤酒花个体总的Shanon-Wiener指数为0.397,Nei多样性指数为0.249。野生个体与栽培个体的遗传多样性具有一定的差异,整体表现为野生个体总的遗传多样性(0.454)大于栽培个体总的遗传多样性(0.398);总的Nei多样性指数为0.249,其中野生个体的Nei多样性指数(0.293)大于栽培个体(0.250)。分子方差分析表明野生个体与栽培个体之间存在较大的遗传分化,其遗传变异主要来源于野生个体与栽培个体之间。
4 讨论
简化基因组测序技术通过寻找合适的限制性内切酶来降低基因组的复杂程度,可显著降低测序成本,同时还能获得数量可观的基因组变异信息并且能有效地克服基因组复杂的问题,已经被应用于多种植物的遗传变异研究上[24-25]。基于SALF-seq简化基因组数据对啤酒花的20个野生个体及18个栽培个体的遗传结构分析表明,野生个体与栽培个体整体上存在较明显的遗传分化,但是在昌吉阜康区域的野生个体与栽培个体之间的遗传分化并不显著,存在一定的基因交流。这可能与当地进行大范围的野生资源育种相关,尤其是昌吉地区啤酒花种植面积可达13.3 km2[26],大面积的栽培种植也增加了野生种之间的基因交流。
从聚类分析及PCA分析均表明,绝大多数栽培个体与野生个体(昌吉及阜康地区除外)之间存在明显的遗传差异,在DNA水平上单独聚类,形成各自的遗传结构。其中个别野生个体与当地栽培个体具有较近的亲缘关系,说明不同类群间,尤其是利用野生资源育种的栽培个体和野生个体之间存在一定的基因交流。可见栽培种与野生种之间非绝对的生殖隔离,可以通过杂交的方式获得变异植株,实现啤酒花种质创新和遗传基础拓宽。利用遗传信息对新疆啤酒花的野生个体与栽培个体进行遗传多样性分析的报道较少,原俊凤等[5-6]利用分子标记技术对新疆4个野生居群的遗传多样性研究结果表明新疆的野生啤酒花居群具有很高的遗传多样性。本研究也发现新疆野生啤酒花个体的遗传多样性高于栽培个体,丰富的遗传资源是野生啤酒花育种中不可或缺的宝贵资源,同时也为新疆野生啤酒花资源的利用及本土品种的培育提供了一定的指导价值。
利益冲突所有作者均声明不存在利益冲突

