铁氧化菌菌株EEELCW01的基因组分析及砷转化功能
熊潇然,邹奇,吴川,夏礼兵,潘炜松*
铁氧化菌菌株EEELCW01的基因组分析及砷转化功能
熊潇然1,邹奇2,吴川3,夏礼兵1,潘炜松1*
(1.湖南农业大学生物科学技术学院,湖南 长沙 410128;2.华南师范大学生命科学学院,广东 广州 510006;3.中南大学冶金与环境学院,湖南 长沙 410083)
以从砷污染农田土壤中分离的1株铁氧化细菌EEELCW01为研究对象,对该菌进行全基因组分析,通过GO、KEGG、COG等数据库比对,预测该菌砷相关基因的功能;采用水培试验,验证该菌的砷转化能力。结果表明:菌株EEELCW01基因组大小为4 714 242 bp,具有2条大小分别为2 065 078 bp和2 649 164 bp的染色体,GC含量为55.99%,染色体上有4588个CDS,包含58个tRNA和12个rRNA;COG数据库注释表明该菌基因功能主要集中在氨基酸转运代谢、无机离子转运代谢等过程,GO数据库注释表明该菌主要包含膜组成部分、氧化还原过程和相关酶活性等功能,KEGG注释显示代谢相关的基因占比最高;菌株EEELCW01基因组中含有多种与砷代谢相关的基因(、、、、、、和);菌株EEELCW01将As(Ⅴ)还原为As(Ⅲ)的能力强,有氧条件下,3 d时还原率达40.1%。可见,可利用该菌株促进砷的生物还原,并联合砷超富集植物进行环境砷污染修复。
砷;铁氧化菌;砷转化;基因组分析;功能注释
据估计,中国土壤中砷的平均质量分数为11.2 mg/kg,远高于世界平均值(7.2 mg/kg)[1]。除了火山活动、岩石风化等自然因素外,矿产资源的开采、运输、冶炼及工业废渣和废水排放等人为因素[2–3]均会加速环境中砷元素的释放,从而引发不同程度的土壤砷污染。土壤砷污染在导致植物中毒、作物减产的同时,还会严重威胁人体健康[4]。
传统的砷污染治理,主要基于氧化、共沉淀、过滤、吸附、离子交换等方法[5]。相对于高成本的传统治理方法,微生物修复技术因其效果好、投资小、易于管理与操作及不产生二次污染等优点,正日益受到人们的重视,成为土壤砷污染修复的研究热点。微生物通过改变砷的氧化还原状态,进而改变土壤砷的离子价态及活性,使亚砷酸盐氧化为砷酸盐,从而降低砷的毒性。铁氧化细菌属于化能无机营养型菌,可以氧化Fe(Ⅱ)生成铁的(氢)氧化物矿物,包括针铁矿、水铁矿、赤铁矿、纤铁矿和磁铁矿等[6–9]。这些生物合成的含铁矿物通过吸附、沉降和共沉淀等过程,可有效去除水体或土壤中的砷。具有铁氧化能力的微生物分布较广,在细菌域和古菌域中均有分布[10],细菌域中主要分布在变形杆菌门(Proteobacteria)、厚壁菌门(Firmicute)、硝化螺菌门(Nitrospira)和绿细菌门(Chlorobi)中,古菌域中主要分布在广古菌门(Euryarchaeota)[11]。已经报道的砷氧化菌株属于变形菌门中的α–、β–、γ–变形菌纲及厚壁菌门,且广泛分布于土壤、沉积物、地下水、矿山尾矿和酸性矿山废水及热泉中[12–14]。研究[15]表明,三价砷的氧化、五价砷的还原外排、三价砷的甲基化外排、通过抗性蛋白扣押砷离子等机制往往由其遗传学基础所决定。微生物主要通过细胞膜上的甘油通道蛋白GlpF或者同源蛋白FpslP使亚砷酸盐进入到微生物细胞内[16],由基因编码的ATP亚砷酸盐转运蛋白外排[17]。As(Ⅴ)还原机制由操纵子控制实现,主要包括构成操纵子模式的、、、、和基因[18]。前期研究从稻田土壤中筛选出1株铁氧化菌EEELCW01,盆栽试验结果显示,EEELCW01可显著降低土壤有效态As的含量[19–20]。本研究中,笔者对该铁氧化菌进行基因组生物信息学分析及其砷转化功能研究,阐述该菌影响砷形态转换和环境行为的分子机制,挖掘其环境砷修复潜力,以期为建立高效、低成本的砷污染生物修复方法提供依据。
1 材料与方法
1.1 供试材料
供试铁氧化菌菌株EEELCW01分离于湖南省长沙市芙蓉区某典型砷污染农田,其可在微好氧条件下进行亚铁氧化。该菌在NCBI GenBank上的登录号为CP047599,前期研究鉴定其属于苍白杆菌属()[19]。
1.2 EEELCW01基因组DNA的提取及测序
将纯化后的菌株EEELCW01接种到LB液体培养基,于28 ℃、150 r/min条件下培养至稳定期,采用DNA提取试剂盒(MagaBio Soil/Feces Genomic DNA Purification Kit)提取菌株的基因组DNA,DNA片段化处理后构建文库,并送往上海美吉生物医药科技有限公司进行测序。
1.3 EEELCW01全基因组功能基因分析
原始测序数据运用FastQC(v0.11.8)、cutadapt (v1.18)进行质控和去接头后获得干净序列;运用SOAPdenovo进行基因组组装,运用GapCloser对组装得到的初步结果进行补洞和碱基矫正,以获得最终组装结果;运用SSpace对组装得到的Scaffolds进行拼接延伸,最终获得组装的EEELCW01基因组。运用CG View Server进行EEELCW01基因组圈图绘制。
参照文献[21–22]的方法,采用GeneMark对EEELCW01全基因组进行ORF预测,主要利用GO(Gene Ontology)、KEGG(Kyoto Encyclopedia of Genes and Genomes)和COG(Cluster of Orthologous Groups of Proteins)数据库并运用diamond(v0.7.9)进行基因注释和分析。同时,将菌株EEELCW01的基因组核酸序列与砷相关基因的数据库进行tblastx比对,得到ORF在基因组Scaffolds的坐标,从而将砷的相关基因定位到菌株的基因组中。运用Blastn(http://blast.ncbi.nlm.nih.gov/)将EEELCW01基因组的砷相关基因簇与YNTRS–40、ATCC 17100、ES–1和spAS–1进行对比,并运用TB Tools(v1.108)进行基因组共线性分析。
1.4 EEELCW01的砷形态转化功能分析
将1 mL含有纯化EEELCW01的菌液接种在体积分数为1%的重碳酸盐缓冲体系中,在28 ℃下振荡培养2 d后对培养液进行过滤,以去除生成的铁沉淀物;将滤液在8000 r/min下离心10 min,收集沉淀下来的菌体,用pH 7.0的无菌去离子水洗3次后制成细菌悬浮液,富集培养。EEELCW01的砷形态转化功能分析试验设4个处理:T1,厌氧条件下(氮气吹扫后密封),同时添加富集的4% EEELCW01菌液和灭菌的1 mmol/L As(Ⅲ)溶液;T2,厌氧条件下,只添加灭菌的1 mmol/L As(Ⅲ)溶液;T3,有氧条件下,同时添加富集的4%EEELCW01菌液和灭菌的1 mmol/L As(Ⅴ)溶液;T4,有氧条件下,只添加灭菌的1 mmol/L As(Ⅴ)溶液。4种处理均在25 ℃预先紫外灭菌的恒温培养箱中培养3 d,分别于培养1、2、3 d采样。离心(10 000 r/min、15 min)后将各处理组的菌液和菌体分离,上清液经0.45 μm滤膜过滤,采用液相色谱(岛津LC–15C)–原子荧光(吉天仪器AFS–8230)联用仪测定As(Ⅲ)、As(Ⅴ)含量。
2 结果与分析
2.1 EEELCW01的基因组信息
EEELCW01的基因组大小为4 714 242 bp,GC含量为55.99%,其2条染色体上基因大小分别为2 065 078 bp和2 649 164 bp,共包含4588个CDS(编码序列)。运用CGView Server绘出的EEELCW01的基因组圈图如图1所示。EEELCW01的基因组中含有58个tRNA和12个rRNA,EEELCW01在NR、GO、KEGG和COG数据库中得到注释的基因数分别为4588、3376、2479和4081个,分别占基因总数的100%、73.58%、54.03%和88.95%。

图1 EEELCW01的全基因组圈图
2.2 EEELCW01的基因组功能注释结果
采用COG数据库对基因产物进行直系同源分类发现,鉴定EEELCW01基因得到的4081个编码蛋白可分为20个功能类别,其中大部分COG注释的基因与细菌细胞的基本功能有关,如氨基酸转运代谢、无机离子转运代谢、转录、碳水化合物转运代谢、细胞壁/膜/被膜生物合成、能量生成转换等(表1)。注释基因数量最多的为氨基酸转运代谢(447个),其次为无机离子转运代谢(330个),具备次级代谢物生物合成、转运和代谢(Q)功能的基因共有51个,具备胞内转运、分泌和膜泡运输(U)功能的基因有65个,具备碳水化合物转运代谢(G)功能的基因共有290个,表明该菌可能与重金属离子的转化和运输有关。

表1 EEELCW01基因组的eggNOG/COG注释结果
采用KEGG对EEELCW01基因组进行注释,结果如表2所示。EEELCW01共有2479个基因分布在196种代谢通路中,与代谢相关的基因在注释基因中的占比最高,主要包括碳水化合物代谢、辅助因子和维生素代谢、能量代谢、脂代谢、核苷酸和氨基酸代谢等。较高比例的膜转运、辅助因子和维生素代谢等基因表明菌株EEELCW01对环境胁迫有较强的适应能力。

表2 EEELCW01基因组的KEGG注释结果
运用GO数据库对EEELCW01的基因组进行功能注释,结果如图2所示。EEELCW01的基因在生物学过程、分子功能和细胞组分3个分支中共有42类,其中ATP、DNA等结合和转录因子活性以及氧化还原酶、水解酶等酶活性在分子功能中发挥重要作用;膜组成部分、质膜和细胞质在细胞组分内占优势;而氧化还原过程、转录调控和DNA模板、转运和跨膜运输等在生物学进程中表现活跃。GO注释结果表明,EEELCW01有着复杂的代谢调控网络,主要集中在代谢途径。

图2 EEELCW01基因组的GO注释结果
综合分析NR、GO、KEGG和COG数据库,根据值和同源性分析发现,该菌株基因组中含有、、、、、、和等与砷代谢相关的基因(表3)。比较菌株EEELCW01和其他4种已测序的铁氧化菌株(YNTRS–40[23]、ATCC 17100[24]、ES–1[25]及砷氧化菌sp. AS–1[26])的砷代谢相关基因簇(图3),发现这几种菌株中均含有与砷抗性相关的基因和。和的多拷贝特点可能与菌株EEELCW01能够耐受高浓度的砷及高效的砷转化能力有关。EEELCW01和其他4株菌株的砷代谢基因簇有差异:菌株EEELCW01含有砷甲基化基因簇,而其他4种菌株没有;菌株EEELCW01的基因簇在染色体上的顺序与菌株YNTRS–40及ES–1的基因顺序相反,说明不同铁氧化菌株砷功能基因可能有不同的来源。
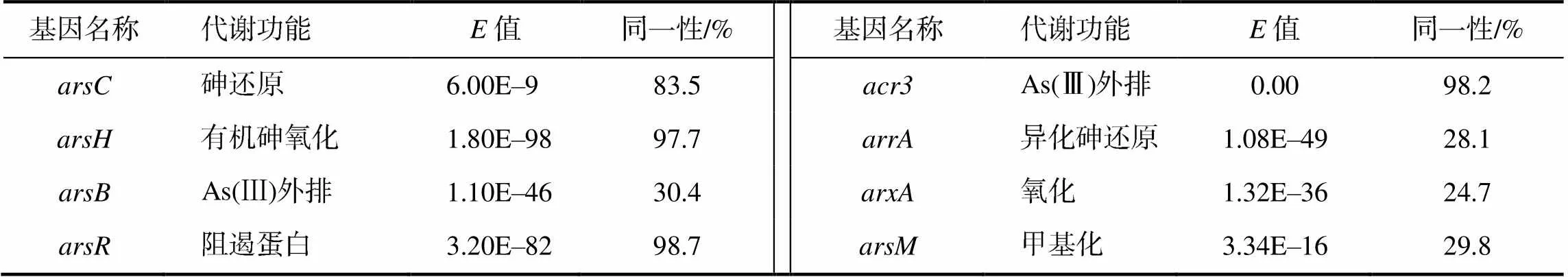
表3 EEELCW01基因组中砷代谢相关基因

图3 铁氧化菌砷代谢基因簇
2.3 EEELCW01对砷形态转化的影响
从图4可知,厌氧条件下,T1处理组培养液中的As(Ⅲ)浓度有略微下降,这可能是由细菌的吸附和转化共同造成的;T1和T2的As(Ⅴ)浓度基本为0,且两者的As(Ⅲ)浓度无明显区别,表明在厌氧条件下该菌没有介导砷的氧化过程。有氧条件下,T4的As(Ⅴ)浓度均无明显变化,As(Ⅲ)浓度基本为0;T3中生成了较多的As(Ⅲ),同时As(Ⅴ)浓度下降,随着培养的持续进行,As(Ⅴ)浓度持续降低,3 d时,As(Ⅴ)浓度最低,下降了0.367 mmol/L,还原率为40.1%,表明在有氧条件下EEELCW01菌株也可将As(Ⅴ)还原为As(Ⅲ)。
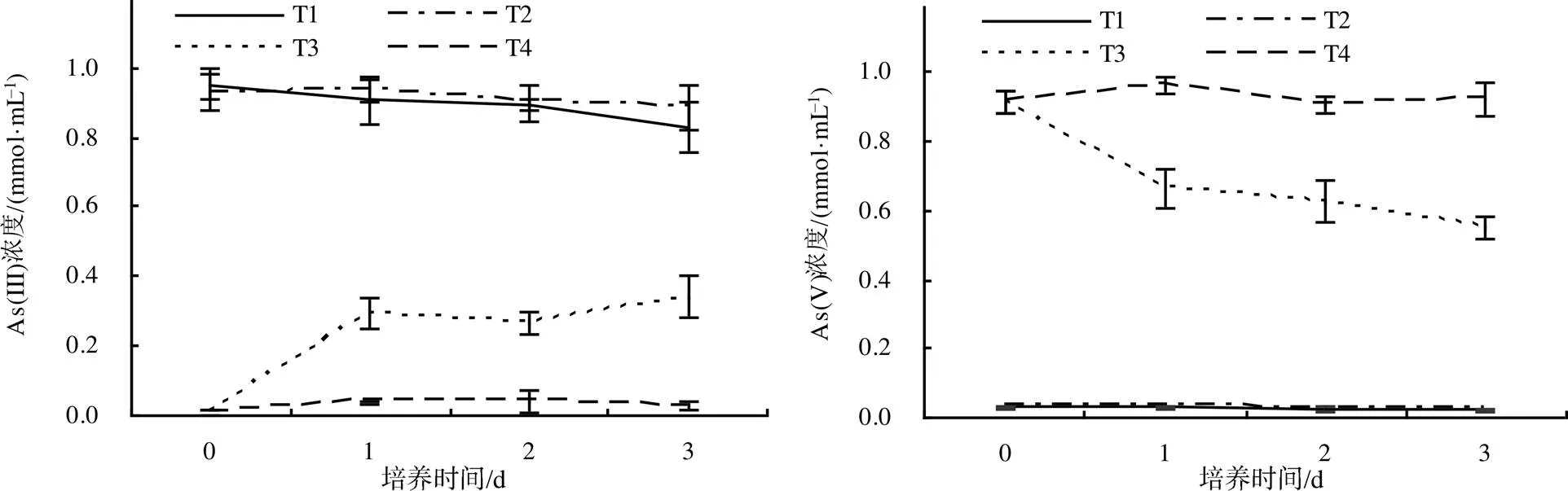
图4 EEELCW01对砷的氧化还原状况
3 结论与讨论
本研究中,笔者对自湖南省长沙市芙蓉区某典型砷污染农田中分离获得的1株苍白杆菌EEELCW01进行了基因序列分析和砷转化相关功能研究。结果表明,该菌株中和代谢相关的基因最多,代谢功能包括碳水化合物、核苷酸、能量和氨基酸代谢等。这和柳朝阳等[26]报道的1株砷氧化菌sp. AS–1类似。AS–1菌株同时具备氧化Sb(Ⅲ)的能力,表明对砷具有高耐受性的菌株可能具备类似的代谢途径。菌株EEELCW01基因组中含有许多与砷代谢相关的基因,如、、、、、、和。为阻遏蛋白基因,与启动子结合时会抑制其他基因的转录与表达;是载体蛋白基因,与利用膜电势将As(Ⅲ)排出有关;基因负责编码As(Ⅴ)还原酶,在As(Ⅴ)还原中起关键作用[27]。EEELCW01能将进入胞内的As(Ⅴ)还原成As(Ⅲ),并由膜蛋白将As(Ⅲ)泵出,具有细胞质砷还原功能,可能就是由于该菌具有这些基因所致。前期研究[19]发现,EEELCW01为嗜中性微好氧铁氧化菌,通过生物铁氧化代谢产生还原型辅酶和三磷酸腺苷,并含有、、和,可耦合进行硝酸盐和铁代谢过程,其拥有完整的硝酸盐还原代谢路径。前期研究[19]还表明,EEELCW01促进了Fe(II)的氧化和亚硝酸根的还原,添加2%或更多的EEELCW01细菌可显著去除水体中的砷,从而形成鳞铈石和2种含砷矿物(霰石、菱铁矿),表明该砷耐性菌有望用于修复土壤或者水体砷污染。
本研究中,菌株EEELCW01在无氧和有氧条件下均无明显砷氧化现象。刘琼[28]的研究表明,在好氧及厌氧2种不同条件下,好氧sp. GE–1及厌氧Strain2002在无铁的环境下不吸收或生物吸附As来降低砷的浓度,且不会改变As的价态。菌株EEELCW01虽具有砷氧化基因,但未体现砷氧化功能,可能是由于缺乏铁离子导致的。环境中矿物对As(Ⅲ)的吸附性较低,而一些铁铝化合物对As(Ⅴ)的吸附能力较强,从而限制了As(Ⅴ)的流动性[29],As(Ⅲ)的迁移率大于As(Ⅴ)的迁移率。菌株EEELCW01在有氧条件下仍具有较强的砷还原功能,3 d可还原0.367 mmol/L As(Ⅴ),还原率达40.1%。在砷污染治理过程中,可通过菌株EEELCW01将As(Ⅴ)还原为As(Ⅲ),并与超富集砷的植物(如蜈蚣草[30])协同进行土壤砷的植物提取修复。
[1] ZHAO F J,MA Y B,ZHU Y G,et al.Soil contamination in China:current status and mitigation strategies[J]. Environmental Science & Technology,2015,49(2):750–759.
[2] 安礼航,刘敏超,张建强,等.土壤中砷的来源及迁移释放影响因素研究进展[J].土壤,2020,52(2):234–246.
[3] 吴佳,谢明吉,杨倩,等.砷污染微生物修复的进展研究[J].环境科学,2011,32(3):817–824.
[4] HARTLEY T N,MACDONALD A J,MCGRATH S P,et al.Historical arsenic contamination of soil due to long- term phosphate fertiliser applications[J].Environmental Pollution,2013,180:259–264.
[5] YAN L,YIN H H,ZHANG S,et al.Biosorption of inorganic and organic arsenic from aqueous solution byBY-3[J].Journal of Hazar- dous Materials,2010,178(1/2/3):209–217.
[6] LIU Q,GUO H M,LI Y,et al.Acclimation of arsenic- resistant Fe(II)-oxidizing bacteria in aqueous environment [J].International Biodeterioration & Biodegradation,2013,76:86–91.
[7] PARK J,HAN Y,LEE E,et al.Bioleaching of highly concentrated arsenic mine tailings by[J].Separation and Purification Technology,2014,133:291–296.
[8] XIU W,GUO H M,SHEN J X,et al.Stimulation of Fe(II) oxidation,biogenic lepidocrocite formation,and arsenic immobilization bysp.strain 2002 [J].Environmental Science & Technology,2016,50(12):6449–6458.
[9] CORKHILL C L,WINCOTT P L,LLOYD J R,et al.The oxidative dissolution of arsenopyrite (FeAsS) and enargite (Cu3AsS4) by[J]. Geochimica et Cosmochimica Acta,2008,72(23):5616–5633.
[10] LANE D J,JR HARRISON A P,STAHL D,et al. Evolutionary relationships among sulfur- and iron- oxidizing eubacteria[J].Journal of Bacteriology,1992,174(1):269–278.
[11] WEBER K A,ACHENBACH L A,COATES J D. Microorganisms pumping iron:anaerobic microbial iron oxidation and reduction[J].Nature Reviews Microbiology,2006,4(10):752–764.
[12] OREMLAND R S,STOLZ J F,HOLLIBAUGH J T.The microbial arsenic cycle in Mono Lake,California[J]. FEMS Microbiology Ecology,2004,48(1):15–27.
[13] QUÉMÉNEUR M,HEINRICH-SALMERON A,MULLER D,et al.Diversity surveys and evolutionary relationships ofgenes in aerobic arsenite-oxidizing bacteria[J].Applied and Environmental Microbiology,2008,74(14):4567–4573.
[14] FAN H,SU C,WANG Y,et al.Sedimentary arsenite- oxidizing and arsenate-reducing bacteria associated with high arsenic groundwater from Shanyin,Northwestern China[J].Journal of Applied Microbiology,2008,105(2):529–539.
[15] YANG H C,FU H L,LIN Y F,et al.Pathways of arsenic uptake and efflux[J].Current Topics in Membranes,2012,69:325–358.
[16] WYSOCKI R,CHÉRY C C,WAWRZYCKA D,et al. The glycerol channel Fps1p mediates the uptake of arsenite and antimonite in[J]. Molecular Microbiology,2001,40(6):1391–1401.
[17] MARTIN P,DEMEL S,SHI J,et al.Insights into the structure,solvation,and mechanism of ArsC arsenate reductase,a novel arsenic detoxification enzyme[J]. Structure,2001,9(11):1071–1081.
[18] 陈淋霞,张萌,石佳佳,等.铁氧化菌耐砷机制及其砷污染修复应用的研究进展[J].微生物学通报,2020,47(9):3054–3064.
[19] LUO X H,JIANG X X,XUE S G,et al.Arsenic biomineralization by iron oxidizing strain (sp.) isolated from a paddy soil in Hunan,China[J].Land Degradation & Development,2021,32(6):2082–2093.
[20] WU C,CUI M Q,XUE S G,et al.Remediation of arsenic- contaminated paddy soil by iron-modified biochar[J]. Environmental Science and Pollution Research,2018,25(21):20792–20801.
[21] WANG H T,ZHU D,LI G,et al.Effects of arsenic on gut microbiota and its biotransformation genes in earthworm[J].Environmental Science & Technology,2019,53(7):3841–3849.
[22] ZHAO Y,SU J Q,YE J,et al.AsChip:a high-throughput qPCR chip for comprehensive profiling of genes linked to microbial cycling of arsenic[J].Environmental Science & Technology,2019,53(2):798–807.
[23] ZHANG Y,ZHANG S,ZHAO D,et al.Complete genome sequence ofYNTRS-40,a strain of the ferrous iron- and sulfur- oxidizing acidophile[J].Microorganisms,2019,8(1):2.
[24] BROWN P J B,KYSELA D T,BUECHLEIN A,et al. Genome sequences of eight morphologically diverse Alphaproteobacteria[J].Journal of Bacteriology,2011,193(17):4567–4568.
[25] HÄDRICH A,TAILLEFERT M,AKOB D M,et al. Microbial Fe(II) oxidation byES-1 in the presence of Schlöppnerbrunnen Fen-derived humic acids[J].FEMS Microbiology Ecology,2019,95(4):fiz034.
[26] 柳朝阳,向利,王红梅,等.砷氧化菌株的筛选及sp.AS-1基因组分析[J].微生物学报,2020,60(11):2538–2554.
[27] YANG J B,RAWAT S,STEMMLER T L,et al.Arsenic binding and transfer by the ArsD As(Ⅲ) metallo- chaperone[J].Biochemistry,2010,49(17):3658–3666.
[28] 刘琼.铁氧化菌的耐砷性能及除砷特征[D].北京:中国地质大学(北京),2012.
[29] OREMLAND R S,STOLZ J F.The ecology of arsenic[J].Science,2003,300:939–944.
[30] MA L Q,KOMAR K M,TU C,et al.A fern that hyperaccumulates arsenic[J].Nature,2001,409:579.
Genomic analysis and arsenic transformation of iron-oxidizing strain EEELCW01
XIONG Xiaoran1,ZOU Qi2,WU Chuan3,XIA Libin1,PAN Weisong1*
(1.College of Bioscience and Biotechnology, Hunan Agricultural University, Changsha, Hunan 410128, China; 2.College of Life Science, South China Normal University, Guangzhou, Guangdong 510006, China; 3.School of Metallurgy and Environment, Central South University, Changsha, Hunan 410083, China)
In this study, the iron-oxidizing bacterium EEELCW01, isolated from As-contaminated soils, underwent comprehensive genome analysis. The potential function of As-related genes was assessed through comparative analysis with GO, KEGG and COG databases, while the As transformation capacity of this strain was investigated via hydroponic experiments. Results unveiled that EEELCW01 possessed a genome size of 4 714 242 bp, encompassing two chromosomes sized at 2 065 078 bp and 2 649 164 bp, respectively, with a GC content of 55.99%. The chromosomes harbored 4588 CDSs, 58 tRNAs and 12 rRNAs. COG annotation emphasized gene functions centered on amino acid transport and metabolism, as well as inorganic ion transport and metabolism. GO annotation highlighted functions such as integral membrane components, oxidation-reduction processes and related enzyme activities. KEGG annotation predominantly indicated metabolism-related genes. The strain’s genome featured multiple As metabolism-related genes, including,,,,,,and. Hydroponic experiments exhibited the strain’s robust capability to reduce As(Ⅴ) to As(Ⅲ), manifesting a reduction rate of 40.1% after 3 days of incubation. In conclusion, through promoting As bioreduction and being in combination with hyperaccumulator, the EEELCW01 could be expected to conduct for the remediation of As-contaminated environments.
arsenic; iron oxidation bacteria(FeOB); arsenic transformation; genome analysis; function prediction
X172
A
1007–1032(2023)04–0428–08
10.13331/j.cnki.jhau.2023.04.008
2022–05–26
2023–08–01
国家自然科学基金项目(42177392)
熊潇然(1996—),男,云南大理人,硕士研究生,主要从事土壤重金属修复研究,1433859346@qq.com;*通信作者,潘炜松,博士,副教授,主要从事土壤微生物修复研究,joux19@163.com
熊潇然,邹奇,吴川,夏礼兵,潘炜松.铁氧化菌菌株EEELCW01的基因组分析及砷转化功能[J].湖南农业大学学报(自然科学版),2023,49(4):428–435.
XIONG X R,ZOU Q,WU C,XIA L B,PAN W S.Genomic analysis and arsenic transformation of iron-oxidizing strain EEELCW01[J].Journal of Hunan Agricultural University(Natural Sciences),2023,49(4):428–435.
http://xb.hunau.edu.cn

责任编辑:邹慧玲
英文编辑:柳正

