POU1F1基因SNP位点与尼罗罗非鱼体质量和形态性状的相关性
高风英,佟延南,曹建萌,刘志刚,王 淼,衣萌萌,可小丽,卢迈新,朱 海
(1.中国水产科学研究院珠江水产研究所/农业部热带亚热带水产资源利用与养殖重点实验室,广东 广州 510380;2.海南省海洋与渔业科学院,海南 海口 570206)
垂体特异性正转录因子1(POU1F1)(亦称PIT-1 或GHF-1)是一种在垂体前叶中表达的组织特异性转录因子[1,2],在生长激素(GH)和催乳素(PRL)基因的转录调节中有一定作用[3,4],还参与激活促甲状腺激素b 亚基(TSHb)、POU1F1基因[5-6]和生长激素释放激素受体基因[7],对促体细胞、促乳细胞以及促甲状腺细胞的分化、繁殖和生存亦至关重要[5],是有育种潜力的候选基因,值得在罗非鱼中深入研究。此外,由于POU1F1的下游靶基因和调控细胞在物理生长中起着直接作用,POU1F1中的一些SNP 与畜禽的生长性状密切相关[8-13]。在水产领域,仅杜芳芳等[14]对大口黑鲈(Micropterus salmoides)POU1F1基因多态性与生长相关性状关联性进行了研究,获得了生长相关基因型。
目前,已广泛实施分子标记辅助育种(Marker Assisted Selection,MAS),以改善水产动物生产性能[15-16]。罗非鱼是我国重要的淡水养殖鱼类之一,以尼罗罗非鱼为主。据《2022 中国渔业统计年鉴》,2020 和2021 年我国罗非鱼产量超过世界总产量的30%,在国民经济中占有重要地位,罗非鱼的选育研究有重要意义。罗非鱼的选育目标主要是生长速度。尼罗罗非鱼引入我国后,经多年的选育,不同选育群体间因选育技术不同,生长速度不同。为继续选育尼罗罗非鱼,筛选生长相关分子标记以辅助选育非常必要。POU1F1基因多态性与家畜的生长和肉质性状相关,为探索罗非鱼中是否存在与体质量以及全长、体长、头长、体高、体宽等形态性状相关的POU1F1多态位点,本研究选取POU1F1基因,在尼罗罗非鱼高要亲代群体中筛选SNP,并与生长数据相结合,筛选与体质量和形态性状相关的SNP,并将这些SNP在高要子代群体、番禺群体以及海南群体中进行普适性验证,旨在为罗非鱼以生长性状为目标的选育提供有效分子标记。
1 材料与方法
1.1 材料
筛选SNP 位点样品:尼罗罗非鱼(Oreochromis niloticus)吉富品系,取自珠江水产研究所水产良种基地,40 尾鱼的鳍条样本,置20 ℃无水乙醇中保存。
体质量和形态性状关联分析样品:尼罗罗非鱼吉富品系高要亲代及子代群体取自珠江水产研究所水产良种基地,“广特超”新吉富罗非鱼(番禺群体)取自广东罗非鱼良种场,尼罗罗非鱼海南群体取自海南省水产科学院。高要亲代群体为2020 年的秋苗,取1 000 尾同塘养殖越冬至次年7 月;高要子代群体为2021 年用传统土塘繁殖的春苗,取200尾同水泥池养殖3个月(经激素处理,全雄);番禺群体为2021 年春苗(经激素处理,全雄),取200 尾,同水泥池养殖3个月;海南群体为2021年春苗1万尾,放至1/3 hm2的土塘中养殖3 个月(未经激素处理的正常雌雄群体)。取高要群体162 尾,番禺群体175尾,分别测量体质量、全长、体长、头长、提高、体宽,海南群体300尾,测量体质量。
1.2 方法
1.2.1 基因组DNA 的提取 采用磁珠法基因组DNA 提取试剂盒(NanoMagBio)提取基因组DNA,具体方法步骤参照试剂盒说明书。用琼脂糖凝胶电泳检测基因组DNA质量。
1.2.2POU1F1基因SNP 位点的筛查 根据POU1F1基因(GenBank accession No.XM_019352661,NC_031987)3ʹ区域序列,利用Primer Primer 5.0 软件设计2 对上下游引物:POU1F1-3end_1-F(15631-15655)(CGTACAAAATATGCCATTTACCAAG)、POU1F1-3end_1-R(16976-16995)(ACTTTATCAAGGCAGCGACG),POU1F1-3end_2-F(16749-16772)(GCTTTAACCAA ATGAATGTAGCTG)、POU1F1-3end_2-R(18139-18160)(TTTCAGTCTCCACTTTATCCCC),以及测序引物POU1F1-3end_2-seqf1(GAGTGGTTTTCACTGTGAC)。以上述高要亲代群体中40个个体的DNA为模板,进行PCR 扩增。反应体系(30µL):2×Taq PCR Master Mix 15µL,基因组DNA 1µL(约20 ng),10 pmol/µL上下游引物各1µL,ddH2O 12µL。反应条件:95 ℃5 min;95 ℃30 s、55 ℃30 s、72 ℃45 s,35 个循环;72 ℃5 min,16 ℃1 min,PCR 产物4 ℃下保存。条带符合理论目的大小的PCR 产物送Sanger 测序,测序平台为ABI 公司的3730xl DNA Analyzer 测序仪。2 对引物分别做双向测序后正反向拼接,且两段检测区域有重叠,以保证2 对引物测通全长。由于尾端有POLY 结构,因此多设计一条加测引物pou1f1-3end_2-seqf1,以避开POLY 结构。将测序良好的全长序列在SeqMan 软件中进行多序列比对,筛查POU1F1基因中的SNP位点。
1.2.3 POU1F1 基因SNP 位点的分型分析 采用
1.2.2 中PCR 产物测序法分析SNP 位点分型。反应体系和反应条件同1.2.2.。共分析3 个群体:首先分析高要亲代群体213 尾的SNP 位点分型,并分析SNP 位点基因型与体质量和形态性状的相关性,将获得的与体质量和形态性状相关的SNP 位点在高要子代群体(162 尾)以及番禺群体(175 尾)中进行分型分析,分析SNP 位点基因型(下文基因型中“I”代表TC 插入、CC 插入、TTTG 插入、C 插入、CTC 插入、T 插入;“D”代表缺失)与体质量和形态性状的相关性,验证亲子代群体间体质量和形态性状相关SNP 位点的可重复性,以及不同群体间的可重复性。
1.2.4POU1F1基因SNP 位点的分型验证 将与高要亲代群体、高要子代群体以及番禺群体体质量和形态性状相关的位点,在海南群体中进行扩增,并用SeqMan 软件统计基因型,分析SNP 位点基因型与体质量和形态性状的相关性,从而进一步验证所获得的体质量和形态性状相关SNP 位点的可重复性。
1.2.5 遗传信息统计 利用PIC-CALC 0.6 分析SNP位点的多态信息含量(polymorphism information content,PIC)。利用Popgene 3.2 进行哈迪-温伯格平衡(Hardy-Weinberg equilibrium,HWE)状态的卡方检验,并计算观测杂合度(Ho)、期望杂合度(He))、有效等位基因数(Ne)、基因频率及等位基因频率。采用HaploView 4.2 软件对SNP 位点进行连锁不平衡分析和单倍块构建。利用单倍型手动分析出双倍型。
1.3 数据的关联分析
采用SPSS17 软件一般线性模型中的多元方差分析模块分析SNP 位点不同基因型与全长、体长、头长、体高、体宽、体质量等6个生长性状的相关性。数据以平均值±标准差形式表达,差异显著性水平α=0.05。
2 结果分析
2.1 POU1F1基因部分序列SNP位点
将PCR 扩增获得的2 341 bp片段(登录号:NC_031987)进行序列比对,获得28个SNP位点(表1)。
2.2 各群体中POU1F1基因SNP位点的遗传多样性
2.2.1 高要亲代群体 表2 可见:高要亲代群体POU1F1基因28 个SNP 位点的多样性较为丰富,有效等位基因数(Ne)、观测杂合度(Ho)、期望杂合度(He)平均值分别为1.359 3、0.240 7、0.156 6;多态信息含量(PIC)平均为0.298,有6 个位点低度多态(PIC <0.25),20 个位点中度多态(0.25 <PIC <0.50),2 个位点高度多态。位点S1(A-198G)、S2(C-201T)、S4(A-469T)、S6(A-881G)、S7(A-888G) 在 群体中符合Hardy-Weinberg 平衡定律(下称“H-W 平衡”),其余23个位点均偏离H-W平衡。
2.2.2 高要子代群体 表3 可见:高要子代群体POU1F1基因12 个SNP 位点的多样性比较丰富,Ne、Ho、He平均值分别为1.1873、0.1542、0.1556;PIC平均为0.155,12个位点均低度多态(PIC <0.25);12个位点在群体中均符合H-W平衡。

表3 POU1F1基因12个SNP位点在尼罗罗非鱼高要子代群体的遗传信息Table 3 Genetic information of twelve SNP loci of POU1F1 gene in Gaoyao offspring population of Nile tilapia
2.2.3 番禺群体 表4 可知:番禺群体POU1F1基因12 个SNP 位点的多样性比较丰富,Ne、Ho、He平均值分别为1.2643、0.2038、0.2044;PIC 平均为0.182,有10 个位点低度多态(PIC <0.25),2 个位点高度多态;有11 个位点在群体中均符合H-W 平衡,有1 个位点偏离H-W平衡。

表4 POU1F1基因12个SNP位点在尼罗罗非鱼番禺群体的遗传信息Table 4 Genetic information of twelve SNP loci of POU1F1 gene in Panyu population of Nile tilapia
2.2.4 海南群体 表5 表明:海南群体雌性个体POU1F1基因6 个SNP 位点的多样性比较丰富,Ne、Ho、He平均值分别为1.2918、0.1933、0.2201;PIC 平均为0.202,有5 个位点低度多态(PIC <0.25);有3个位点在群体中均符合H-W 平衡,有3 个位点偏离H-W平衡。
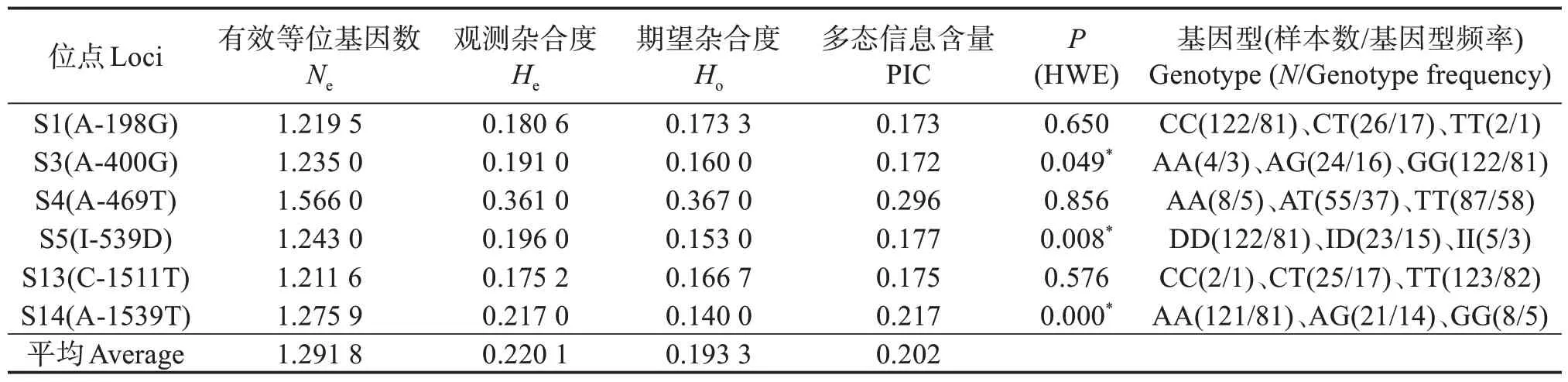
表5 POU1F1基因3个SNP位点在尼罗罗非鱼海南雌性群体的遗传信息Table 5 Genetic information of 3 SNP loci of POU1F1 gene in Hainan female population of Nile tilapia
表6表明:海南群体雄性个体POU1F1基因6个SNP 位点的多样性比较丰富,Ne、Ho、He平均值分别为1.2434、0.1767、0.1936;PIC 平均为0.185,6 个位点均低度多态(PIC <0.25);有5 个位点在群体中符合H-W平衡(表6),有1个位点偏离H-W平衡。
2.3 POU1F1基因SNP位点与各群体体质量和形态性状的相关性
2.3.1 高要亲代群体 表7 表明:POU1F1基因S3(A-400G)位点的AA 型个体与AG、GG 型个体在头长上有显著差异。S4(A-469T)位点TT、AT 型与AA型在体质量与全长上均有显著差异。S5(I-539D)位点DD 型和ID 型与II 型在体宽上有显著差异。S6(A-881G)位点AA 型与AG、GG 型在头长上存在显著差异。S7(A-888G)位点的GG 型与AA、AG 型在头长上存在显著差异。S12(C-1365T)位点的CT 与TT型在体质量和全长、体高、体宽上存在显著差异,该位点CC 与TT 型在体长上有显著差异,CC 与CT型在头长上有显著差异。差异显著性水平均为0.05。

表7 POU1F1基因中与尼罗罗非鱼高要亲代群体体质量或形态性状显著相关的6个位点Table 7 6 SNP loci in POU1F1 gene significantly associated with body mass or morphological traits of Gaoyao parent population of Nile tilapia
2.3.2 高要子代群体 将高要亲代群体中筛选出的与体质量和形态相关联的SNP 位点,在高要子代群体中进行分型分析,这些SNP 位点各基因型与高要子代群体体质量和形态性状关联分析表明:S3(A-400G)位点的AA型个体与AG、GG型个体间在体质量、全长、体长、头长、体高、体宽方面均有显著差异;AA 型个体各指标平均值均低于AG、GG 型个体。S5(I-539D)位点的DD 型个体与ID、II型个体间在体质量、全长、体长、头长、体高、体宽方面均存在显著差异;S11(I-1358D)位点ID 型与II 型在体质量和体高方面有显著差异,ID 型个体体质量和体高显著高于TT 型;S13(C-1511T)位点CC 型与CT、TT 型个体间在体质量、体长、头长、体高、体宽方面均有显著差异,CC 型个体平均值均高于CT、TT 型;S14(A-1539T)位点AT 型与TT 型个体间在体质量和体高方面有显著差异,AT 型个体的体质量、体高均值均高于TT型(表8)。
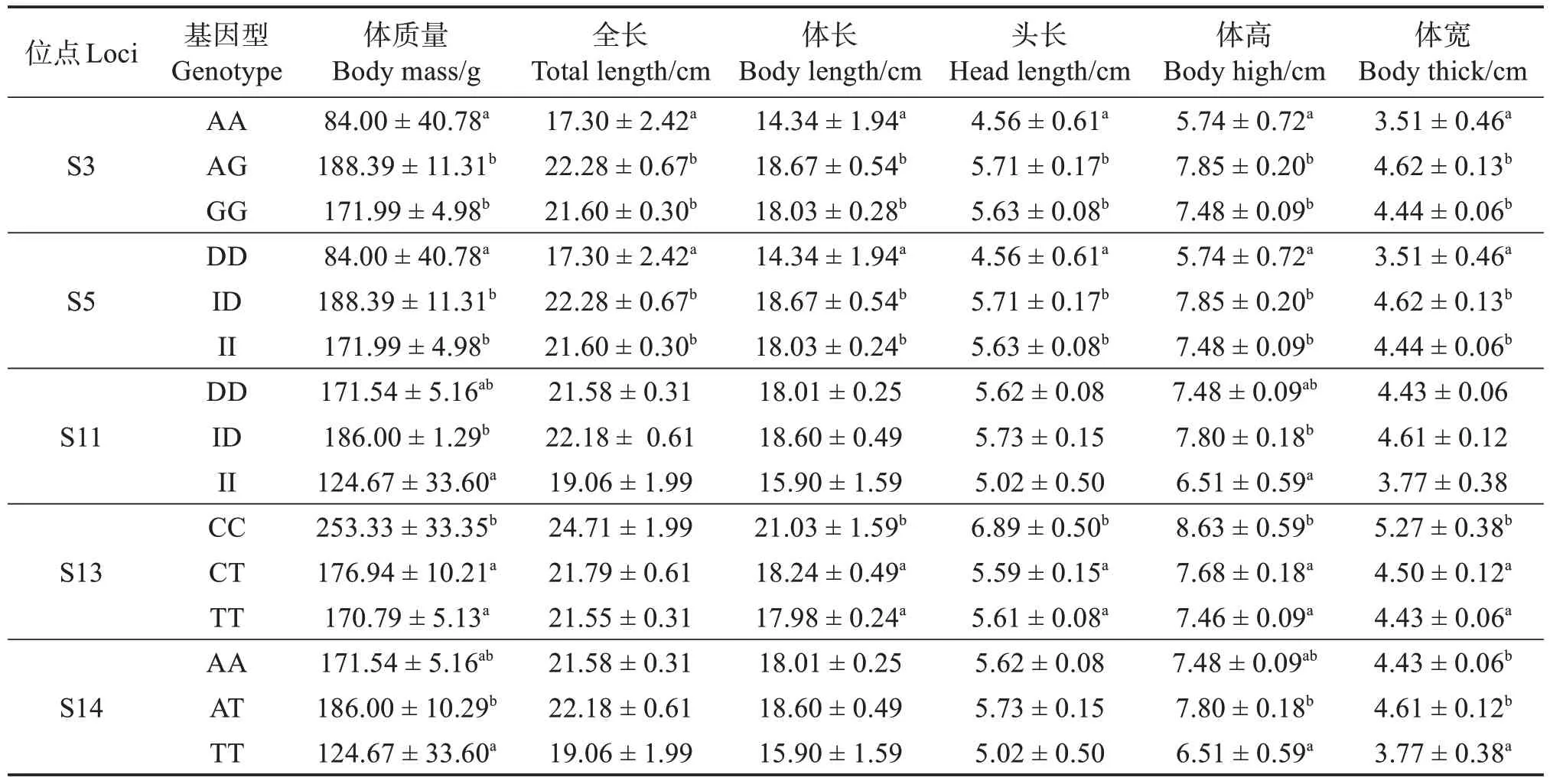
表8 POU1F1基因中与尼罗罗非鱼高要子代群体体质量或形态性状显著相关的5个位点Table 8 5 SNP loci in POU1F1 gene significantly associated with body mass or morphological traits of Gaoyao offspring population of Nile tilapia
2.3.3 番禺群体POU1F1基因SNP 位点与番禺群体体质量和形态性状关联分析表明,12 个SNP 位点与番禺群体体质量和形态性状均无关联(数据略)。
2.3.4 海南群体POU1F1基因SNP 位点与海南雌性群体体质量性状关联分析表明,S4(A-469T)位点与海南雌性群体体质量具有相关性,AA 型个体体质量明显高于AT、TT型(表9)。

表9 POU1F1基因中各SNP位点与尼罗罗非鱼海南雌、雄性群体体质量性状的关联分析Table 9 Association analysis of SNPs in POU1F1 gene with body mass traits of Hainan female and male population of Nile tilapia
表9 表明,海南雌性群体S3(A-400G)和S5(I-539D)位点与体质量性状显著相关,S3(A-400G)位点的AA 型个体体质量显著高于AG、GG 型个体,S5(I-539D)位点的DD型个体体质量显著高于ID、II型个体。
2.4 POU1F1基因双倍型与各群体体质量和形态性状的关联分析
2.4.1 高要亲代群体 将27 个SNPs 位点进行单倍型分析,得出双倍型(图1,表10),除去出现频率低于3%的双倍型,将其余5种双倍型与体质量和形态性状进行关联性分析,结果表明:双倍型D4、D7 个体的体宽显著高于双倍型D8 的个体。在实际的育种应用中,应选择D4、D7双倍型个体,以增加体宽。

图1 尼罗罗非鱼POU1F1基因SNPs在高要亲代群体中的连锁不平衡分析Fig.1 Map of pair-wise LD between POU1F1 SNPs in Gaoyao parent populations of Nile tilapia

表10 POU1F1基因双倍型与尼罗罗非鱼高要亲代群体体质量和形态性状关联分析Table 10 Correlation analysis between body mass and morphological traits and diplotype in the POU1F1 gene in Gaoyao parent population of Nile tilapia
2.4.2 高要子代群体 将12 个SNPs 位点进行单倍型分析,利用单倍型分析出双倍型,除去出现频率低于3%的双倍型,将其余2种双倍型与体质量和形态性状进行关联性分析(表11)。结果表明,2 种双倍型与高要子代群体体质量和形态性状均无关联。

表11 POU1F1基因双倍型与尼罗罗非鱼高要子代群体体质量和形态性状关联分析Table 11 Correlation analysis between body mass and morphological traits and diplotype in the POU1F1 gene in Gaoyao offspring population of Nile tilapia
2.4.3 番禺群体 将12 个SNPs 位点进行单倍型分析,利用单倍型分析出双倍型,除去出现频率低于3%的双倍型,将其余3种双倍型与体质量和形态性状进行关联性分析发现,3 种双倍型与番禺群体体质量和形态性状均无关联(表12)。

表12 POU1F1基因双倍型与尼罗罗非鱼番禺群体体质量和形态性状关联分析Table 12 Correlation analysis between body mass and morphological traits and diplotype in POU1F1 gene in Panyu population of Nile tilapia
2.4.4 海南群体 6 个SNP 位点在雄性群体中获得的双倍型,除去出现频率低于3%的双倍型,剩余4种双倍型。关联性分析发现,4 种双倍型与海南雄性群体体质量性状均无关联(表13)。

表13 POU1F1基因双倍型与尼罗罗非鱼海南雄性群体生长性状关联分析Table 13 Correlation analysis between body mass and diplotype in POU1F1 gene in Hainan male population of Nile tilapia
6 个SNP 位点在雌性群体中获得的双倍型,除去出现频率低于3%的双倍型,剩余5种双倍型。关联性分析发现,双倍型为D6 的个体体质量显著高于双倍型为D2的个体(表14,图2)。

图2 尼罗罗非鱼POU1F1基因SNPs在海南雌性群体中的连锁不平衡分析Fig.2 Map of pair-wise LD between POU1F1 SNPs in Hainan female population of Nile tilapia
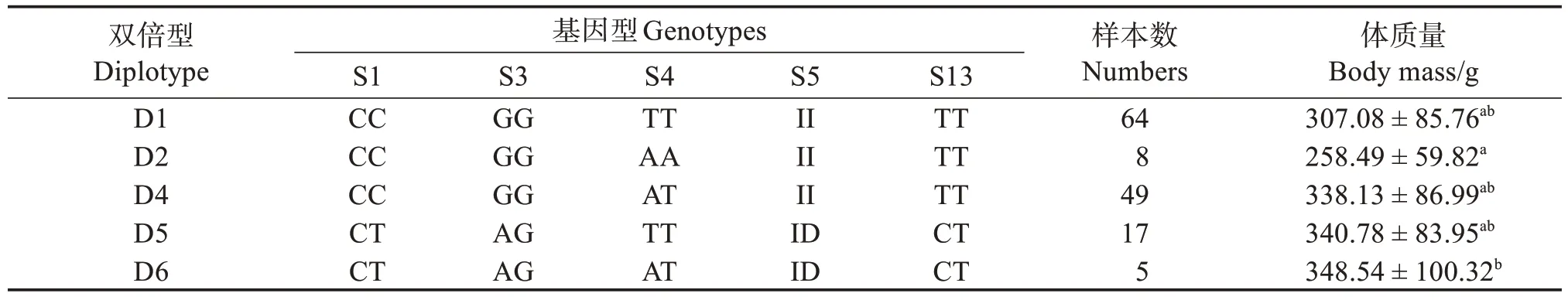
表14 POU1F1基因双倍型与尼罗罗非鱼海南雌性群体体质量性状关联分析Table 14 Correlation analysis between body mass traits and diplotype in the POU1F1 gene in Hainan female population of Nile tilapia
3 讨论
3.1 POU1F1多态性
单基因的遗传多样性可反映某群体的遗传多样性,某个群体的遗传多样性比较低,则在某个基因上可反映出来[17],本研究从单基因多态性方面分析群体遗传多样性,有一定实用性。
等位基因数、杂合度和多态信息含量等遗传参数均可反映直接反映群体的基因丰富度和遗传多样性[18]。本研究的高要子代群体Ne、Ho两个多样性指标与亲代群体相当,但He和PIC 低于亲代群体。可能由子代群体分析中所采用的SNP 位点数少于亲代群体导致,子代群体分析只采用了与亲代群体生长相关的位点。高要亲代群体PIC 均值为0.298,番禺群体为0.182,海南雌性群体为0.202,均稍低于王春晓等2015 年对这两个群体在GHSR基因中检测到的多样性(0.278,0.310)[19]。这可能是因为本研究所用的高要群体和番禺群体经过几年的选育后多样性有所下降,也可能是因为不同基因的SNP 多样性有所不同。本研究表明,各罗非鱼群体具有较高的遗传多样性,可在此基础上继续进行良种选育。
3.2 生长相关位点的筛选
本研究筛选生长相关SNP 位点所用的区域均位于POU1F1基因的3ʹ端非编码区。这些位于非编码区的SNP 位点,不参与蛋白编码,也不能改变蛋白序列及结构,因此这些基因座对性状的影响可能不直接,但这些位于非编码区的突变位点可能与负责改变蛋白序列及结构的位点连锁不平衡(具有连锁不平衡的位点可影响动物的生产性状[20])。单倍型或双倍型与性状进行关联分析,解决了单个标记分子存在的位点检测和统计效率低等问题,从而能更真实反映所分析位点与性状的关联性[21]。王春晓等[19]在尼罗罗非鱼GHSR基因中筛选的3 个SNP位点均与生长性状无关联,但3 个位点组成的双倍型与生长性状显著相关。本研究在高要亲代群体中获得体宽相关双倍型2 个,在海南雌性群体中获得与体质量相关的双倍型1个;但在高要子代群体、海南雄性群体中获得与生长性状相关的SNP 位点,而未获得与生长性状相关双倍型:SNP 位点之间出现了拮抗。这与李红霞等[22]对建鲤(Cyprinus carpiovar.Jian)鸟氨酸脱酶基因多态性与生长性状的关联分析结果类似,不同位点之间可能存在拮抗或协同的互作。
3.3 生长相关SNP位点在各群体中的普适性
本研究在高要亲代群体中获得的体质量和形态相关位点,与子代中筛选到的相关位点部分重合,但是亲子代群体与重合位点关联的体质量和形态性状有所不同,如:亲代群体位点S3 与头长显著相关,S4 与体质量、全长相关,S5 与体宽显著相关,S11 与头长显著相关;但在子代群体中,位点S3 和S5与体质量、全长、体长、头长、体高、体宽均显著相关,S11 与体质量和体高显著相关。这可能是亲代群体与子代群体生长状态不同导致的;也可能是因为鱼类生长性状是受多种基因控制的数量性状,主效基因和微效基因在不同世代中会出现分离或整合现象,从而导致筛选的生长相关SNP 位点在不同世代之间的适用性较差[23];还可能是本研究子代群体采样数量少于亲本导致的,增加子代采样数量,有可能提高亲代子代之间生长相关位点的契合度。本研究在高要子代群体中获得的SNP 位点与番禺群体中相关SNP 位点没有重叠,在番禺群体未筛选到体质量和形态性状相关位点。高要子代中与生长性状显著相关的SNP 位点,在番禺群体中各基因型的体质量数据亦有部分差异,只是在统计学上未达到显著性水平。这可能由取样数量导致。影响标记-性状连锁分析准确性的一个重要因素是群体数量;但目前对用于标记-性状连锁分析的群体数量须达到什么标准筛选出的标记才可信,尚无明确的依据,因而并不能说在另外一个群体中未得到验证的关联标记不可信[24]。与高要子代群体生长性状相关的这些位点在海南群体中进行进一步验证表明:仅S4 位点与海南雌性群体体质量显著相关,S3 和S5 位点与雄性群体体质量相关,其余位点与体质量性状均不具有相关性。这可能是因为不同群体的遗传背景差异会导致筛选到的生长相关SNP 位点在不同群体中的关联性急剧下降。
本实验获得的与高要亲代群体生长相关位点,在高要亲代与子代之间普适性较弱,可能是因为取样数量不够大,导致亲代、子代之间生长相关位点有些差异;这些与高要亲代群体生长相关SNP 位点在不同群体间的普适性较差,可能是不同群体间遗传背景的差异导致,也可能是因为取样数量不够大导致。扩大群体取样数量,这些生长相关SNP 标记在不同群体之间的普适性可能就会提高。

