鼠伤寒沙门菌PipD蛋白结构及抗原表位的生物信息学分析
宋晓蕊 刘玉春 丁聪 贾蕊 李瑞静 马嘉悦 李利锋


摘要:目的 用生物信息学方法预测分析沙门菌PipD蛋白的结构和抗原表位,为沙门菌相关疾病的诊疗和疫苗的研发提供理论依据。方法 通过NCBI数据库获得沙门菌典型致病血清型:鼠伤寒沙门菌(Salmonella typhimurium)ATCC 14028S标准菌株STM14_1240基因的核苷酸序列及其编码蛋白PipD 的氨基酸序列,运用ProtParam、ProtScale、SignalP 4.1 Server、TMHMM Serverv.2.0、Netphos 3.1 Server、NetNGlyc-1.0、NCBI BLAST、SOMPA、SWISSMODEL、ABCpred、SYFPEITHI和UniProt等生物信息学工具分别对鼠伤寒沙门菌PipD蛋白的理化性质、亲疏水特性、信号肽、跨膜区、磷酸化位点、糖基化位点、结构域、二级结构、三级结构、B细胞抗原表位、T细胞抗原表位及蛋白同源性进行预测分析。结果 PipD是由520个氨基酸组成的亲水蛋白,分子式为C2608H3963N721O780S16,理论相对分子质量为58.4 kDa,理论等电点为7.34,脂溶指数为67.81,平均亲水系数为-0.508。PipD蛋白无信号肽序列,在其35~57氨基酸残基位置含1个长度为23个氨基酸的跨膜螺旋,属于跨膜蛋白。预测PipD蛋白含有 57个磷酸化位点和1个糖基化位点。PipD蛋白二级结构以无规则卷曲居多,占36.35%,其次为α螺旋、β折叠、β转角分别占15.77%、3.27%、44.62%。预测PipD蛋白可形成22个B细胞优势抗原表位,17个CTL细胞优势表位和30个Th细胞优势表位。PipD蛋白与人类基因BCLA3、SCRN2和MRPL4的编码蛋白同源性分别为29%、24.6%和31.8%,同源性较低。结论 PipD蛋白为亲水蛋白,热稳定性好;存在多个B细胞和T细胞抗原表位,与人类宿主蛋白同源性低,不易发生交叉免疫反应,可作为鼠伤寒沙门菌血清学诊断和疫苗候选蛋白。
关键词:鼠伤寒沙门菌;PipD;生物信息;蛋白结构;抗原表位
中图分类号:S852.6 文献标识码:A DOI:10.3969/j.issn.1006-1959.2024.04.001
文章編号:1006-1959(2024)04-0001-07
Bioinformatics Analysis of PipD Protein Structure and Epitope of Salmonella Typhimurium PipD
SONG Xiao-rui1,2,LIU Yu-chun1,DING Cong1,JIA Rui1,LI Rui-jing1,MA Jia-yue1,LI Li-feng1
(Henan International Joint Laboratory of Children's Infectious Diseases1,Department of Otorhinolaryngology Head and Neck Surgery2,Children's Hospital Affiliated to Zhengzhou University,Zhengzhou 450000,Henan,China)
Abstract:Objective To predict and analyze the structure and antigenic epitopes of Salmonella PipD protein by bioinformatics methods, and to provide a theoretical basis for the diagnosis and treatment of Salmonella-related diseases and the development of vaccines.Methods The nucleotide sequence of Salmonella typhimurium ATCC 14028S standard strain STM14_1240 gene and the amino acid sequence of its encoded protein PipD were obtained by NCBI database. Bioinformatics tools such as ProtParam, ProtScale, SignalP 4.1 Server, TMHMM Serverv.2.0, Netphos 3.1 Server, NetNGlyc-1.0, NCBI BLAST, SOMPA, SWISSMODEL, ABCpred, SYFPEITHI and UniProt were used to predict and analyze the physicochemical properties, hydrophilicity and hydrophobicity, signal peptide, transmembrane region, phosphorylation site, glycosylation site, domain, secondary structure, tertiary structure, B cell epitope, T cell epitope and protein homology of Salmonella typhimurium PipD protein.Results PipD was a hydrophilic protein composed of 520 amino acids, the molecular formula was C2608H3963N721O780S16, the theoretical relative molecular mass was 58.4 kDa, the theoretical isoelectric point was 7.34, the fat solubility index was 67.81, and the average hydrophilic coefficient was -0.508. PipD protein had no signal peptide sequence, and contained a transmembrane helix with a length of 23 amino acids at its 35-57 amino acid residues, which belonged to transmembrane protein. It was predicted that PipD protein contained 57 phosphorylation sites and 1 glycosylation site. The secondary structure of PipD protein was mostly random coil, accounting for 36.35%, followed by α-helix, β-sheet and β-turn, accounting for 15.77%, 3.27% and 44.62%, respectively. It was predicted that PipD protein could form 22 B cell dominant epitopes, 17 CTL cell dominant epitopes and 30 Th cell dominant epitopes. The homology of PipD protein with human genes BCLA3, SCRN2 and MRPL4 was 29%, 24.6% and 31.8%, respectively.Conclusion PipD protein is a hydrophilic protein with good thermal stability. There are multiple B cell and T cell epitopes, which have low homology with human host proteins and are not prone to cross-immune reactions. It can be used as a candidate protein for serological diagnosis and vaccine of Salmonella typhimurium.
Key words:Salmonella typhimurium;PipD;Bioinformatics;Protein structure;Antigenic epitope
沙门菌(Salmonella)是一种重要的人畜共患革兰氏阴性肠道病原菌,主要通过污染的水源和食物传播,可感染人和猪、牛等多种哺乳动物,在宿主中引起自限性肠胃炎和菌血症等多种疾病。有关沙门菌感染的报道逐年增多,加重了预防和控制的负担[1,2]。沙门菌引起系统性疾病的两个关键阶段包括侵袭肠上皮细胞和在巨噬细胞内生存复制。沙门菌的致病能力主要依赖于沙门致病岛(Salmonella pathogenicity islands,SPI)编码的毒力基因,SPI-1和SPI-2是沙门菌两个最重要的致病岛[3,4]。由SPI-1编码的三型分泌系统(type three secretion system,T3SS)-1 是沙门菌入侵肠道上皮细胞所必需的[5,6];由SPI-2编码的三型分泌系统T3SS-2是沙门菌在巨噬细胞中复制和系统性感染所必需的[7,8]。基于沙门致病岛编码基因在沙门菌致病性中的重要作用,本研究以沙门菌典型致病血清型:鼠伤寒沙门菌ATCC 14028S菌株为材料,应用多种生物信息学工具对鼠伤寒沙门菌SPI-5[9]上一个与鼠伤寒沙门菌引起的肠道炎症相关的毒力基因STM14_1240的编码蛋白PipD[10-12]进行预测分析,进一步明确其结构、抗原表位以及在鼠伤寒沙门菌致病过程中的可能功能,为沙门菌相关疾病的诊疗和疫苗研发提供理论依。
1资料与方法
1.1 PipD蛋白的基因组信息 通过NCBI GeneBank数据库获取鼠伤寒沙门菌(Salmonella enterica subsp. enterica serovar typhimurium)ATCC14028S标准菌株STM14_1240基因的核苷酸序列及其编码蛋白PipD 的氨基酸序列。PipD蛋白在GenBank数据库中的基因序列登录号为NC_016856.1,蛋白氨基酸序列登录号为WP_000760228.1。PipD蛋白编码基因STM14_1240全长1563 bp,位于鼠伤寒沙门菌14028S菌株基因组的3729722~3731284位置,编码520个氨基酸,GenBank数据库中PipD蛋白的FASTA格式氨基酸序列如下:
1.2 PipD蛋白的基本理化性质分析 运用Expasy-ProParam工具分析PipD蛋白的基本理化学性质;使用Expasy-ProScale工具分析PipD蛋白的亲疏水特性。
1.3 PipD蛋白的信号肽、跨膜区、磷酸化位点、糖基化位点和保守域分析 使用SignalP4.1 Server工具预测分析PipD蛋白的信号肽序列;使用TMHMM Serverv.2.0工具预测PipD蛋白的跨膜区;使用Netphos 3.1 Server工具预测PipD蛋白的翻译后磷酸化修饰位点;使用NetNGlyc -1.0工具预测PipD蛋白的糖基化位点;应用NCBI BLAST软件预测PipD蛋白的保守域。
1.4 PipD蛋白的结构预测 使用SOPMA工具分析PipD蛋白的二级结构及组成;使用SWISS MODEL工具对PipD蛋白的三级结构进行分析和建模。
1.5 PipD蛋白的抗原表位预测 使用ABCpred和SYFPEITHI工具预测PipD蛋白的B细胞和T细胞抗原表位。
1.6 PipD蛋白的同源性分析 使用UniProt网站分析PipD蛋白的同源性。
2结果
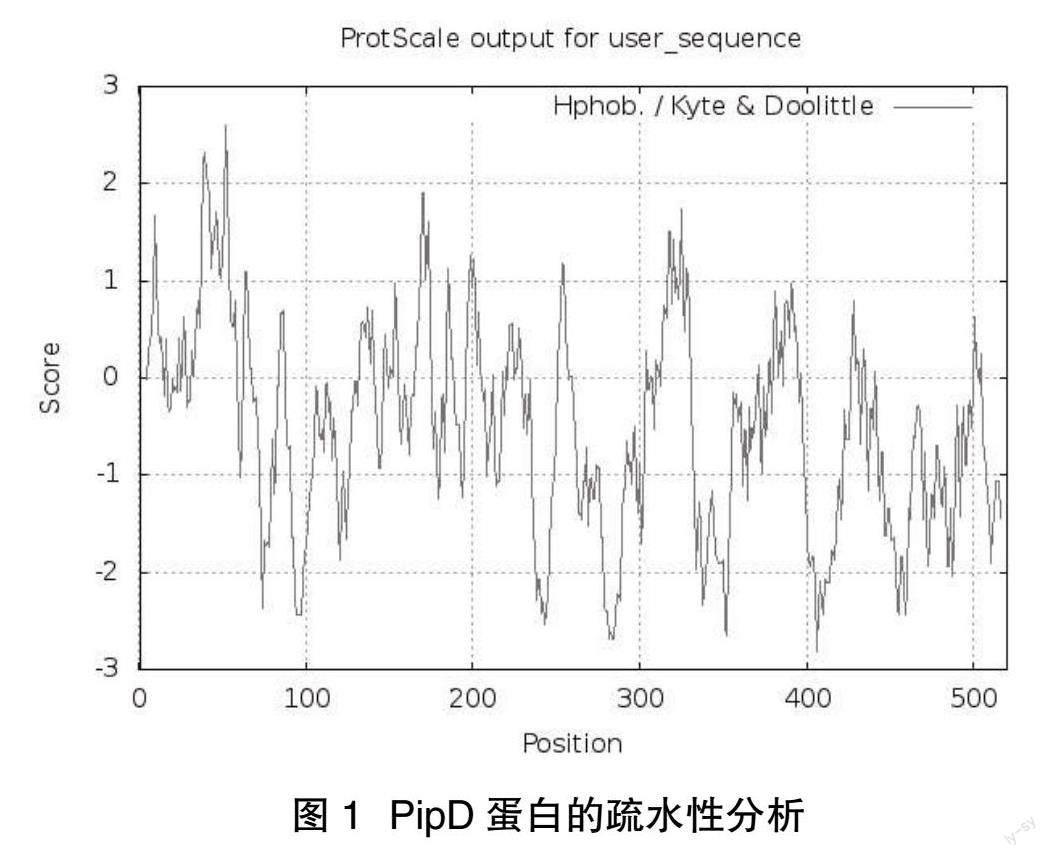
2.1 PipD蛋白的基本理化性质分析 使用Expasy-ProParam工具对PipD蛋白的基本理化性质进行预测分析,结果显示PipD蛋白的分子式为C2608H3963N721O780S16,由8088个原子组成,理論相对分子质量为58.4 kDa,理论等电点为7.34。在PipD蛋白的氨基酸组成中,丙氨酸(Ala)、苏氨酸(Thr)、丝氨酸(Ser)占比较高,分别占9.8%、7.5%和6.5%;其中带负电荷的氨基酸(天冬氨酸+谷氨酸)和带正电荷的氨基酸(精氨酸+赖氨酸)数量均为47个。PipD蛋白在哺乳动物网织红细胞体外实验中的半衰期为30 h,脂肪族指数为67.81,不稳定指数为43.37,为不稳定蛋白。通过Expasy-ProScale工具对PipD蛋白的亲疏水特性进行分析显示,PipD蛋白总平均亲水性为-0.508,氨基酸得分最大值为2.6,最小值为-2.822,属于具有良好亲水性的蛋白,见图1。
2.2 PipD蛋白的信号肽、跨膜区、磷酸化位点、糖基化位点和保守域分析 使用SignalP 4.1 Server对PipD蛋白进行分析得到D值为0.192,表明PipD蛋白不含信号肽序列(图2)。使用TMHMM Serverv.2.0工具预测在PipD蛋白第35~57位氨基酸处含有1个长度为23的跨膜螺旋(图3)。Netphos 3.1 Server预测PipD蛋白有22个丝氨酸磷酸化位点、22个苏氨酸磷酸化位点和13个酪氨酸磷酸化位点(图4)。NetNGlyc-1.0预测PipD蛋白含有1个糖基化位点,位于第101位,概率为63.49%(图5)。应用NCBI BLAST预测PipD蛋白包含PepD超家族、Ntn-hydrolase超家族结构域,属于PepD家族蛋白(图6)。
2.3 PipD蛋白的结构预测 使用SOPMA工具分析显示PipD蛋白含α螺旋(Hh)189个,占36.35%;β折叠(Ee)82个,占15.77%;β转角(Tt)17个,占3.27%;无规则卷曲(Cc)232个,占44.62%(图7)。运用Expasy-SWISS MODEL工具预测PipD蛋白三级结构并进行同源建模,模型GMQE评分为0.65,QMEAN值为0.68,表明该模型预测效果较好(图8)。
2.4 PipD蛋白的抗原表位预测 使用ABCpred软件预测PipD蛋白中存在22個潜在的B细胞抗原表位(分值>0.8),见表1。使用SYFPEITHI软件预测PipD蛋白含有17个限制性CTL表位(分值>20)和30个限制性Th表位(分值>20),见表2、表3。
2.5 PipD蛋白的同源性分析 在 UniProt网站中输入PipD蛋白的FASTA格式氨基酸序列,物种选择 Homo sapiens对PipD蛋白的同源性进行分析发现PipD蛋白与人类基因BCLA3、SCRN2和MRPL4编码的蛋白同源性分别为29%、24.6%和31.8%(图9),同源性较低,不易发生交叉免疫反应,表明其免疫原性较好,可作为设计疫苗的潜在蛋白。
3讨论
沙门菌是威胁人类健康的一个重要公共卫生问题,每年引起约9300万例感染[13],导致约15万人死亡[14],其中,鼠伤寒沙门菌是引发感染的最常见血清型[1]。毒力因子是近年来沙门菌研究的热点问题,探究沙门菌毒力蛋白的功能,提高沙门菌感染相关疾病的防治水平具有重要的公共卫生意义。
沙门菌的致病性与沙门致病岛编码的毒力基因有关[15],STM14_1240(pipD)是鼠伤寒沙门菌SPI-5上一个重要的毒力基因,编码T3SS-1的效应蛋白PipD,与鼠伤寒沙门菌引起肠道炎症相关[10]。通过检测pipD等毒力基因在鼠伤寒沙门菌分离株中的存在情况可评估沙门菌的潜在毒力及其导致持续感染的风险[16,17]。生物信息学分析通常基于生物信息数据库,通过生物信息学工具对目标基因和蛋白的结构和功能进行预测和分析,为致病机制研究和疫苗研发提供理论依据[18]。
本研究通过NCBI GeneBank数据库获取PipD蛋白的氨基酸序列及其编码基因的核苷酸序列,应用生物信息学工具预测和分析PipD蛋白的结构特性和功能,结果显示该蛋白理论相对分子质量为58.4 kDa,理论等电点为7.34。在PipD蛋白的氨基酸组成中,脂肪族氨基酸如丙氨酸、苏氨酸和丝氨酸等所占比例较高,分别为9.8%、7.5%和6.5%,提示该蛋白蛋白热稳定性较好。PipD蛋白的半衰期为30 h,提示该蛋白作为免疫抗原可在体内发挥稳定持久作用。亲疏水性分析显示PipD蛋白有多个较强的亲水区,有利于抗原表位的形成[19]。
生物信息学分析显示,PipD蛋白不含信号肽,在该蛋白第35~57氨基酸位置含1个长度为23个氨基酸的跨膜螺旋,其跨膜转运机制还不清楚。PipD蛋白存在多个潜在磷酸化位点,提示PipD蛋白可能受到磷酸化调控,参与细胞信号转导过程。预测PipD蛋白含有1个糖基化位点,提示该蛋白可作潜在标志物用于鼠伤寒沙门菌感染的诊断。
二级结构分析显示,PipD蛋白中无规则卷曲所占比例较大,其次为α-螺旋、β-折叠和β-转角,表明该蛋白具有良好的抗体嵌合性,易形成抗原表位。三级结构分析显示PipD蛋白的无规则卷曲大多位于蛋白质外层区域,提示PipD蛋白易在空间构象上形成抗原表位,有利于抗原和抗体相结合。抗原表位是抗原分子上能与相应抗体或淋巴细胞的表面受体结合从而激活机体免疫应答反应的特殊化学集团[20]。ABCpred软件和SYFPEITHI软件预测显示PipD蛋白共有22个B细胞优势表位,17个 CTL细胞优势表位和30个Th细胞优势表位,且与人类宿主蛋白同源性较低,不易发生交叉免疫反应,表明该蛋白免疫原性好,可作为潜在的鼠伤寒沙门菌疫苗候选蛋白,也可用于血清学诊断以提高沙门菌感染检查的灵敏度和特异性。
综上所述,PipD蛋白为亲水性蛋白,含有B、T细胞抗原表位,即具有抗原性。本研究通过多种生物信息学工具对PipD蛋白的理化性质和结构功能进行预测和分析,可为pipD在鼠伤寒沙门菌致病过程中的可能功能提供更为深入的信息,为研发有效的鼠伤寒沙门菌疫苗提供新的靶点,也为研究鼠伤寒沙门菌的致病机理提供理论参考。
参考文献:
[1]Fierer J.Invasive Non-typhoidal Salmonella (iNTS) Infections[J].Clin Infect Dis,2022,75(4):732-738.
[2]Sun H,Wan Y,Du P,et al.The Epidemiology of Monophasic Salmonella Typhimurium[J].Foodborne Pathog Dis,2020,17(2):87-97.
[3]Dos SA,Ferrari RG,Conte-Junior CA.Virulence Factors in Salmonella Typhimurium: The Sagacity of a Bacterium[J].Curr Microbiol,2019,76(6):762-773.
[4]Hume PJ,Singh V,Davidson AC,et al.Swiss Army Pathogen: The Salmonella Entry Toolkit[J].Front Cell Infect Microbiol,2017,7:348.
[5]Lou L,Zhang P,Piao R,et al.Salmonella Pathogenicity Island 1 (SPI-1) and Its Complex Regulatory Network[J].Front Cell Infect Microbiol,2019,9:270.
[6]Egan F,Barret M,O'Gara F.The SPI-1-like Type III secretion system: more roles than you think[J].Front Plant Sci,2014,5:34.
[7]Cerny O,Holden DW.Salmonella SPI-2 type III secretion system-dependent inhibition of antigen presentation and T cell function[J].Immunol Lett,2019,215:35-39.
[8]Jennings E,Thurston T,Holden DW.Salmonella SPI-2 Type III Secretion System Effectors: Molecular Mechanisms And Physiological Consequences[J].Cell Host Microbe,2017,22(2):217-231.
[9]吕雪莲,周薇,刘松艳,等.肠炎沙门氏菌三种主要毒力岛基因缺失株的构建及其免疫效力研究[C]//第十届全国免疫学学术大会汇编.2015.
[10]Hughes LA,Shopland S,Wigley P,et al.Characterisation of Salmonella enterica serotype Typhimurium isolates from wild birds in northern England from 2005-2006[J].BMC Vet Res,2008,4:4.
[11]Chen Z,Bai J,Wang S,et al.Prevalence, Antimicrobial Resistance, Virulence Genes and Genetic Diversity of Salmonella Isolated from Retail Duck Meat in Southern China[J].Microorganisms,2020,8(3):444.
[12]Soubeiga AP,Kpoda DS,Compaoré MKA,et al.Molecular Characterization and the Antimicrobial Resistance Profile of Salmonella spp. Isolated from Ready-to-Eat Foods in Ouagadougou, Burkina Faso[J].International Journal of Microbiology,2022,2022:1-10.
[13]Stanaway JD,Parisi A,Sarkar K,et al.The global burden of non-typhoidal Salmonella invasive disease: a systematic analysis for the Global Burden of Disease Study 2017[J].The Lancet Infectious Diseases,2019,19(12):1312-1324.
[14]Haselbeck AH,Panzner U,Im J,et al.Current perspectives on invasive nontyphoidal Salmonella disease[J].Curr Opin Infect Dis,2017,30(5):498-503.
[15]Azimi T,Zamirnasta M,Sani MA,et al.Molecular Mechanisms of Salmonella Effector Proteins: A Comprehensive Review[J].Infect Drug Resist,2020,13:11-26.
[16]Thung TY,Radu S,Mahyudin NA,et al.Prevalence, Virulence Genes and Antimicrobial Resistance Profiles of Salmonella Serovars from Retail Beef in Selangor, Malaysia[J].Front Microbiol,2017,8:2697.
[17]Ramtahal MA,Somboro AM,Amoako DG,et al.Molecular Epidemiology of Salmonella enterica in Poultry in South Africa Using the Farm-to-Fork Approach[J].International Journal of Microbiology,2022,2022:1-12.
[18]Canzoneri R,Lacunza E,Abba MC.Genomics and bioinformatics as pillars of precision medicine in oncology[J].Medicina (B Aires),2019,79(Spec 6/1):587-592.
[19]Polasa A,Tabari SH,Moradi M.Developing Efficient Transfer Free Energy Calculation Methods for Hydrophobicity Predictions[J].Biophysical Journal,2021,120(3):115a.
[20]El-Manzalawy Y,Dobbs D,Honavar VG.In Silico Prediction of Linear B-Cell Epitopes on Proteins[J].Methods Mol Biol,2017,1484:255-264.
收稿日期:2023-04-06;修回日期:2023-05-10
編辑/成森

