糖尿病肾病患者DNA甲基化表达谱分析
王晓楠 徐婷婷 赵婷婷 秦卫松
近年来糖尿病的发病率显著增加。糖尿病肾病(DN)已经成为中国继肾小球肾炎之后引起终末期肾病的第二大因素[1]。多种因素参与了DN的发病和进展,包括环境因素、基因背景以及表观遗传修饰等,其中表观遗传修饰近年来逐渐受到了关注和重视[2-6]。常见的表观遗传学修饰包括:DNA甲基化、组蛋白转录后修饰及非编码RNA等[4]。其中,DNA甲基化是目前研究最广泛的表观遗传学机制之一[7-8],是在DNA甲基转移酶的作用下,胞嘧啶-鸟嘌呤(CpG)二核苷酸中的胞嘧啶第5位碳原子接受一个甲基,被修饰为5-甲基胞嘧啶[8]。DNA甲基化能引起染色质结构、DNA构象和稳定性、DNA与蛋白质相互作用方式的改变,从而调控基因表达。
已有研究表明,与糖尿病非肾病患者相比,DN患者基因组甲基化整体水平存在差异[9],其中一些CpG位点甲基化水平的改变与DN的关系密切,表明DNA甲基化对DN的发生发展发挥重要作用。 但目前有关DN的DNA甲基化机制的研究所采取的标本主要是唾液或外周血单个核细胞DNA[10-11]。由于表观遗传机制具有明显的细胞特异性,有关DN肾组织和肾脏固有细胞的甲基化研究将更有利于揭示DNA甲基化参与DN发生发展的机制。
研究对象和方法
研究对象
纳入标准及分组 分为DN组、糖尿病非肾病组(DM组)和正常对照组,每组各3例。DN组入选标准:(1)符合WHO 1999年糖尿病的诊断标准;(2)经肾活检符合DN的病理特点。DM组选取在南京总医院泌尿外科因肾脏肿瘤接受肾摘除手术的肾功能正常且微量白蛋白尿<30 μg/mg的2型糖尿病患者,留取癌旁肾组织。正常对照组选取因肾脏肿瘤在南京总医院泌尿外科接受肾摘除手术的血糖正常患者,经实验室检查排除糖尿病、肾脏疾病及其他系统疾病,留取癌旁肾组织。所有受试者均为汉族人,彼此之间无亲缘关系。
排除标准 排除其他内分泌疾病、肝胆疾病、慢性肾炎、泌尿系感染、原发性高血压病等疾病;且在近期无糖尿病酮症酸中毒及其他急性并发症,无肾损害药物用药史。
主要试剂及仪器
试剂 TruSeq®DNA LT Sample Prep Kit v2(Illumina);TruSeq PE Cluster Kit(Illumina);TruSeq SBS Kit(Illumina);DNA微量提取试剂盒(Qiagen);亚硫酸盐转化试剂盒(Qiagen)等。
仪器 Hisq3000 高通量测序仪(Illumina);CBOT 簇生成仪(Illumina);冷冻离心机(Scan speed);浓缩仪 (Thermo);常温离心机(Thermo)等。
全基因组甲基化测序
肾组织DNA制备 取肾组织25 mg,切成小块状,置于离心管中,加入蛋白酶K 10 μl,56℃过夜孵育,按照DNA微量提取试剂盒(Cat#56304,QIAGEN,Germany)说明书,提取DNA。
DNA质检 取1 μl DNA使用荧光定量仪进行定量,根据定量结果,取50 ng DNA进行电泳检测,结束后在紫外透射光下观察。合格样本电泳结果为单一条带;如出现条带拖尾或弥散,说明DNA样本有降解,完整性不佳;如有杂带,说明含其他杂质。
WGBS(Whole Genome Bisulfite Sequencing)文库构建 取2.2 μg DNA(14 μl),用缓冲液稀释至86 μl,用超声使DNA 片段化,大小在200~500 bp;用末端修复缓冲液将DNA 末端补平;加A尾缓冲液至DNA中;加入连接缓冲液、接头,充分混匀;用2%的琼脂糖凝胶电泳筛选250~350 bp的DNA,按照胶回收试剂盒(Cat#28604,QIAGEN,Germany)对DNA进行回收;将回收到的DNA按照亚硫酸盐转化试剂盒(Cat#59104,QIAGEN,Germany)说明书对DNA进行重亚硫酸盐处理;最后进行PCR富集,使用荧光定量仪定量WGBS文库。
簇生成 将WGBS文库稀释至10 nmol/L;取4 μl 10 nmol/L文库,加1 μl 2 nmol/L NaOH,加15 μl Tris·Cl,混匀,室温放置5 min;取出6 μl,加入994 μl冷的杂交缓冲液;取140 μl上述溶液,放在八连管中,置于cBot的template栏中(cBot为成簇的仪器,template为成簇仪上面的放八连管的架子);开始簇生成。
Illumina Hiseq3000测序 使用Hiseq3000上机测序;将得到的原始数据转换成 Fastq格式;数据质量达到 Q30>80%(Q30为碱基质量评估值,指每个碱基错误的可能在1/1 000,所有质量达到Q30的碱基占总碱基量的80%)。
统计学方法采用SPSS 20.0统计软件进行数据处理,运用单因素方差分析进行比较,计量资料以均数±标准差表示,P<0.05为差异有统计学意义。
结 果
DN患者肾组织DNA的CpG甲基化水平升高所有样本的DNA经重亚硫酸盐处理后,未被甲基化的胞嘧啶转变为尿嘧啶,转化率均>99.92%;经全基因组甲基化测序一共得到604 829 312~1 029 008 460条原始序列数,去除低质量片段后,得到589 964 228~1 012 361 576条序列数,占原始序列数的比例为96.50%~98.38%。所有样本的唯一比对率为66.17%~78.00%。DN患者肾组织DNA的CpG甲基化水平为72.32±0.58%,高于糖尿病非肾病患者(71.84±0.92%)和正常对照(71.70±0.90%)。
DN患者肾组织DMR分析差异甲基化区域(differentially methylated regions,DMR)是指在多种样本(组织、细胞、个体等)中基因组里甲基化状态不相同的区域,是参与基因转录水平调控的功能性区域。DMR的判定条件:该区域中甲基化有差异的C碱基占总C碱基的比例大于50%;长度大于50 bp;DMR区域内至少含5个C碱基。根据全基因组甲基化测序的结果,筛选得到DN患者肾组织的DMR,在染色体的分布情况如图1所示。可见DMR在每条染色体上均有分布,在每条染色体上,DN患者与糖尿病非肾病患者之间的DMR数目均高于这两组患者与正常对照之间的DMR数目。
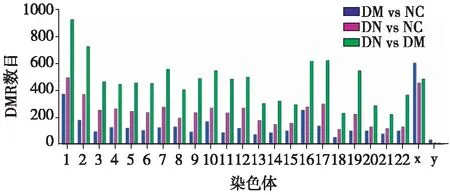
图1 DN患者肾组织DMR在各染色体的分布DMR:差异甲基化区域;DM:糖尿病非肾病组;DN:糖尿病肾病组;NC:正常对照组
将DN组与DM组进行比较,得到13 247个DMR,关联到7 379个差异甲基化基因(DMR-related genes,DMG)。其中,DN患者有10 802个DMR、6 246 个DMG的甲基化水平明显高于糖尿病非肾病患者;2 445个DMR、1 133个DMG的甲基化水平明显低于糖尿病非肾病患者(表1)。
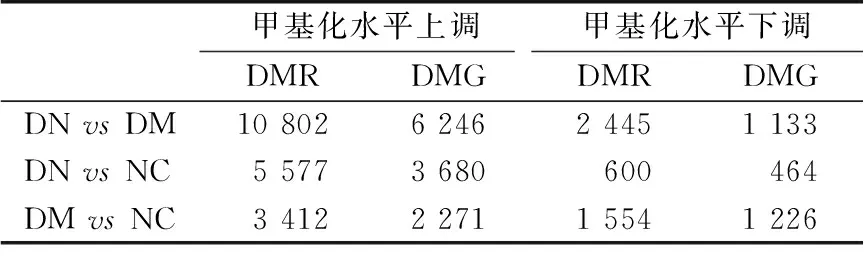
表1 三组患者肾组织DMR甲基化水平的变化
DMR:差异甲基化区域;DN:糖尿病肾病组;DM:糖尿病非肾
病组;NC:正常对照组;DMG:差异甲基化基因
DMR关联基因分析对DN组与DM组比较得到的13 247个DMR关联的7 379个DMG进行差异基因功能分类注释(GO)分析,富集到14 119条GO条目,其中2 190条GO条目有显著差异(P<0.05)(图2)。差异基因生物学路径(KEGG)分析得到267条通路,其中有87条通路显著富集(P<0.05),包括局灶黏连,黏合连接,MAPK 信号通路等。
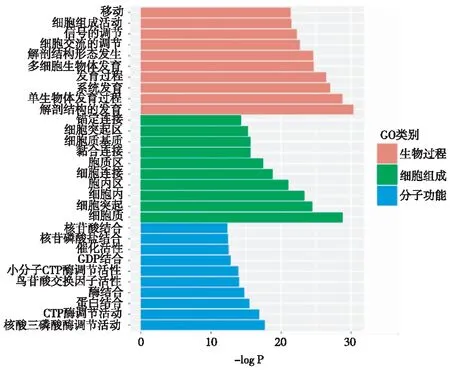
图2 糖尿病肾病组与糖尿病非肾病组差异基因的GO分析结果
对富集于局灶黏连通路的DMG进行分析,发现一些整合素基因的甲基化水平在DN患者和糖尿病非肾病患者中存在显著差异(表2)。
DMR和DMG的关联分析结果根据GEO数据库DN肾组织的转录组学数据(GSE30122)[12], 分析得到DN患者的差异表达基因。将差异表达基因和由DNA甲基化表达谱得到的DMG进行联合分析,探索DNA的甲基化水平和基因的表达水平之间的相关性。结果发现共有90个DMR关联到的62个差异基因的甲基化水平和表达水平之间存在负相关,这些基因包括ARHGAP28、FOXC1、ITGA8、NEU1、COL4A3、FLT1、LAMA1、MYLK3、FGF9、MYC等。
在这些负相关的基因里,62个基因富集到49条GO条目,其中24条GO条目差异显著(P<0.05)。包括肌动蛋白细胞骨架,细胞外基质结构组成等。另外,有28个基因富集到9条KEGG通路,其中7条通路有显著差异(P<0.05),包括局灶黏连,PI3K-Akt信号通路,细胞外基质-受体相互作用等。Fms样酪氨酸激酶1(fms related tyrosine kinase 1,FLT1)显著富集于局灶黏连(P=0.001)和PI3K-Akt信号通路(P=0.002)。
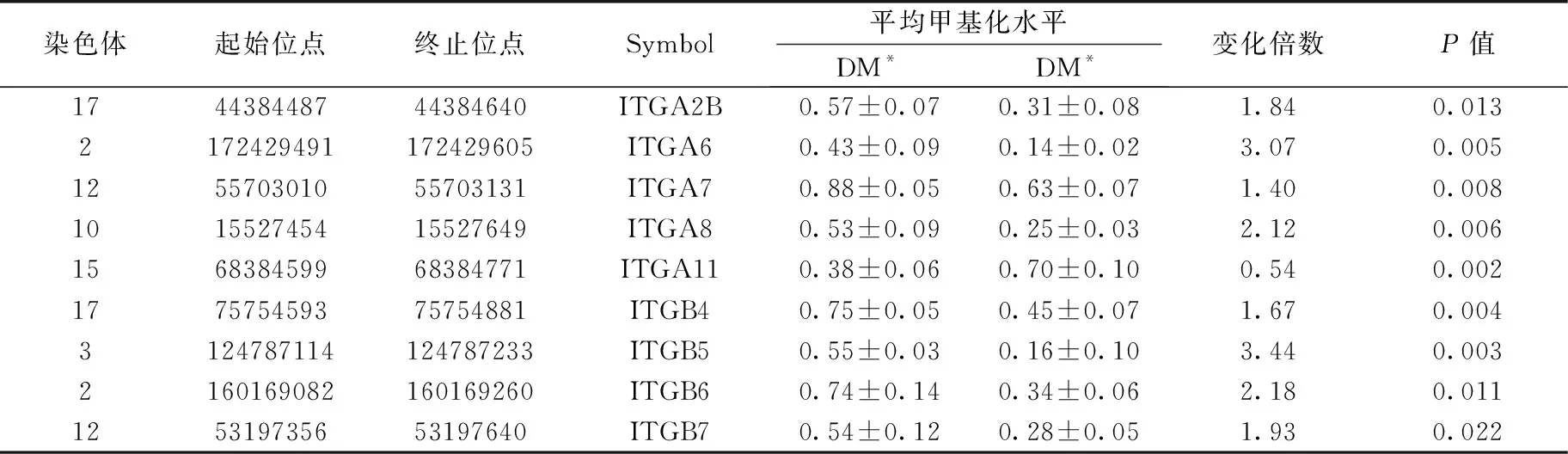
表2 DN患者与DM患者整合素基因甲基化水平的比较
DN:糖尿病肾病组;DM:糖尿病非肾病组;ITGA2B/6/7/8/11:整合素α 2B/6/7/8/11;ITGB4/5/6/7:整合素β 4/5/6/7;*:数据用
均数±标准差表示
讨 论
近年来表观遗传修饰参与DN的发病机制逐渐受到关注和重视,已有一些研究利用DN患者外周血白细胞或唾液细胞DNA,通过甲基化芯片检测的方法,发现高糖微环境导致的DNA甲基化改变在DN的发生发展中发挥重要作用[10-11,13-14],但有关DN患者肾组织DNA甲基化谱的研究,目前尚未见报道。
CpG岛是基因的一种重要标志,参与基因表达的调控,影响染色质的结构,正常细胞CpG岛由于被保护而处于非甲基化状态[15]。本研究通过对DN患者肾组织进行全基因组测序分析发现,DN患者的整体CpG甲基化水平高于DM组和正常对照组,这和Maghbooli等[9]的研究结果相似。
DMR是指基因组里甲基化状态不相同的区域,可通过改变染色体的结构或者转录效率来调节基因表达。利用全基因组甲基化数据,我们进一步对DMR进行分析,发现DN患者与糖尿病非肾病患者之间的DMR数目明显高于这两组患者与正常对照之间的DMR数目,表明众多DMR参与了DN发生发展的过程。与DM组相比,DN患者大部分DMR呈现高甲基化水平;与正常对照组相比,糖尿病非肾病患者的大部分DMR也呈现高甲基化,进一步表明DNA的甲基化改变参与糖尿病和DN的发生发展过程。
本研究发现,DN组与DM组比较得到的DMG主要和局灶黏连、黏合连接、MAPK 信号通路有关。局灶黏连作为细胞和外界环境的机械连接和生物信号中心,在整合蛋白绑定和聚集处集中并引导许多信号蛋白质。有文章报道ITGA11在CCL4诱导的肝纤维化小鼠模型以及人类肝纤维化患者中表达上调[16],同时敲除ITGA11 可以显著减少TGFβ诱导的α-SMA的分化和collagen-I等细胞外基质的积聚。另有研究表明整合素拮抗剂可减少糖尿病肾病大鼠模型中蛋白尿和肾纤维化的情况[17]。本研究中,整合素家族基因ITGA11、ITGA2B、ITGA6、ITGA7、ITGA8、ITGB4、ITGB5、ITGB6和ITGB7的甲基化水平在DN组和DM组之间均有显著差异。而在先前的相关研究中,Sapienza等[11]曾利用基因芯片对糖尿病肾病患者唾液DNA中27578个CpG位点进行甲基化分析,其结果发现ITGB4、ITGB6、ITGB8等基因的多个CpG位点存在显著差异。本结果与之类似。进一步表明整合素基因甲基化水平的改变在DN的发生发展中可能起重要作用。
DNA的甲基化对基因表达水平发挥重要的调控作用,启动子区域的甲基化对于基因的表达有重要影响。通常情况下,启动子区DNA甲基化水平增加,可通过阻断转录起始因子与启动子的结合直接抑制基因转录;可招募甲基化CpG结合蛋白,与5-甲基胞嘧啶结合后,进一步阻碍转录起始因子与启动子结合;还可招募组蛋白去乙酰化酶形成复合物,使组蛋白去乙酰化,导致染色质聚缩成非活性致密结构,间接抑制基因转录;而启动子区DNA甲基化水平降低则会导致基因表达的增加[18-19]。
GSE30122采用的是人肾组织。其中DN患者取其肾活检组织。入选标准:符合糖尿病的诊断,有蛋白尿,GFR<60 ml/min,组织学改变符合DN的特点;排除肝炎、狼疮及其他可引起肾炎的因素。正常对照选取移植肾或肾肿瘤切除术后的癌旁肾组织。入选标准:GFR>60 ml/min,无蛋白尿,血清肌酐、尿素氮正常,肾小球及肾小管间质纤维硬化<10%。利用激光微分离技术将肾组织切割为肾小管或肾小球。GSE30122包括三个系列,分别为:GSE30528为肾小球的测序结果,包括9例DN和13例正常对照;GSE30529 为肾小管的测序结果,包括10例DN和12例正常对照;GSE30566 为正常对照的13例肾小球和12例肾小管之间的测序结果。而我们的数据分析,是按疾病状态进行分组,即仅进行DN患者(包括肾小管和肾小球两部分的数据)和正常对照之间的比较,对于肾小球和肾小管之间不做差异表达基因的相关分析。
DMG和差异表达基因联合分析发现,62个差异基因的甲基化水平和表达水平之间呈现负相关,且这些差异基因显著富集到局灶黏连和PI3K-Akt信号通路。其中包括FLt-1,其为血管内皮生长因子的特异性受体,二者结合可以引起血管内皮细胞通透性增加,造成血浆蛋白外渗。足细胞Flt-1表达减少,会导致足细胞的细胞骨架重构,引起足突融合,使肾小球的滤过屏障受损,进一步导致大量蛋白尿和肾功能衰竭。在我们的研究结果中,Flt-1的甲基化水平在DN患者和正常对照中存在显著差异,进一步表明DNA甲基化在DN的发生发展中可能起重要作用。
小结:本研究利用全基因组甲基化测序技术,首次建立了DN患者肾组织的DNA甲基化表达谱,发现DN患者的整体CpG甲基化水平明显高于糖尿病非肾病患者,DN患者肾组织的DMR数目增加,关联的差异基因的甲基化水平和mRNA表达水平之间存在负相关,表明DNA甲基化在DN的发生发展中可能起重要作用,为DN发病机制的研究提供了新思路。
1 Liu ZH.Nephrology in china.Nat Rev Nephrol,2013,9(9):523-528.
2 Badal SS,Danesh FR.New insights into molecular mechanisms of diabetic kidney disease.Am J Kidney Dis,2014,63(2 Suppl 2):S63-83.
3 Reidy K,Kang HM,Hostetter T,et al.Molecular mechanisms of diabetic kidney disease.J Clin Invest,2014,124(6):2333-2340.
4 Kato M,Natarajan R.Diabetic nephropathy--emerging epigenetic mechanisms.Nat Rev Nephrol,2014,10(9):517-530.
5 Lu Z,Liu N,Wang F.Epigenetic Regulations in Diabetic Nephropathy.J Diabetes Res,2017,2017:7805058.
6 Wanner N,Bechtel-Walz W.Epigenetics of kidney disease.Cell Tissue Res, 2017,369(1):75-92.
7 Reddy MA,Tak Park J,Natarajan R.Epigenetic modifications in the pathogenesis of diabetic nephropathy.Semin Nephrol,2013,33(4):341-353.
8 Reddy MA,Natarajan R.Epigenetics in diabetic kidney disease.J Am Nephrol,2011,22(12):2182-2185.
9 Maghbooli Z,Larijani B,Emamgholipour S,et al.Aberrant DNA methylation patterns in diabetic nephropathy.J Diabetes Metab Disord,2014,13:69.
10 Bell CG,Teschendorff AE,Rakyan VK,et al.Genome-wide DNA methylation analysis for diabetic nephropathy in type 1 diabetes mellitus.BMC Med Genomics,2010,3:33.
11 Sapienza C,Lee J,Powell J,et al.DNA methylation profiling identifies epigenetic differences between diabetes patients with ESRD and diabetes patients without nephropathy.Epigenetics,2014,6(1):20-28.
12 Woroniecka KI,Park AS,Mohtat D,et al.Transcriptome analysis of human diabetic kidney disease.Diabetes,2011,60(9):2354-2369.
13 Zhang H,Cai X,Yi B,et al.Correlation of CTGF gene promoter methylation with CTGF expression in type 2 diabetes mellitus with or without nephropathy.Mol Med Rep,2014,9(6):2138-2144.
14 Yang XH,Cao RF,Yu Y,et al.A study on the correlation between MTHFR promoter methylation and diabetic nephropathy.Am J Transl Res,2016,8(11):4960-4967.
15 Ehrlich M,Gama-Sosa MA,Huang LH,et al.Amount and distribution of 5-methylcytosine in human DNA from different types of tissues of cells.Nucleic Acids Res,1982,10(8):2709-2721.
16 Bansal R,Nakagawa S,Yazdani S,et al.Integrin alpha 11 in the regulation of the myofibroblast phenotype:implications for fibrotic diseases.Exp Mol Med,2017,49(11):e396.
17 Zhou X,Zhang J,Haimbach R,et al.An integrin antagonist (MK-0429) decreases proteinuria and renal fibrosis in the ZSF1 rat diabetic nephropathy model.Pharmacol Res Perspect,2017,5(5).
18 Burgess DJ.Epigenetics:Bacterial DNA methylation gets SMRT.Nat Rev Genet,2013,14(1):4.
19 Jones PA.Functions of DNA methylation:islands,start sites,gene bodies and beyond.Nat Rev Genet,2012,13(7):484-492.

