大豆抗大豆花叶病毒病基因研究进展
王大刚,李凯,智海剑
大豆抗大豆花叶病毒病基因研究进展
王大刚1,李凯2,智海剑2
(1安徽省农业科学院作物研究所/安徽省农作物品质改良重点实验室,合肥 230031;2南京农业大学大豆研究所/国家大豆改良中心/ 作物遗传与种质创新国家重点实验室,南京 210095)
大豆花叶病毒(soybean mosaic virus,SMV)病是严重危害世界大豆((L.) Merr.)生产的主要病害之一。近十年来,国内外关于大豆对SMV抗病基因的遗传标记定位、候选抗病基因的分析及大豆抗SMV的调控网络等研究取得许多新进展。大豆对SMV的抗性遗传主要分为数量抗性和质量抗性,其中数量抗性的遗传主要由1对加性主基因+加性-显性多基因共同控制;对不同SMV株系的质量抗性遗传分别由1对不同的显性基因控制。标记定位研究发现,大豆对SMV数量抗性位点主要分布在大豆的第6、10和13等染色体上。22个对SMV具有单显性质量抗性的基因位点已被标记定位在大豆的第2、6、13和14染色体上,且定位的多数抗病基因位点两侧标记间的物理距离都在1 Mb以内。其中第13染色体上的基因位点数最多,有、、R、R和R等10个,定位在第2染色体上的基因位点有8个,如、R、R、R和R等,第6和14染色体上各有2个基因位点,分别为R、R和、R。参考大豆全基因组序列(http://www.phytozome.net/soybean),利用生物信息学方法、表达谱分析及克隆测序技术等进一步缩小了大豆抗SMV候选基因的筛选范围。目前,在大豆第2染色体上确定的抗SMV候选基因主要有8个:、、、、、、和,在第6染色体上的是,在第13和14染色体上的抗SMV候选基因分别有9个和6个:、、、、、、、和、、、、、。基于病毒诱导的基因沉默VIGS(virus induced gene silencing,VIGS)和转基因操作等技术,研究发现抗SMV相关基因、、等参与大豆对SMV的抗性,属于正调控因子;而和等则增加大豆对SMV的易感性,为负调控因子。在综合SMV抗病基因的相关研究基础上,构建了基于和介导对SMV极端抗性的调控网络模型。介导的大豆对SMV极端抗性调控模型的建立为大豆抗SMV信号网络的研究提供了新的方向。介导的大豆对SMV极端抗性的主要机制是通过ABA信号的传导,从而使胞间连丝处的胼胝质沉积以抑制病毒从最初侵染的细胞向健康细胞的转移。本文系统综述了SMV抗病基因方面的最新研究成果并对该领域未来的研究方向进行了展望,以期为大豆抗SMV分子设计育种和抗病基因的机理研究提供参考。
大豆;大豆花叶病毒;抗病基因;标记定位;功能研究
大豆((L.) Merr.)是世界上食用植物油和植物蛋白的主要来源,生长过程中受多种病害侵染。其中,大豆花叶病毒(soybean mosaic virus,SMV)病对大豆的产量和品质均可造成严重危害。由于SMV分布广、危害重、化学药剂难以防治,一直是大豆种植面临的主要问题。选育和种植抗病品种是控制该病最经济、有效且对环境友好的防治方法。
筛选新的抗病种质并发掘新的抗病基因是拓宽大豆抗病遗传基础、成功培育抗SMV品种的关键。国家和各省大豆新品种审定实行SMV抗性“一票否决”制[1],对抗SMV育种起到了极大的推进作用。随着大豆抗SMV研究的深入,王大刚等[2]、李凯等[3]曾对国内外学者在SMV抗源筛选、抗性遗传方式和抗病基因分子标记定位等方面的研究进行了阐述。
本文主要聚焦SMV抗病基因的标记定位、候选抗病基因的分析及抗SMV相关基因的功能鉴定和参与的调控网络等研究,通过系统的梳理和总结,以期为大豆分子设计育种、抗SMV功能基因的利用和新基因的发掘提供参考。
1 大豆对SMV抗病基因的标记定位
大豆对SMV的抗性主要存在数量抗性和质量抗性两类[4]。数量抗性遗传主要由1对加性主基因+加性-显性多基因共同控制[5],利用中豆29×中豆32的重组自交系RIL(recombinant inbred lines,RIL)群体,郭丹丹等[6]研究还发现大豆对SMV株系SC3的数量抗性遗传由2对等加性主基因控制。大豆对SMV质量抗性遗传,国内外普遍认为是由1对显性抗病基因控制[7-11]。通过抗病品种与感病品种的正反交组合研究发现,大白麻对SMV株系SC4及齐黄22对SC8均是由单显性核基因控制,且抗性不受细胞质影响[12]。
迄今为止,已经命名和定位的大豆抗SMV单显性基因位点主要有“”(resistance of soybean mosaic virus,)系列和“R”(resistance of soybean mosaic virus in China,R)系列两类。其中“”系列抗病基因位点主要有、、和等,由美国、加拿大和韩国等学者鉴定、命名和定位[13-28](表1);“R”系列抗病基因位点主要有R、R—R、R—R、R、R、R—R、R等,由中国和印度学者鉴定、命名和定位[27-47](表1)。
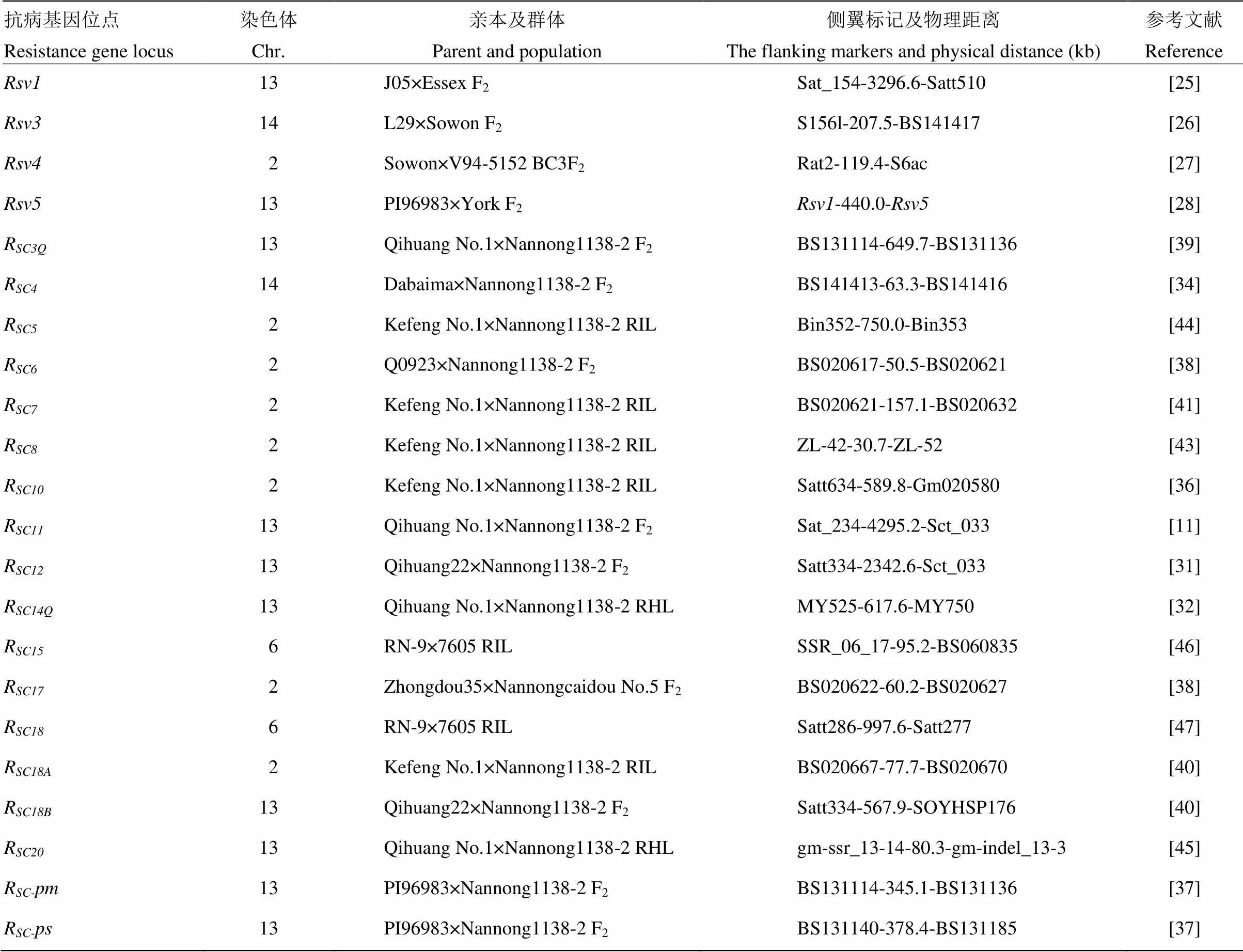
表1 大豆花叶病毒(SMV)抗病基因位点的标记定位
BARCSOYSSR_02_、BARCSOYSSR_06_、BARCSOYSSR_13_和BARCSOYSSR_14_简写为BS02、BS06、BS13和BS14。下同
BARCSOYSSR_02_, BARCSOYSSR_06_, BARCSOYSSR_13_ and BARCSOYSSR_14_ abbreviated as BS02, BS06, BS13 and BS14 respectively. the same as below
由表1可知,目前国内外已标记定位的SMV抗病基因位点有22个,主要分布在大豆第2、6、13和14染色体上,其中第13染色体上基因位点最多,有10个,定位在第2染色体上的基因位点有8个,而在第6和14染色体上均有2个SMV抗病基因位点。除少数几个基因位点两侧分子标记的物理距离较远外,多数抗病基因位点两侧分子标记的物理距离均在1 Mb以内(表1)。
SMV抗病基因位点最早由Yu等[15]利用SSR(simple sequence repeats,SSR)标记SM176定位在大豆F连锁群(第13染色体)上,遗传距离为0.5 cM。利用RFLP(restriction fragment length polymorphism,RFLP)、RAPD(random amplified polymorphic DNA,RAPD)和SSR标记,Gore等[18]构建了包含和花生斑驳病毒抗病基因位点在内的6.8 cM精细遗传图谱。Shi等[25]构建J05与Essex杂交的F2群体,进一步将位点定位在SSR标记Sat_154和Satt510之间,遗传距离分别为0.2和2.3 cM(表1)。参考大豆Willimas82全基因组序列(http://www.phytozome.net/soybean,Wm82.a2.v1),通过分析发现SMV抗病基因位点所在的物理区间起止位置为28 506 083—31 802 676 bp,物理距离3 296.6 kb(表1)。尽管是位点的一个等位基因[20],但其在大豆第13染色体上的物理位置并不确定[48]。利用Suweon97和Willimas82的F2群体,Ma等[48]研究发现Suweon97携带的抗病基因定位在标记BARCSOYSSR_13_1114和BARCSOYSSR_13_1115之间,与PI96983携带的位点属于不同的区域。两标记间的物理距离为97.5 kb(http://www.phytozome.net/soybean,Wm82.a2.v1)。
位点被定位在大豆连锁群B2(第14染色体)上标记A519和M3Satt之间,遗传距离分别为0.9和0.8 cM[19],而基于标记A519开发的3个SNP(single nucleotide polymorphism,SNP)标记A519-460、A519-956和A519-1165与紧密连锁[24]。Shi等[25]发现SSR标记Sat_424与大豆J05携带的紧密连锁,遗传距离为1.5 cM。利用新开发的标记,Suh等[26]进一步将定位在分子标记S156l和BARCSOYSSR_14_1417之间,遗传距离分别为0.3和0.5 cM,候选区域在第14染色体上的起止位置为46 852 283—47 059 763 bp(http://www.phytozome.net/ soybean,Wm82.a2.v1),物理距离207.5 kb(表1)。
Hayes等[49]利用BSA(bulked segregant analysis,BSA)法和F2群体将抗病基因定位在大豆D1b连锁群(第2染色体)上,SSR标记Satt558和Satt542与之间的遗传距离分别为7.8和4.7 cM。利用比较作图策略,Hwang等[50]进一步将定位在EST(expressed sequence tag,EST)标记AI856415-g和AW471852A之间,遗传距离分别为2.8和2.4 cM。基于大豆基因组鸟枪测序法,Saghai Maroof等[51]将锁定在1.3 cM的精细作图区域内。2016年,Ilut等[27]对区域进行了深度重测序,发现与两侧的标记Rat2和S6ac的遗传距离均为0.2 cM(表1),起止位置为12 044 285—12 163 994 bp(http://www. phytozome.net/soybean,Wm82.a2.v1),物理区间119.7 kb(表1)。
美国大豆品种York携带的抗病基因早先被命名为[52],被推测是PI96983携带抗病基因位点的等位基因,2个品种对SMV株系G1–G7的抗性反应存在差异[52]。Klepadlo等[28]利用PI96983()×York()构建包含3 000个F2:3家系的群体检测了2个抗病基因之间的等位性关系,结果发现和是2个紧密连锁的基因位点,遗传距离为2.2 cM,物理距离约为440 kb,最后作者将York携带的抗病基因重新命名为[28]。
科丰1号是中国科学院遗传与发育生物学研究所从河北地方大豆品种系统选择育成,对中国的18个SMV株系(SC1–SC13、SC16和SC18—SC21)和美国的7个株系(G1—G7)均具有抗性[53-55]。利用科丰1号×南农1138-2组配的F2、RIL和剩余杂合系RHL(residual heterozygous lines,RHL)群体,Adhimoolam等[44]、Fu等[29]、Yan等[41]、Wang等[33]、Zhao等[43]、李春燕等[36]、郭东全等[56]和Li等[40]先后将科丰1号对SMV株系SC5、SC7、SC8、SC10、SC13、SC18携带的抗病基因R、R、R、R、R、R定位在大豆的第2染色体上(表1和图1)。此外,阳小凤等[38]将大豆品系Q0923对SC6株系具有的抗病基因R和中豆35携带对SC17的抗病基因R也均定位在第2染色体上(表1和图1)。
根据大豆Willimas82的参考基因组序列(http:// www.phytozome.net/soybean,Wm82.a2.v1),结合上述标记定位的结果分析可以发现,大豆第2染色体上的SMV抗病基因位点主要分布在11 188 722—13 104 514 bp(表1,图1)。其中有6个SMV抗病基因位点的候选区段交叉重叠,集中分布在12 044 285—12 313 444 bp约269.2 kb的区间内,推测这6个基因位点可能紧密连锁或具有等位关系。进一步对这一区间分析发现,该区间内有20个候选基因(—)(图1,表2),但均不具有NBS-LRR(nucleotide binding site-leucine rich repeat,NBS-LRR)抗病基因保守结构域。
齐黄1号是山东省农业科学院从山东寿张地方品种系统选择育成的另一个对SMV具有广谱抗性的品种,对中国的18个SMV株系(SC1—SC8、SC11—SC14、SC16—SC18和SC20—SC22)和美国的7个株系(G1—G7)也均具有抗性[53-55]。利用F2、RIL和RHL群体,Zheng等[39]、白丽等[11]、Li等[30]、Ma等[31-32]、Li等[40]和Adhimoolam等[45]将齐黄1号及其衍生系齐黄22携带的抗SMV基因R、R、R、R、R和R均定位在大豆的第13染色体上(表1和图2)。Yang等[37]将美国抗源PI96983携带对SMV株系SC3、SC6、SC17的抗病基因pm以及对SC7的抗病基因Rscps也均定位在第13染色体上相邻的区域内(表1和图2)。

表2 抗病基因位点Rsv4、RSC5、RSC6、RSC7、RSC8和RSC17在大豆第2染色体上可能的候选基因a
a数据来源于大豆的全基因组数据库(http://www.phytozome.net/soybean,Wm82.a2.v1,2018年3月12日),加粗字体表示第2染色体上可能性较大的SMV抗病候选基因。下同
aThe data from the soybean reference genome sequence (http://www.phytozome.net/soybean, Wm82.a2.v1, March 12, 2018),the bold font indicates the putative candidate genes to SMV in soybean on chromosome 2. the same as below
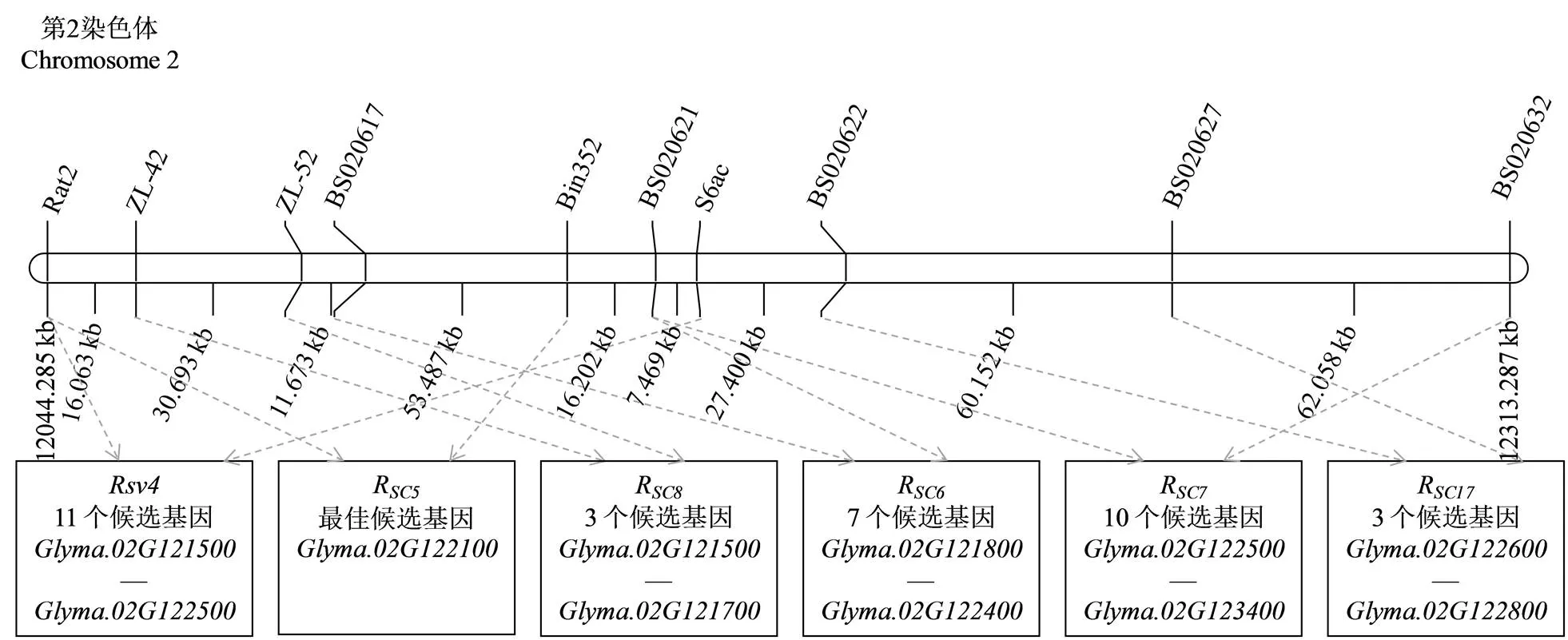
虚线箭头部分表示单个SMV抗病基因的物理区间;最下面的框内是SMV抗病候选基因的个数及范围。下同

图2 大豆抗SMV基因在第13染色体上的遗传、物理图谱及抗病候选基因
基于大豆Willimas82的参考基因组序列(http:// www.phytozome.net/soybean,Wm82.a2.v1),结合标记定位结果分析发现,国内外定位于第13染色体上的SMV抗病基因位点的候选区段主要在27 656 895—31 951 960 bp成簇存在(图2),物理距离为4.295 Mb,且这一区段内富集着许多具NBS-LRR结构域的候选基因(表3)。从图2可以发现,在4.295 Mb的物理区段内,SMV抗病基因位点的候选区段均存在相互重叠的现象,相较于第2染色体上候选区段内的基因数,第13染色体上候选区段内的基因数量更多,推测该候选区段内的多数基因连锁较为紧密,难以发生交换。
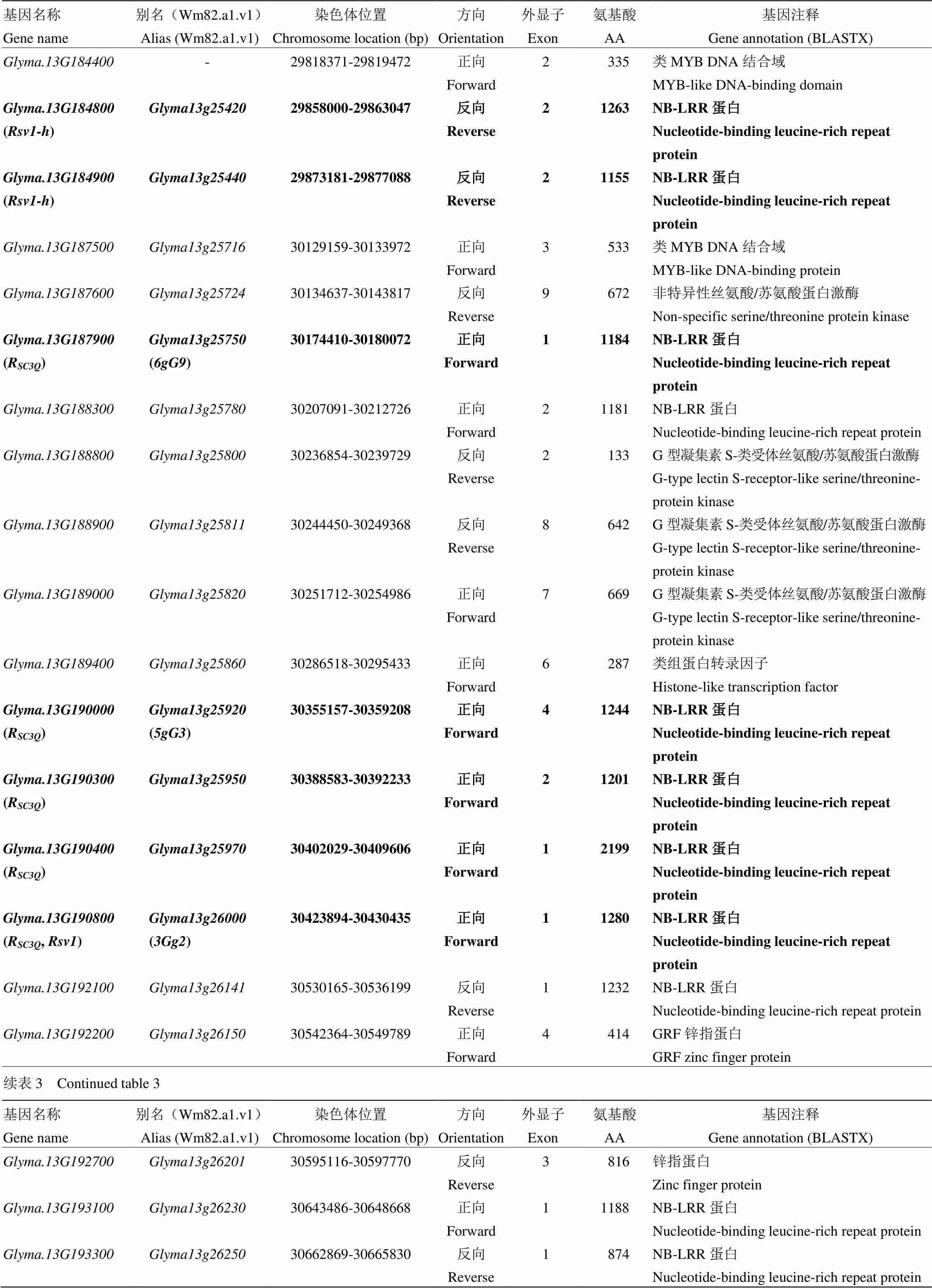
表3 SMV抗病基因位点Rsv1、Rsv5、RSC3Q、RSC11、RSC12、RSC14Q、RSC18B、RSC20、Rsc-pm和Rsc-ps在大豆第13染色体上部分可能的候选基因
RN-9是继科丰1号和齐黄1号后发现的又一个广谱抗源,对15个SMV株系均表现极端抗性,特别是对能感染所有鉴别寄主的SC15株系表现抗病[35,47]。利用RN-9×7605的RIL群体,Yang等[35]将RN-9携带的对SMV株系SC15具有的抗病基因R定位在大豆第6染色体上,该基因与SSR标记Sat_213和Satt286之间的遗传距离分别为8.0和6.6 cM。2017年,Ren等[46]进一步将R锁定在标记SSR_06_17和BS060835之间,遗传距离分别为1.44和0.63 cM(表1),起止位置为15 635 537—15 730 967 bp(http://www. phytozome.net/soybean,Wm82.a2.v1),物理区间为95.2 kb(表1,图3-a)。
此外,李凯等[47]将RN-9携带对SC18的抗性基因R定位在标记Satt286和Satt277之间,遗传距离分别为6.12和4.69 cM,物理区间为997.6 kb。参考大豆Willimas82的基因组序列(http://www.phytozome. net/soybean,Wm82.a2.v1),分析发现抗病基因R和R在第6染色体上的候选区段没有重叠的区域(图3-a)。其中,在95.4 kb的R候选基因区段内存在5个候选基因(—),且均不具有NBS-LRR抗病基因保守结构域(表4,图3-a),在R候选基因区段内,有73个候选基因存在(—)。
利用大白麻×南农1138-2的F2作图群体,Wang等[34]将大白麻携带的抗病基因R精细定位在大豆第14染色体上,紧密连锁的2个基因组SSR标记BARCSOYSSR_14_1413和BARCSOYSSR_14_1416与R的遗传距离分别为0.17和0.27 cM(表1),物理距离63.4 kb(表1,图3-b)。参考大豆Willimas82的基因组序列(http://www.phytozome.net/soybean,Wm82.a2.v1),结合R和定位结果分析可以发现,在大豆第14染色体46 852 283—47 135 871 bp共有17个可能的候选基因(表5),其中有5个候选基因具NBS-LRR保守结构域。从图3-b还可以发现,2个SMV抗病基因位点R和的候选基因位置具有重叠的物理区段,推测R与可能是等位或紧密连锁的。
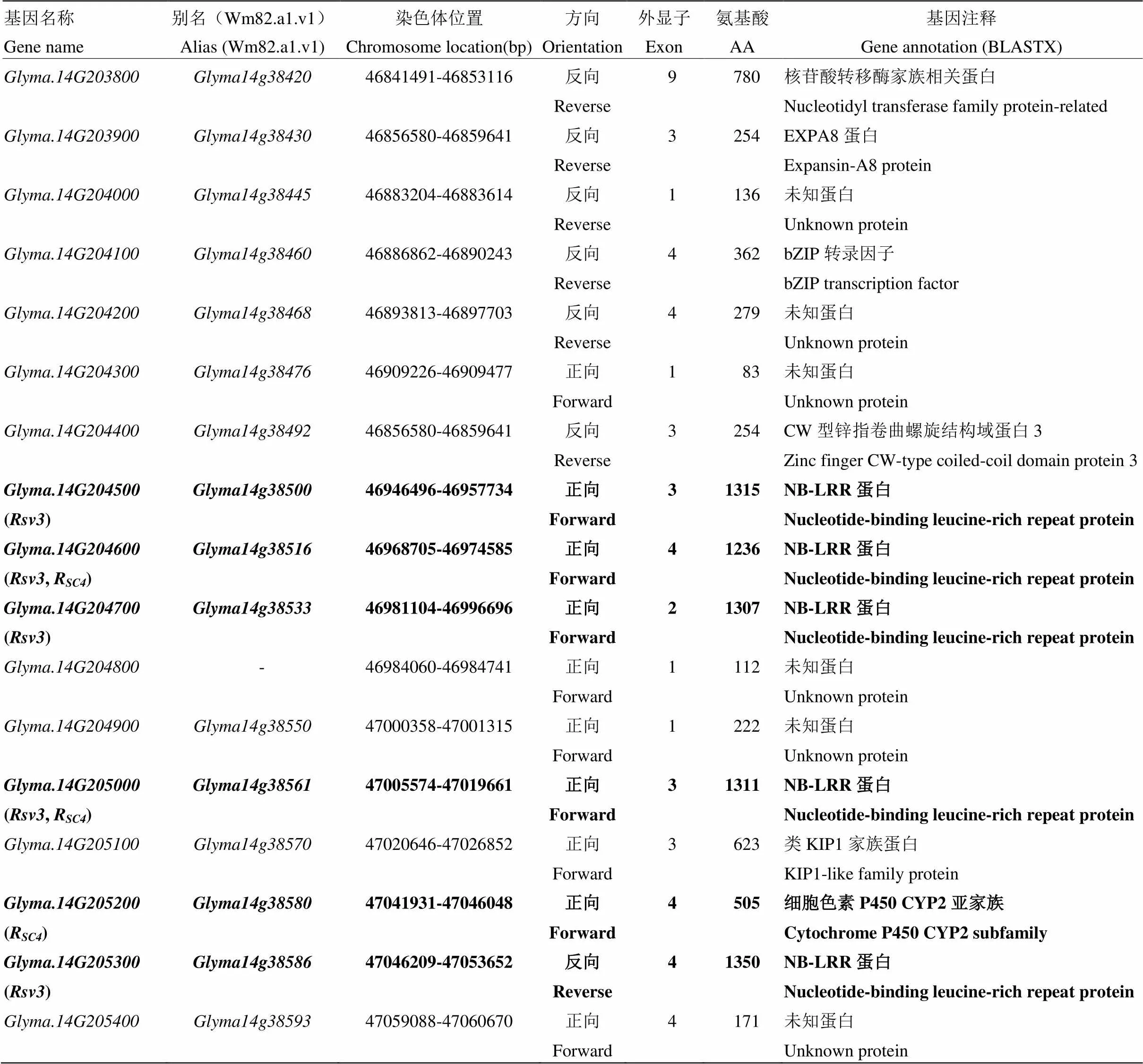
表5 抗病基因位点Rsv3和RSC4在大豆第14染色体上可能的候选基因
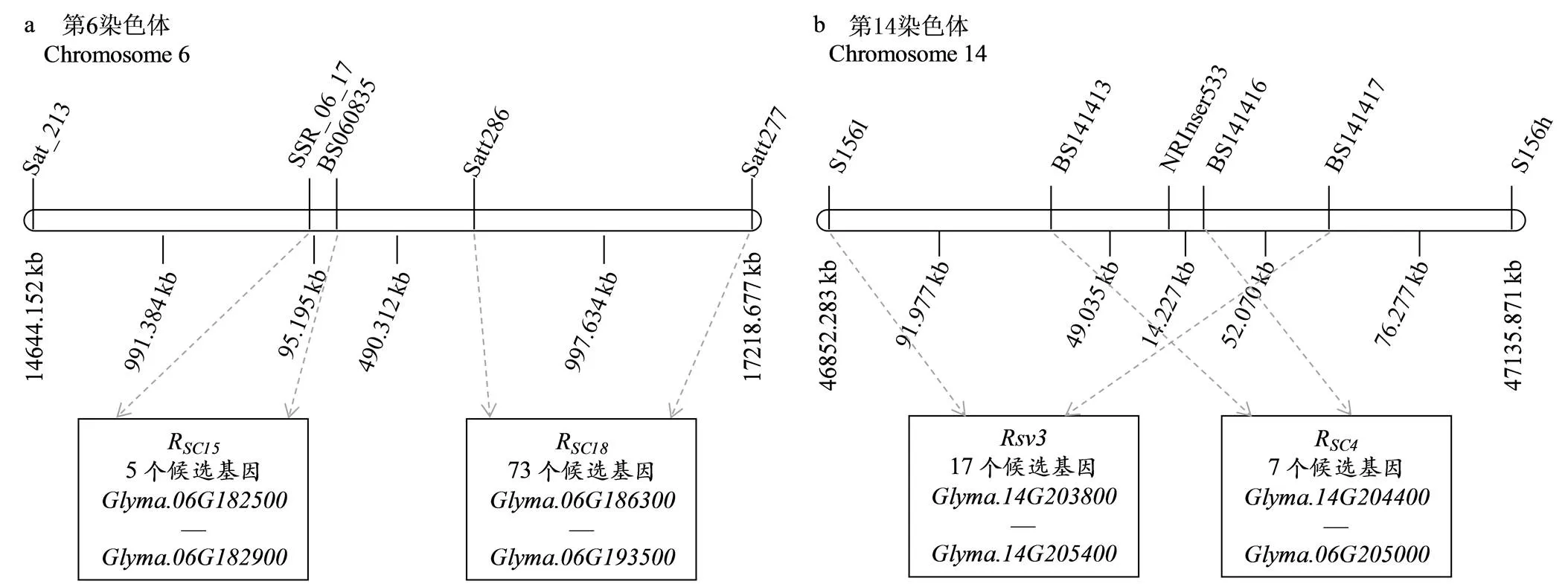
图3 大豆抗SMV基因在第6和14染色体上的遗传、物理图谱及抗病候选基因
2 SMV抗病候选基因预测
随着大豆对SMV抗病基因不断地发现、标记和精细定位,大豆对SMV候选抗病基因保守结构域的生物信息学分析、qRT-PCR(quantitative real time polymerase chain reaction,qRT-PCR)表达分析及克隆测序分析等也在不断地拓展和深入,极大地缩小了候选基因的筛选范围。
在第2染色体上,Saghai Maroof等[51]对的候选基因序列(http://www.phytozome.net/soybean,Wm82.a1.v1)进行分析发现,这些基因均不具有NBS-LRR结构域(图1,表2),推测属于一种新类型的抗病基因,其可能是通过干扰病毒感染和细胞间运动,并延迟病毒在维管束间移动,从而达到抗病的目的。Ilut等[27]通过对SMV株系G1和G7具有不同抗性反应的6个野生大豆和13个栽培大豆位点所在的120 kb的区域进行深度测序分析发现,这一区域至少存在2个单倍型,其中最可能的一个单倍型包含4个候选基因:1个胁迫响应基因、2个RD类受体蛋白酶基因(和)和1个铜转运蛋白基因()(表2)。从基因结构可以看出,这几个候选基因均不具有经典的NBS-LRR型抗病基因结构,验证了可能属于一种新类型抗病基因的推测[27,50]。作者对该区域的单倍型系统发育分析发现位点是最新才从野生大豆进化到栽培大豆中的[27]。不过,Klepadlo等[28]通过对10个大豆品种的测序却证实可能是的候选基因(表2)。
Adhimoolam等[44]将SNP标记及抗性鉴定表型数据进行关联分析和qRT-PCR表达分析发现,科丰1号携带对SC5最有可能的抗病候选基因是()(表2)。
根据候选基因的结构分析,Yan等[41]推测具NBS-LRR结构域的基因,具有热休克蛋白40(HSP40)结构的基因()和具有丝氨酸羧肽酶型(Serine carboxypeptidase-type)的基因可能是R的候选基因,不过基因和在最新的大豆参考基因组序列中已经被重新进行整合(http://www.phytozome.net/soybean,Wm82.a2.v1,2018年3月12日)。CHE等[57]通过GWAS(genome wide association study,GWAS)和qRT-PCR技术研究发现,与拟南芥转录调控因子、真核生物延伸因子和同源的3个大豆基因(、和)可能参与了大豆抗SC7株系的过程。
Wang等[33]运用qRT-PCR技术对精细定位的R抗病候选基因分析发现,其中5个候选基因的表达量在抗感品种间的响应模式或时间上有很大的差异,推测这些基因可能参与了大豆对SMV株系的抗性反应。Zhao等[43]进一步将R的候选基因确定为具MADS-box保守结构域的基因和(表2)。
通过对SC15的抗病品种RN-9和感病品种7605的5个候选基因(—)DNA和cDNA进行测序和qRT-PCR分析,Ren等[46]推测过氧化物膜蛋白基因()是R最有可能的候选基因[46]。根据测序和表达分析,Li等[40]研究发现、和可能是科丰1号对SMV株系SC18的抗病候选基因(表4)。
通过分析大豆抗病基因位点定位区域的NBS-LRR基因簇,Hayes等[58]从抗SMV品种PI96983中克隆到一个在结构上具有non-TIR(toll interleukin1 receptor,TIR)-NBS分子特征的抗病候选基因(),该基因全长3 390 bp,包含一个完整的编码框,SHAO等[59]从抗病基因进化的角度证实了()是的一个候选基因。Ma等[48]对抗病基因的8个候选基因序列进行分析发现,和具有CC(coiled-coil,CC)-NBS-LRR抗病基因的保守结构,推测这两个基因可能是的最佳候选基因(表3)。
在精细定位的基础上,Zheng等[39]通过qRT-PCR分析发现,、()、()、()和()等5个基因可能是R的候选基因(表3)。Li等[42]进一步通过转录组和qRT-PCR研究表明齐黄1号携带对SC3抗病基因R可能的候选基因为()、()、()、()等4个具NBS-LRR抗病基因保守结构域的基因(表3),其中有3个候选基因与Zheng等[39]的研究结果相一致。
Adhimoolam等[45]综合候选基因的测序结果和qRT-PCR表达分析发现,齐黄1号携带对SC20最有可能的抗病候选基因是和(表2)。
在大豆的第14染色体上,基于抗病基因R精细定位的结果,Wang等[34]参考Willimas82的参考基因组序列和qRT-PCR技术发现3个抗病候选基因()、()和()在抗感品种间不同时间点的表达量具有差异,初步推测这些基因可能参与了大豆对SC4株系的抗性反应(表5)。
对抗病基因所在的154 kb区段进行分析,Suh等[26]发现这个区段是一个富集多个抗病基因的基因簇,其中有5个抗病候选基因()、()、()、()和()编码NB-LRR结构的抗病蛋白(表5),作者认为可能是NB-LRR家族中的一员[26]。值得注意的是,SMV抗病基因R和均在这个区段内,说明R和属于同一个抗病基因簇。Suh等[26]推测的基因和与Wang等[34]通过qRT-PCR获得的抗病候选基因是一样的,不过2个基因位点是不是同一个基因仍需进一步的研究分析。
通过分析抗病品种L29接种SMV株系G7后1—3 h的表达量,Redekar等[60]发现,基因()是5个候选基因中表达量最大的一个基因。比较测序研究表明,在抗病基因型(型)和感病基因型(3型)间有150个SNP和6个Indel差异,而且这种差异主要集中在LRR编码区。因此,作者推测最有可能是的候选基因[60](表5)。
3 SMV抗病相关基因筛选
大豆对SMV抗病基因的发掘除了利用精细定位图位克隆外,还有通过同源克隆等方法获得了一系列抗SMV相关基因的报道。
贺超英等[61]和Wang等[62]利用同源克隆的方法从科丰1号中获得2个SMV抗性相关基因和,它们都具有NBS和LRR等抗病基因的结构特征,RT-PCR分析表明二者均受SMV株系Sa接种和水杨酸处理的影响,推测和可能参与了大豆抗SMV的过程[62-63]。此外,Wang等[64]研究发现大豆同源异型-亮氨酸拉链蛋白基因具有HD-Zip结构域,是植物特有的转录因子。接种SMV株系N3后,该基因在感病品种中的表达量上调,而在抗病品种中则下调,初步证实可能作为转录激活因子参与大豆对SMV的抗性(表6)。
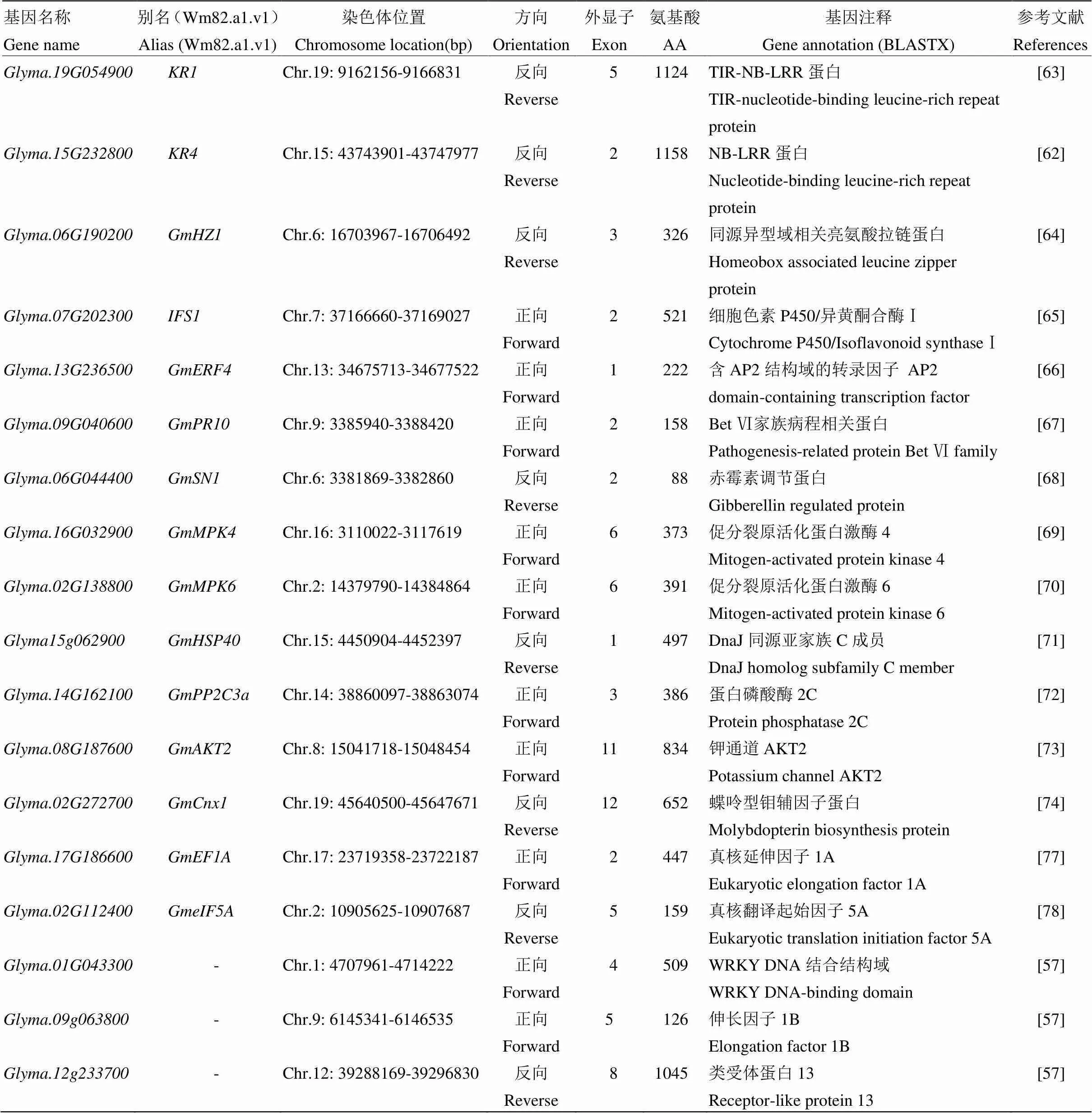
表6 参与大豆抗大豆花叶病毒可能的相关基因
关联分析研究发现,大豆异黄酮合成酶基因与大豆对SC3的抗性显著相关[65]。表达分析发现接种SC3后7 d,基因在抗病品种中的表达量显著上调,而在感病品种中没有变化,推测该基因可能参与了大豆抗SMV侵染的过程[65]。Zhang等[66]对大豆中一个耐胁迫基因研究发现,其能够被SMV诱导表达,推测该基因可能是大豆对SMV抗性反应的一个相关因子。是大豆中的一个PR10蛋白基因,RT-PCR分析表明,大豆接种SMV后该基因在叶片中被强烈诱导并高效表达,推测其可能在病毒诱导下使大豆获得系统抗性以抵抗外来病原菌的侵袭[67]。
通过转录组分析,He等[68]发现GAST(GA- stimulated transcript,GAST)基因亚家族成员在接种SMV后,在感病品种(感病型:)中的表达量下调,而在抗病品种(抗病型:)中没变化。进一步测序和聚类比较分析表明,GAST家族成员之一基因与土豆中的抗病基因同源,可能参与了大豆抗SMV的过程[68]。
4 大豆抗SMV相关基因的功能鉴定
在利用生物信息学、qRT-PCR及克隆测序等一系列技术对SMV抗病候选基因进行预测和抗SMV相关基因进行筛选的基础上,相关学者进一步通过病毒诱导的基因沉默VIGS(virus induced gene silencing,VIGS)、转基因操作等技术对抗SMV相关基因的功能进行了鉴定和研究。
与空载对照相比,Liu等[69-70]研究发现沉默大豆蛋白激酶()和()的植株均对SMV表现更好的抗性;双分子荧光互补(bimolecular fluorescence complementation,BiFC)试验表明,和与相互作用,同时和在的上游起作用;与相互作用,在的上游起作用[70](表6)。
热激蛋白在植物防御生物及非生物胁迫中具有至关重要的作用,Liu等[71]研究发现沉默具有DnaJ结构域的大豆热激蛋白基因Seo等[72]通过大豆与SMV互作研究表明大豆2C型蛋白磷酸酶基因()是介导的对SMV抗性中的一个调控因子。
Zhou等[73]研究表明在多施钾肥的情况下能够提高大豆对SMV的抗性,过表达大豆钾离子通道基因发现,转基因阳性植株对SMV的抗性明显增强。其可能的原因是大豆的过量表达导致其韧皮部发生改变,进而不利于外部病毒的侵染、移动和复制[73]。转基因研究发现SMV对过表达大豆硝酸还原酶及黄嘌呤脱氢酶辅助因子转基因植株的侵染率显著低于对照品种,转基因植株显著增强大豆对SMV株系的抗性[74]。
、和在抗感品种间表达量有显著差异且具有NB-LRR抗病基因保守结构域,它们被作为抗病基因R的3个候选基因。Li等[75]研究发现,沉默3个基因后的抗病品种显著感病,3个基因在抗感品种间的序列比对发现它们在抗感品种间均有氨基酸上的差异,推测这3个基因与大豆对SMV的抗病均有关系[75]。
Tran等[76]对的候选基因进行研究发现,在缺失基因型的大豆品种中过量表达基因后SMV的侵染被明显抑制;而对携带基因型的大豆品种L29进行瞬时沉默后发现,原来对L29不致病的病毒株系表现较多的SMV积累[76]。将SMV抗病候选基因过表达转化到拟南芥和大豆中,He等[68]发现与对照相比,过表达转基因拟南芥植株能够增强对萝卜花叶病毒的抗性,转基因大豆植株能够增强对SMV的抗性。
通过酵母双杂交和VIGS研究发现,大豆转录延伸因子家族()蛋白基因能够增进SMV的复制,调控病原侵染;沉默该基因可降低SMV的积累,提高大豆对SMV的抗性[77]。敲除大豆真核翻译起始因子(),Chen等[78]研究发现植株减少了系统过敏性坏死反应,病毒积累增强,说明在介导的对SMV株系G7的系统过敏性坏死反应信号通路中起重要作用。
5 SMV抗病候选基因的调控网络
对介导的极端抗性(extreme resistance,ER)候选基因、和进行沉默,Zhang等[79]研究发现沉默后的植株表现感病,由介导的ER丧失。同时,通过VIGS研究发现基因、、、、和2个WRKY转录因子(和)都参与了介导的抗性[79]。基于上述研究结果并结合以前的研究,Zhang等[79]形成了一个由介导的ER调控网络:其中non-TIR-NBS-LRR基因及热激蛋白基因和伴侣蛋白基因和位于调控网络顶端,和脂肪酶位于的下游但在转录因子的上游,和最有可能在与和到转录因子的通路中是平行起作用的,最终表现对SMV的极端抗性(图4-a)。
Seo等[72]构建了一个介导对SMV的ER调控模型:在大豆最初感染的细胞中,SMV复制并产生效应因子。通过R蛋白识别,从而导致激素脱落酸(abscisic acid,ABA)在大豆体内的积累,进而激活ABA信号通路。ABA信号通路与介导的极端抗性相关联,通过上调信号传导正调控因子,进而刺激胼胝质沉积。最终,在胞间连丝处的胼胝质沉积抑制病毒从最初侵染的细胞向健康细胞的移动并限制病毒的积累[72,81](图4-b)。
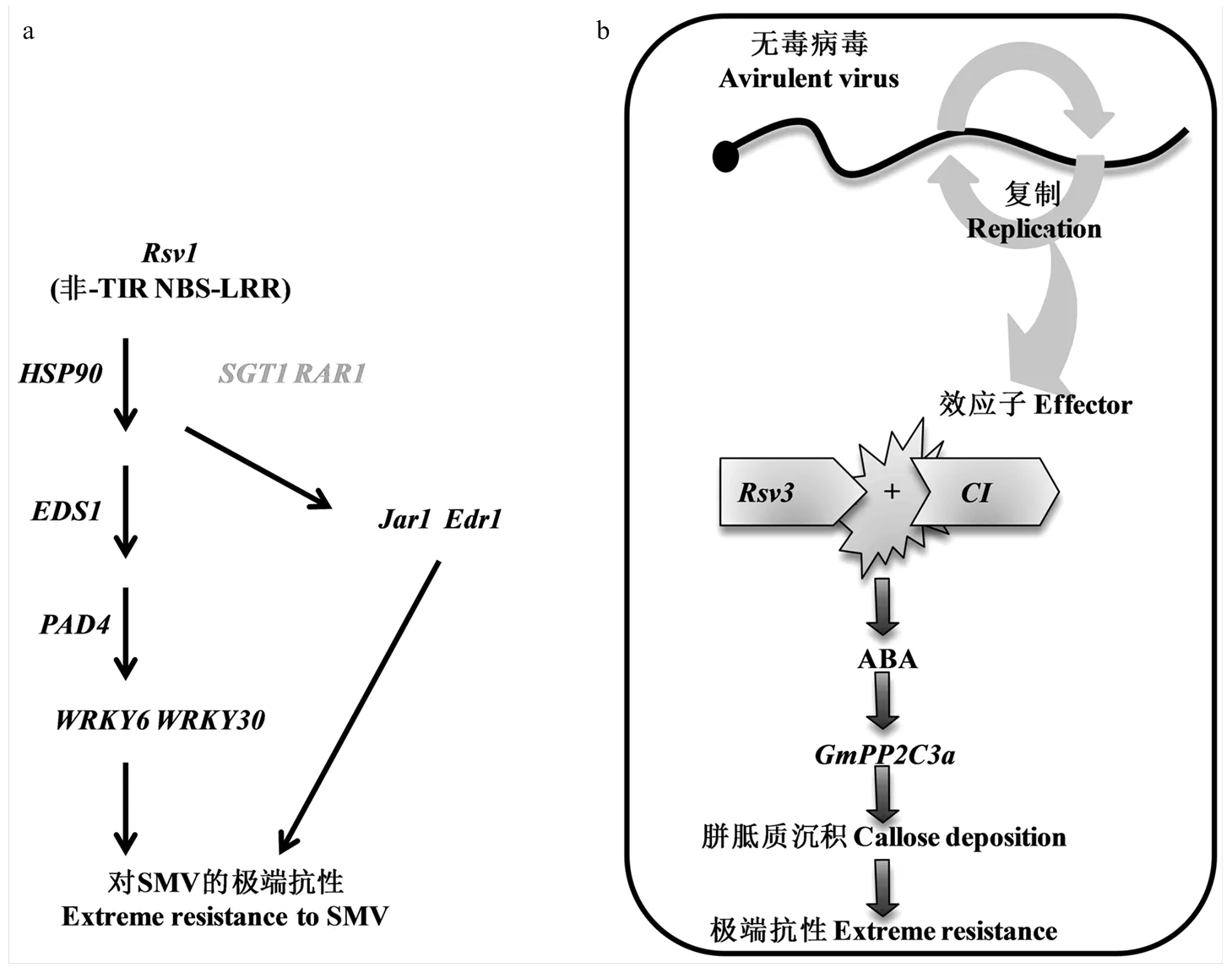
灰色字体的基因由Fu等[80]鉴定 Genes indicated in gray were identified by Fu et al.[80]
通过对转基因拟南芥进行转录组表达分析,He等[68]研究发现,该基因主要参与植物抗病的信号传导和免疫反应,而且在转基因大豆中发现另外一个与SMV抗性相关的基因也能够上调表达,表明这两个基因协同提高大豆对SMV的抗性。
6 展望
近年来,随着对大豆抗SMV研究的不断深入,在抗病基因的分子标记及候选基因的鉴定方面取得很大的进展。在大豆第2、6、13和14染色体上研究发现的SSR、SNP、Indel等分子标记与SMV抗病基因之间均紧密连锁或共分离,为分子标记辅助选择及分子设计育种奠定了良好的基础。Ma等[31]研究表明:单独使用标记Satt334或Sct_033对R在齐黄22×南农1138-2的3个世代以及对R在齐黄1号×南农1138的2个世代分子标记辅助选择的准确率均在85%以上,同时使用2个标记的准确率达95%以上。Saghai Maroof等[82]、Shi等[83]和Wang等[84]利用与SMV抗病基因紧密连锁的分子标记均成功地将分布在3条不同染色体上抗病基因精准地聚合在同一个大豆品系中,为广谱高抗SMV育种提供了优异的抗源材料。
齐黄1号对中国的18个SMV株系和美国的7个株系均具有抗性[53-55],其所携带的单显性抗病基因均定位在第13染色体上[11,32,39-40,45]。郑桂杰[85]收集整理骨干亲本齐黄1号的衍生品种(系)发现,齐黄1号在全国11个省市衍生出92个高产优质品种和优异品系,其中黄淮海地区衍生出87个品种(系)[86]。通过对90个衍生品种(系)接种SC3和SC7发现,生产上大面积推广的品种徐豆1号、跃进5号、中黄19、豫豆19和齐黄22等对两个株系均表现抗病[85]。此外,对SMV具有广谱抗性的科丰1号及大白麻的衍生品种在生产上的推广也对控制SMV的危害起到了关键的作用[87]。
然而,在日本和韩国相继发现了使具有单个抗病基因的品种失去抗性的SMV毒株[88-90],特别是在加拿大发现的SMV分离物L-RB能够攻克对美国所有7个株系都表现抗病的基因[91]。最近,WANG等[92]研究发现大豆花叶病毒P3蛋白单个氨基酸的改变就能使无毒株系攻克的抗性而成为致病株系。此外,中国报道的强致病株系及一些新发现的株系和重组株系也预示着SMV与大豆寄主之间一直处在动态平衡中[53-54,93-95]。因此,持续寻找新的抗病材料并发掘新的抗病基因是抗SMV育种的一个长期目标。
中国是大豆的起源地,不仅具有大量的栽培大豆类型,而且野生资源丰富多样,目前发现的野生大豆占世界90%以上[96]。Wen等[97]通过野生大豆与地方品种的遗传多样性比较发现,野生大豆中仅有51%的等位基因在地方大豆品种中保留下来,大豆的重测序研究结果也印证了这一发现[98]。可见,经过多次驯化和人工选择,野生大豆中大量优异的遗传变异没有保留在栽培大豆中[97-98]。因此,利用野生大豆资源,发掘新的SMV抗病基因,对减少SMV危害,提高抗SMV育种成效具有关键性的作用。
大豆抗SMV的研究为确定SMV抗病基因和阐明大豆抗SMV调控网络奠定了扎实的基础[27-28,42-46]。以往由于大豆遗传转化及再生比较困难,对SMV抗病基因的功能验证研究较少[68,73,76]。目前,随着现代生物技术的发展,利用不断完善的大豆转基因技术、候选基因沉默技术及CRISPR/Cas9基因编辑技术等,对标记定位的SMV抗病候选基因进行功能验证,对尽快确定抗SMV过程中的关键基因将发挥重要作用。此外,从SMV侵染大豆的角度出发,研究者筛选出一些能够协助SMV侵染大豆的基因,通过沉默这些基因发现,大豆增强了对SMV的抗性[77-78]。因此,后期如果能够通过CRISPR/Cas9基因编辑技术对研究发现的这些基因或一些新发现的负调控关键因子进行改造,将是培育高效广谱抗SMV大豆品种的一个新途径。
解决抗SMV育种最根本的方法是在明确SMV抗病基因功能的基础上,阐明大豆抗SMV过程中调控的信号网络。SMV抗病基因的标记定位及分析对理解大豆抗SMV的调控信号网络以及发掘新的大豆抗病基因具有重要价值。Zhang等[79]和Seo等[72]对SMV抗病基因和在抗病过程中的调控网络进行了构建和模拟,为进一步验证提供了重要线索。因此,探索大豆抗SMV的调控网络及对大豆的抗病性进行改良也是一个很重要的研究方向。
[1] 李凯, 刘志涛, 李海朝, 张锴, 王成坤, 任锐, 卢为国, 智海剑. 国家大豆区域试验品种对SMV和SCN的抗性分析. 大豆科学, 2013, 32(5): 670-675.
LI K, LIU Z T, Li H C, ZHANG K, WANG C K, REN R, LU W G, ZHI H J. Resistance to soybean mosaic virus and soybean cyst nematode of soybean cultivars from China national soybean uniform trials., 2013, 32(5): 670-675. (in Chinese)
[2] 王大刚, 智海剑, 张磊. 大豆抗大豆花叶病毒研究进展. 中国油料作物学报, 2013, 35(3): 341-348.
WANG D G, ZHI H J, ZHANG L. Review on resistance to soybean mosaic virus in soybean., 2013, 35(3): 341-348. (in Chinese)
[3] 李凯, 智海剑. 大豆对大豆花叶病毒病抗性的研究进展. 大豆科学, 2016, 35(4): 525-530.
LI K, ZHI H J. Advances in resistance to soybean mosaic virus disease in soybean., 2016, 35(4): 525-530. (in Chinese)
[4] 智海剑, 盖钧镒. 大豆对SMV数量抗性的表现形式与种质鉴定. 中国农业科学, 2004, 37(10): 1422-1427.
ZHI H J, GAI J Y. Performances and germplasm evaluation of quantitative resistance to soybean mosaic virus in soybeans., 2004, 37(10): 1422-1427. (in Chinese)
[5] 智海剑, 盖钧镒, 何小红. 大豆对SMV数量(程度)抗性的综合分级方法研究. 大豆科学, 2005, 24(2): 5-11.
ZHI H J, GAI J Y, HE X H. Study on methods of classification of quantitative resistance to soybean mosaic virus in soybean., 2005, 24(2): 5-11. (in Chinese)
[6] 郭丹丹, 陈海峰, 杨中路, 单志慧, 朱晓玲, 陈水莲, 周新安, 周蓉. 大豆对SMV SC-3株系的抗性遗传和QTL分析. 大豆科学, 2012, 31(4): 511-516.
GUO D D, CHEN H F, YANG Z L, SHAN Z H, ZHU X L, CHEN S L, ZHOU X A, ZHOU R. Analysis of resistance inheritance to soybean mosaic virus (SMV) strain SC-3 and mapping related QTL in soybean., 2012, 31(4): 511-516. (in Chinese)
[7] KIIHL R A S, HARTWIG E E. Inheritance of reaction to a soybean mosaic virus in soybeans., 1979, 19(3): 372-375.
[8] LIM S M. Resistance to soybean mosaic virus in soybeans., 1985, 75(2): 199-201.
[9] 陈怡, 杜维广, 栾晓燕, 张桂茹, 黄承运, 满为群, 谷秀芝, 王彬如. 大豆对两个大豆花叶病毒株系的抗性遗传研究. 黑龙江农业科学, 1991(5): 21-24.
CHEN Y, DU W G, LUAN X Y, ZHANG G R, HUANG C Y, MAN W Q, GU X Z, WANG B R. Inheritance of resistance to two soybean mosaic virus strains in soybean., 1991(5): 21-24. (in Chinese)
[10] 李海朝, 智海剑, 白丽, 杨华, 马莹, 刘宁, 王大刚. 大豆对SMV株系SC-11的抗性遗传及抗性基因的等位性研究. 大豆科学, 2006, 25(4): 365-368.
LI H C, ZHI H J, BAI L, YANG H, MA Y, LIU N, WANG D G. Studies on inheritance and allelism of resistance genes to SMV strain SC-11 in soybean., 2006, 25(4): 365-368. (in Chinese)
[11] 白丽, 李海朝, 马莹, 王大刚, 刘宁, 智海剑. 大豆对大豆花叶病毒SC11株系抗性的遗传及基因定位. 大豆科学, 2009, 28(1): 1-6.
BAI L, LI H C, MA Y, WANG D G, LIU N, ZHI H J. Inheritance and gene mapping of resistance to soybean mosaic virus strain SC11 in soybean., 2009, 28(1): 1-6. (in Chinese)
[12] 王大刚, 马莹, 刘宁, 郑桂杰, 杨中路, 杨永庆, 智海剑. 大豆花叶病毒(SMV)株系SC4和SC8的抗性遗传分析. 作物学报, 2012, 38(2): 202-209.
WANG D G, MA Y, LIU N, ZHENG G J, YANG Z L, YANG Y Q, ZHI H J. Inheritance of resistances to soybean mosaic virus strains SC4 and SC8 in soybean., 2012, 38(2): 202-209. (in Chinese)
[13] BUZZELL R I, TU J C. Inheritance of soybean resistance to soybean mosaic virus., 1984, 75(1): 82.
[14] CHEN P Y, BUSS G R, TOLIN S A. Resistance to soybean mosaic virus conferred by two independent dominant genes in PI486355., 1993, 84(1): 25-28.
[15] Yu Y G, SAGHAI MAROOF M A, BUSS G R, MAUGHAN P J, TOLIN S A. RFLP and microsatellite mapping of a gene for soybean mosaic virus resistance., 1994, 84(1): 60-64.
[16] GUNDUZ I, BUSS G R, MA G, CHEN P Y, TOLIN S A. Genetic analysis of resistance to soybean mosaic virus in OX670 and Harosoy soybean., 2001, 41(6): 1785-1791.
[17] GUNDUZ I, BUSS G R, CHEN P Y, TOLIN S A. Genetic and phenotypic analysis of soybean mosaic virus resistance in PI88788 soybean., 2004, 94(7): 687-692.
[18] GORE M A, HAYES A J, JEONG S C, YUE Y G, BUSS G R, SAGHAI MAROOF M A. Mapping tightly linked genes controllinginfection at theandregion in soybean., 2002, 45(3): 592-599.
[19] JEONG S C, KRISTIPATI S, HAYES A J, MAUGHAN P J, NOFFSINGER S L, GUNDUZ I, BUSS G R, SAGHAI MAROOF M A. Genetic and sequence analysis of markers tightly linked to the soybean mosaic virus resistance gene,., 2002, 42(1): 265-270.
[20] CHEN P Y, BUSS G R, TOLIN S A, GUNDUZ I, CICEK M. A valuable gene in Suweon 97 soybean for resistance to soybean mosaic virus., 2002, 42(2): 333-337.
[21] WANG Y, NELSON R L, HU Y. Genetic analysis of resistance to soybean mosaic virus in four soybean cultivars from China., 1998, 38(4): 922-925.
[22] CHEN P Y, MA G, BUSS G R, GUNDUZ I, ROANE C W, TOLIN S A. Inheritance and allelism tests of Raiden soybean for resistance to soybean mosaic virus., 2001, 92(1): 51-55.
[23] GUNDUZ I, BUSS G R, MA G, CHEN P Y, TOLIN S A. Genetic and phenotypic analysis of soybean mosaic virus resistance in PI88788 soybean.,2004, 94(7): 687-692.
[24] JEONG S C, SAGHAI MAROOGF M A. Detection and genotyping of SNPs tightly linked to two disease resistance loci,and, of soybean., 2004, 123(4): 305-310.
[25] SHI A N, CHEN P Y, LI D X, ZHENG C M, HOU A F, ZHANG B. Genetic confirmation of two independent genes for resistance to soybean mosaic virus in J05 soybean using SSR markers., 2008, 99(6): 598-603.
[26] SUH S J, BOWMAN B C, JEONG N, YANG K, KASTL C, TOLIN S A, SAGHAI MAROOF M A, JEONG S C. Thelocus conferring resistance to soybean mosaic virus is associated with a cluster of coiled-coil nucleotide-binding leucine-rich repeat genes., 2011, 4(1): 55-64.
[27] ILUT D C, LIPKA A E, JEONG N, BAE D N, KIM D H, KIM J H, REDEKAR N, YANG K, PARK W, KANG S T, KIM N, MOON J K, SAGHAI MAROOF M A, GORE M A, JEONG S C. Identification of haplotypes at thegenomic region in soybean associated with durable resistance to soybean mosaic virus., 2016, 129(3): 453-468.
[28] KLEPADLO M, CHEN P Y, SHI A N, MASON R E, KORTH K L, SRIVASTAVA V S, WU C J. Two tightly linked genes for soybean mosaic virus resistance in soybean., 2017, 57(4): 1-10.
[29] FU S X, ZHAN Y, ZHI H J, GAI Y J, YU D Y. Mapping of SMV resistance geneby SSR markers in soybean., 2006, 128(1/3): 63-69.
[30] LI H C, ZHI H J, GAI J Y, GUO D Q, WANG Y W, LI K, LI B, YANG H. Inheritance and gene mapping of resistance to soybean mosaic virus strain SC14 in soybean., 2006, 48(12): 1466-1472.
[31] MA Y, LI H C, WANG D G, LIU N, ZHI H J. Molecular mapping and marker assisted selection of soybean mosaic virus resistance geneRLegume Genomics & Genetics, 2010, 1(8): 41-46.
[32] MA Y, WANG D G, LI H C, ZHENG G J, YANG Y Q, LI H W, ZHI H J. Fine mapping of theRlocus for resistance to soybean mosaic virus in soybean., 2011, 181(1): 127-135.
[33] WANG D G, MA Y, YANG Y Q, LIU N, LI C Y, SONG Y P, ZHI H J. Fine mapping and analyses ofRresistance candidate genes to soybean mosaic virus in soybean.&, 2011, 122(3): 555-565.
[34] WANG D G, MA Y, LIU N, YANG Z L, ZHENG G J, ZHI H J. Fine mapping and identification of the soybeanRresistance candidate gene to soybean mosaic virus., 2011, 130(6): 653-659.
[35] YANG Q H, GAI J Y. Identification, inheritance and gene mapping of resistance to a virulent soybean mosaic virus strain SC15 in soybean., 2011, 130(2): 128-132.
[36] 李春燕, 杨永庆, 王大刚, 李华伟, 郑桂杰, 王涛, 智海剑. 大豆对SMV株系SC10的抗性遗传及抗病基因的定位研究. 中国农业科学, 2012, 45(21): 4335-4342.
LI C Y, YANG Y Q, WANG D G, LI H W, ZHENG G J, WANG T, ZHI H J. Studies on mapping and inheritance of resistance genes to SMV strain SC10 in soybean., 2012, 45(21): 4335-4342. (in Chinese)
[37] YANG Y Q, ZHENG G J, HAN L, WANG D G, YANG X F, YUAN Y, HUANG S H, ZHI H J. Genetic analysis and mapping of genes for resistance to multiple strains of soybean mosaic virus in a single resistant soybean accession PI96983.&, 2013, 126(7): 1783-1791.
[38] 阳小凤, 杨永庆, 郑桂杰, 智海剑, 李晓红. 大豆对大豆花叶病毒株系SC6和SC17抗病基因的精细定位. 作物学报, 2013, 39(2): 216-221.
YANG X F, YANG Y Q, ZHENG G J, ZHI H J, LI X H. Fine mapping of resistance genes to SMV strains SC6 and SC17 in soybean., 2013, 39(2): 216-221. (in Chinese)
[39] ZHENG G J, YANG Y Q, MA Y, YANG X F, CHEN S Y, REN R, WANG D G, YANG Z L, ZHI H J. Fine mapping and candidate gene analysis of resistance geneRto soybean mosaic virus in Qihuang 1., 2014, 13(12): 2608-2615.
[40] LI K, REN R, ADHIMOOLAM K, GAO L, YUAN Y, LIU Z T, ZHONG Y K, ZHI H J. Genetic analysis and identification of two soybean mosaic virus resistance genes in soybean [(L.) Merr]., 2015, 134(6): 684-695.
[41] YAN H L, WANG H, CHENG H, HU Z B, CHU S S, ZHANG G Z, YU D Y. Detection and fine-mapping of SC7 resistance genes via linkage and association analysis in soybean.2015, 57(8): 722-729.
[42] LI C, ADHIMOOLAM K, YUAN Y, YIN J L, REN R, YANG Y Q, ZHI H J. Identification of candidate genes for resistance to soybean mosaic virus strain SC3by using fine mapping and transcriptome analyses., 2017, 68(2): 156-166.
[43] ZHAO L, WANG D G, ZHANG H Y, SHEN Y C, YANG Y Q, LI K, WANG L Q, YANG Y H, ZHI H J. Fine mapping of theRlocus and expression analysis of candidate SMV resistance genes in soybean., 2016, 135(6): 701-706.
[44] ADHIMOOLAM K, LI K, JIANG H, REN R, LI G, ZHI H J, CHEN S Y, GAI J Y. Inheritance, fine-mapping, and candidate gene analyses of resistance to soybean mosaic virus strain SC5in soybean., 2017, 292(4): 811-822.
[45] ADHIMOOLAM K, LI K, LI C, YIN J L, LI N, YANG Y H, SONG Y P, REN R, ZHI H J, GAI J Y. Fine-mapping and identifying candidate genes conferring resistance to soybean mosaic virus strain SC20 in soybean., 2018, 131(2): 461-476.
[46] REN R, LIU S C, ADHIMOOLAM K, WANG T, NIU H P, YIN J L, YANG Y H, WANG L Q, YANG Q H, ZHI H J, LI K. Fine-mapping and identification of a novel locusRscunderlying soybean resistance to soybean mosaic virus., 2017, 130(11): 2395-2410.
[47] 李凯, 任锐, 王涛, 高乐, 落金艳, 刘士超, 智海剑, 盖钧镒. 大豆对大豆花叶病毒SC18株系的抗性遗传和基因定位. 大豆科学, 2017, 36(2): 187-192.
LI K, REN R, WANG T, GAO L, LUO J Y, LIU S C, ZHI H J, GAI J Y. Genetic analysis and mapping of soybean mosaic virus resistance genes to SC18 in soybean., 2017, 36(2): 187-192. (in Chinese)
[48] MA F F, WU X Y, CHEN Y X, LIU Y N, SHAO Z Q, WU P, WU M, LIU C C, WU W P, YANG J Y, LI D X, CHEN J Q, WANG B. Fine mapping of thegene in the soybean cultivar Suweon 97 that confers resistance to two Chinese strains of the soybean mosaic virus., 2016, 129(11): 2227-2236.
[49] HAYES A J, MA G, BUSS G R, SAGHAI MAROOF A S. Molecular marker mapping of, a gene conferring resistance to all known strains of soybean mosaic virus., 2000, 40(5): 1434-1437.
[50] HWANG T Y, MOON J K, YU S, YANG K, MOHANKUMAR S, YU Y H, LEE Y H, KIM S H, KIM H M, SAGHAI MAROOF M A, JEONG S C. Application of comparative genomics in developing molecular markers tightly linked to the virus resistance gene, 2006, 49(4): 380-388.
[51] SAGHAI MAROOF M A, TUCKER D M, SKONECZKA J A, BOWMEN B C, TRIPATHY S, TOLIN S A. Fine mapping and candidate gene discovery of the soybean mosaic virus resistance gene,., 2010, 3(1): 14-22.
[52] CHEN P Y, BUSS G R, ROANE C W, TOLIN S A. Allelism among genes for resistance to soybean mosaic virus in strain-differential soybean cultivars., 1991, 31(2): 305-309.
[53] LI K, YANG Q H, ZHI H J, GAI J Y. Identification and distribution of soybean mosaic virus strains in Southern China., 2010, 94(3): 351-357.
[54] 王大刚, 田震, 李凯, 李华伟, 黄志平, 胡国玉, 张磊, 智海剑. 鲁豫皖大豆产区大豆花叶病毒株系的鉴定及动态变化分析. 大豆科学, 2013, 32(6): 806-809.
WANG D G, TIAN Z, LI K, LI H W, HUANG Z P, HU G Y, ZHANG L, ZHI H J. Identification and variation analysis of soybean mosaic virus strains in Shandong, Henan and Anhui provinces of China., 2013, 32(6): 806-809. (in Chinese)
[55] 蔡春梅, 姜骁, 赵春梅, 马建新. 中国大豆花叶病毒SC7外壳蛋白基因的序列测定及其与美国株系的比较. 病毒学报, 2014, 30(5): 489-494.
CAI C M, JIANG X, ZHAO C M, MA J X. Sequence analysis of the coat protein gene of Chinese soybean mosaic virus strain SC7and comparison with those of SMV strains from the USA., 2014, 30(5): 489-494. (in Chinese)
[56] 郭东全, 王延伟, 智海剑, 盖钧镒, 李海朝, 李凯. 大豆对SMV SC-13株系群的抗性遗传及基因定位的研究. 大豆科学, 2007, 26(1): 21-24.
GUO D Q, WANG Y W, ZHI H J, GAI J Y, LI H C, LI K. Inheritance and gene mapping of resistance to SMV strain group SC-13in soybean., 2007, 26(1): 21-24. (in Chinese)
[57] CHE Z J, LIU H L, YI F L, CHENG H, YANG Y M, WANG L, DU J Y, ZHANG P P, WANG J, YU D Y. Genome-wide association study reveals novel loci for SC7 resistance in a soybean mutant panel., 2017, 8: 1-12.
[58] HAYES A J, JEONG S C, GORE M A, YU Y G, BUSS G R, TOLIN S A, SAGHAI MAROOF M A. Recombination within a nucleotide- binding-site/leucine-rich-repeat gene cluster produces new variants conditioning resistance to soybean mosaic virus in soybeans.,2004, 166(1): 493-503.
[59] SHAO Z Q, ZHANG Y M, HANG Y Y, XUE J Y, ZHOU G C, WU P, WU X Y, WU X Z, WANG Q, WANG B, CHEN J Q. Long-term evolution of nucleotide-binding site-leucine-rich repeat genes: understanding gained from and beyond the legume family., 2014, 166(1): 217-234.
[60] REDEKAR N R, CLEBINGER E M, LASKAR M A, BIYASHEV R M, ASHFIELD T, JENSEN R V, JEONG S C, TOLIN S A, SAGHAI MAROOF M A. Candidate gene sequence analyses toward identifying-type resistance to soybean mosaic virus., 2016, 9(2): 1-12.
[61] 贺超英, 张志永, 陈受宜. 大豆中NBS类抗性基因同源序列的分离与鉴定. 科学通报, 2001, 46(12): 1017-1021.
HE C Y, ZHANG Z Y, CHEN S Y. Isolation and identification of NBS type resistance gene analogs in soybean., 2001, 46(12): 1017-1021. (in Chinese)
[62] WANG B J, WANG Y J, WANG Q, LUO G Z, ZHANG Z G, HE C Y, HE S J, ZHANG J S, GAI J Y, CHEN S Y. Characterization of an NBS-LRR resistance gene homologue from soybean., 2004, 161(7): 815-822.
[63] HE C Y, TIAN A G, ZHANG J S, ZHANG Z Y, GAI J Y, CHEN S Y. Isolation and characterization of a full-length resistance gene homolog from soybean., 2003, 106(5): 786-793.
[64] WANG Y J, LI Y D, TIAN A G, WANG H WM ZHANG J S, CHR S Y. Cloning and characterization of an HDZip I genefrom soybean., 2005, 221(6): 831-843.
[65] CHENG H, YANG H, ZHANG D, GAI J Y, YU D Y. Polymorphisms of soybean isoflavone synthase and flavanone 3-hydroxylase genes are associated with soybean mosaic virus resistance., 2010, 25(1): 13-24.
[66] ZHANG G Y, CHEN M, CHEN X P, XU Z S, LI L C, GUO J M, MA Y Z. Isolation and characterization of a novel EAR-motif-containing genefrom soybean (L.)., 2010, 37(2): 809-818.
[67] 陈华涛, 陈新, 顾和平, 张红梅, 袁星星, 崔晓燕. 大豆GmPR10基因克隆与植物表达载体的构建. 江苏农业学报, 2011, 27(3): 494-499.
CHEN H T, CHEN X, GU H P, ZHANG H M, YUAN X X, CUI X Y. Cloning and plant expression vector construction of GmPR10 in soybean., 2011, 27(3): 494-499. (in Chinese)
[68] HE H L, YANG X D, XUN H W, LOU X , LI S Z, ZHANG Z B, JIANG L L, DONG Y S, WANG S C, PANG J S, LIU B. Over-expression ofenhances virus resistance inand soybean., 2017, 36(9): 1441-1455.
[69] LIU J Z, HORSTMAN H D, BRAUM E, GRAHAM M A, ZHANG C, NAVARRE D, QIU L W, LEE Y, NETTLETON D, HILL J H, WHITHAM S A. Soybean homologs ofnegatively regulate defense responses and positively regulate growth and development.,2011, 157(3): 1363-1378.
[70] LIU J Z, BRAUN E, QIU W L, SHI Y F, MARCELINO- GUIMARAES F C, NAVARRE D, HILL J H, WHITHAM S A. Positive, and negative roles for soybean MPK6 in regulating defense responses., 2014, 27(8): 824-834.
[71] LIU J Z, WHITHAM S A. Overexpression of a soybean nuclear localized type-III DnaJ domain-containing HSP40 reveals its roles in cell death and disease resistance., 2013, 74(1): 110-121.
[72] SEO J K, KWON S J, CHO W K, CHOI H S, KIM K H. Type 2C protein phosphatase is a key regulator of antiviral extreme resistance limiting virus spread., 2014, 4: 5905-5913.
[73] ZHOU L, HE H L, LIU R F, HAN Q, SHOU H X, LIU B. Overexpression ofpotassium channel enhances resistance to soybean mosaic virus., 2014, 14(1): 154-165.
[74] ZHOU Z, HE H L, MA L P, YU X Q, MI Q, PANG J S, TANG G X, LIU B. Overexpression of agene enhanced activity of nitrate reductase and aldehyde oxidase, and boosted mosaic virus resistance in soybean., 2015, 10(4): 1-15.
[75] LI N, YIN J L, LI C, WANG D G, YANG Y Q, ADHIMOOLAM K, LUAN H X, ZHI H J. NB-LRR gene family required for- mediated resistance to soybean mosaic virus., 2016, 67(5): 541-552.
[76] TRAN P T, WIDYASARI K, SEO J K, KIM K H. Isolation and validation of a candidategene from a soybean genotype that confers strain-specific resistance to soybean mosaic virus., 2017, 513: 153-159.
[77] LUAN H X, SHINE M B, CUI X Y, CHEN X, MA N, KACHROO P, ZHI H J, KARCHROO A. The potyviral P3 protein targets eukaryotic elongation factor 1A to promote the unfolded protein response and viral pathogenesis., 2016, 172(1): 221-234.
[78] CHEN H, ARSOVSKI A A, Yu K F, Wang A M. Deep sequencing leads to the identification of eukaryotic translation initiation factor 5a as a key element in-mediated lethal systemic hypersensitive response to soybean mosaic virus infection in soybean., 2016, 18(3): 391-404.
[79] ZHANG C Q, GROSIC S, WHITHAM S A, HILL J H. The requirement of multiple defense genes in soybean-mediated extreme resistance to soybean mosaic virus., 2012, 25(10): 1307-1313.
[80] FU D Q, GHABRIAL S, KACHROO A.andare required for basal, R gene-mediated and systemic acquired resistance in soybean., 2009, 22(1): 86-95.
[81] LI W L, ZHAO Y S, LIU C J, YAO G B, WU S S, HOU C Y, ZHANG M C, WANG D M. Callose deposition at plasmodesmata is a critical factor in restricting the cell-to-cell movement of soybean mosaic virus., 2012, 31(5): 905-916.
[82] SAGHAI MAROOF M A, JEONG S C, GUNDUZ I, TOLIN S. Pyramiding of soybean mosaic virus resistance genes by marker-assisted selection., 2008, 48(2): 517-526.
[83] SHI A N, CHEN P Y, LI D X, ZHENG C M, ZHANG B, HOU A F. Pyramiding multiple genes for resistance to soybean mosaic virus in soybean using molecular markers., 2009, 23(1): 113-124.
[84] WANG D G, ZHAO L, LI K, MA Y, WANG L Q, YANG Y Q, YANG Q H, ZHI H J. Marker-assisted pyramiding of soybean resistance genesR,R, andRto soybean mosaic virus., 2017, 16(11): 2413-2420.
[85] 郑桂杰. 齐黄1号对大豆花叶病毒抗性基因的精细定位、育种应用及候选基因表达分析[D]. 南京: 南京农业大学, 2012.
ZHENG G J. Fine mapping, expression analysis and breeding application of resistance genes to soybean mosaic virus in Qihuang No.1[D]. Nanjing: Nanjing Agriculture University, 2012. (in Chinese)
[86] 徐冉, 石传娥, 张礼凤, 王彩洁, 聂翠琴, 李建和. 黄淮海大豆优异种质齐黄1号的育种应用. 植物遗传资源学报, 2004, 5(2): 170-175.
XU R, SHI C E, ZHANG L F, WANG C J, NIE C Q, LI J H. Utilization of Qihuang 1 in soybean breeding in the Huanghuaihai region., 2004, 5(2): 170-175. (in Chinese)
[87] 张孟臣, 张磊, 刘学义, 李卫东. 黄淮海大豆改良种质. 北京: 中国农业出版社, 2014.
ZHANG M C, ZHANG L, LIU X Y, LI W D.. Beijing: China Agriculture Press, 2014. (in Chinese)
[88] CHOI B K, KOO J M, AHN H J, YUM H J, CHOI C W, RYU K H, CHEN P Y, TOLIN S A. Emergence of-resistance breaking soybean mosaic virus isolates from Korean soybean cultivars., 2005, 112(1): 42-51.
[89] KOO J M, CHOI B K, AHN H J, YUM H J, CHOI C W. First report of anresistance-breaking isolate of soybean mosaic virus in Korea., 2005, 54(4): 573.
[90] SARUTA M, KIKUCHI A, OKABE A, SASAYA T. Molecular characterization of A2 and D strains of soybean mosaic virus, which caused a recent virus outbreak in soybean cultivar Sachiyutaka in Chugoku and Shikoku regions of Japan., 2005, 71(6): 431-435.
[91] GAGARINOVA A G, BABU M, POYSA V, HILL J H, WANG A M. Identification and molecular characterization of two naturally occurring soybean mosaic virus isolates that are closely related but differ in their ability to overcomeresistance., 2008, 138(1/2): 50-56.
[92] WANG Y, HAJIMORAD M R. Gain of virulence by soybean mosaic virus on-genotype soybeans is associated with a relative fitness loss in a susceptible host., 2016, 17(7): 1154-1159.
[93] YANG Y Q, LIN J, ZHENG G J, ZHANG M C, ZHI H J. Recombinant soybean mosaic virus is prevalent in Chinese soybean fields., 159(7): 1793-1796.
[94] YANG Y Q, GONG J W, LI H W, LI C Y, WANG D G, LI K, ZHI H J. Identification of a novel soybean mosaic virus isolate in China that contains a unique5' terminus sharing high sequence homology with bean common mosaic virus., 2011, 157(1): 13-18.
[95] ZHOU G C, SHAO Z Q, MA F F, WU P, WU X Y, XIA Z Y, YU D Y, CHENG H, LIU Z H, JIANG Z F, CHEN Q S, WANG B, CHEN J Q. The evolution of soybean mosaic virus: An updated analysis by obtaining 18 new genomic sequences of Chinese strains/isolates., 2015, 208: 189-198.
[96] 董英山. 中国野生大豆研究进展. 吉林农业大学学报, 2008, 30(4): 394-400.
DONG Y S. Advances of research on wild soybean in China., 2008, 30(4): 394-400. (in Chinese)
[97] WEN Z X, DING Y L, ZHAO T J, GAI J Y. Genetic diversity and peculiarity of annual wild soybean (Sieb. et Zucc.) from various eco-regions in China., 2009, 119(2): 371-381.
[98] ZHOU Z K, JIANG Y, WANG Z, GOU Z H, YU J L, LI W Y, YU Y J, SHU L P, ZHAO Y J, MA Y M, FANG C, SHEN Y T, LIU T F, LI C C, GAO Y D, XIANG H, ZHU B G, LEE S H, WANG W, TIAN Z X. Resequencing 302 wild and cultivated accessions identifies genes related to domestication and improvement in soybean., 2015, 33(4): 408-414.
(责任编辑 李莉)
Progresses of Resistance on Soybean Mosaic Virus in Soybean
WANG DaGang1, LI Kai2, ZHI HaiJian2
(1Crop Institute of Anhui Academy of Agricultural Sciences/Key Laboratory of Crop Quality Improvement of Anhui Province, Hefei 230031;2Soybean Research Institute of Nanjing Agriculture University/National Center for Soybean Improvement/ National Key Laboratory for Crop Genetics and Germplasm Enhancement, Nanjing 210095)
Soybean mosaic virus disease, caused by soybean mosaic virus (SMV), is one of the most serous pathogens of soybean ((L.) Merr.), which give rise to the loss of yield and quality in soybean production worldwide. During the recent decade, the studies on soybean against SMV stress have made some progress, includes the mapping of SMV resistant genes, the functional analysis of candidate resistance genes, and the progress in dissecting the SMV resistant signaling pathways in soybean. There are two kinds of resistance to SMV in soybean, quantitative resistance and qualitative resistance. The quantitative resistance to SMV is controlled by an additive major gene plus additive-dominant polygenes, and the qualitative resistance is mainly controlled by a dominant resistance gene. Research showed that the quantitative trait loci (QTL) were mainly on chromosome 6, 10 and 13. So far, 22 SMV qualitative resistance loci have been successively mapped on soybean chromosome 2, 6, 13 and 14. And most of the physical distance between the markers on both sides of the SMV resistance gene loci is within 1 Mb. Among them, there are 8 gene loci (,R,R,RandRetc.) located on chromosome 2, 10 loci (,,R,RandRetc.) on chromosome 13, 2 loci on chromosome 6 (RandR) and 14 (andR), respectively. According to the Williams 82 whole genomic sequence (http://www.phytozome.net/soybean), the putative candidate genes of SMV qualitative resistance loci were subsequently narrowed down based on their predicted functions, expression patterns and sequence comparison etc. Among these genes,,,,,,andet al. eight genes were considered potential resistance candidate genes to SMV on chromosome 2;as the most promising candidate gene on chromosome 6;,,,,,,,,et al. nine genes and,,,,,et al. six genes were the putative candidate genes to SMV on chromosome 13 and 14, respectively. Using virus induced gene silencing (VIGS) and transgenic technology etc. analyzed the functions of the candidate genes. The results showed genes,andcan participate to SMV resistance in soybean as positive regulator. After knocked downand, viral accumulation level of SMV and pathogenicity increased. They were considered negative regulator to SMV resistance. Based on the research results of SMV resistance gene, regulated model were built forandmediating extreme resistance (ER) against SMV.-mediated ER has provided new insight into the soybean signaling network required for ER against SMV. The primary mechanism of-mediated ER against viruses was the inhibition of viral cell-to-cell movement by callose deposition in an ABA signaling-dependent manner. In this report, the research progress on the mapping and function analysis of SMV resistance genes and the future research directions of SMV resistance in soybean are summarized. It will provide a basis for molecular design breeding and mechanism research of resistance genes to SMV in soybean.
soybean; soybean mosaic virus; resistance gene; marker location; functional research
2018-04-07;
2018-06-05
国家自然科学基金(31571687、31571690和31201235)、安徽省自然科学基金(1708085MC69)、国家大豆产业技术体系建设专项资金(CARS-04)、江苏省现代作物生产协同创新中心(JCIC-MCP)
王大刚,Tel:0551-65149851;E-mail:smvwang@163.com。
智海剑,Tel:025-84396463;E-mail:zhj@njau.edu.cn
10.3864/j.issn.0578-1752.2018.16.002
