CRISPR/Cas9系统中sgRNA设计与脱靶效应评估
谢胜松,张懿,张利生,李广磊,赵长志,倪攀,赵书红
1. 华中农业大学,农业动物遗传育种与繁殖教育部重点实验室,武汉 430070;
2. 中国人民解放军第161医院妇产科,武汉 430010
CRISPR/Cas9系统中sgRNA设计与脱靶效应评估
谢胜松1,张懿2,张利生1,李广磊1,赵长志1,倪攀1,赵书红1
1. 华中农业大学,农业动物遗传育种与繁殖教育部重点实验室,武汉 430070;
2. 中国人民解放军第161医院妇产科,武汉 430010
基于CRISPR/Cas9系统介导的第三代基因组编辑技术,已成功应用于动物、植物和微生物等诸多物种的基因组改造。如何提高CRISPR/Cas9技术的基因组编辑效率和最大限度降低脱靶风险一直是本领域的研究热点,而使用高效且特异的sgRNA(Small guide RNA)是基因组改造成功的关键性因素之一。目前,已有多款针对CRISPR/Cas9技术的sgRNA设计和/或脱靶效应评估软件,但不同的软件各有优缺点。本文重点对16款sgRNA 设计和脱靶效应评估在线和单机版软件的特点进行了阐述,通过制定38项评估指标对不同软件进行了比较分析,最后对11种用于检测基因组编辑效率和脱靶的实验方法,以及如何筛选高效且特异的sgRNA进行了归纳总结。
CRISPR/Cas9系统;基因组编辑;sgRNA;脱靶效应
基于CRISPR(Clustered regularly interspaced short palindromic repeats)/Cas9系统介导的基因组编辑技术,是继锌指核酸酶(Zinc-finger nucleases, ZFNs)和类转录激活因子效应物核酸酶(Transcription activator-like effector nuclease, TALEN)后的第三代基因组编辑技术,主要是源于对细菌的获得性免疫系统改造而成[1]。一经问世,在食蟹猴(Cynomolgus monkey)、小鼠(Mus musculus)、大鼠(Rattus norveaicus)、斑马鱼(Zebrafish)、猪(Sus scrofa)、拟南芥(Arabidopsis thaliala)、烟草(Nicotiana tabacum)、高粱(Sorghum bicolor)和水稻(Oryza sativa)等多个物种中实现了基因组编辑[2~10]。2014年,Nishimasu等[11]解析了Cas9、sgRNA(Small guide RNA)和DNA复合体的晶体结构。研究显示,Cas9核酸酶由两个裂片(lobe)组成:一个参与识别sgRNA,另一个负责切割靶标DNA,导致靶基因功能失活。鉴于此,CRISPR/Cas9技术主要由两部分组成:一是sgRNA,通过碱基互补配对与基因组特异结合;其次是Cas9核酸酶,可靶定到具有下游前间区序列邻近基序(Protospacer adjacent motif, PAM)的特定基因组序列并进行切割[12]。
目前,CRISPR/Cas9技术依然在不断改进中。如图1所示,野生型Cas9可切割DNA并激活DNA双链断裂(DNA double-strand breaks, DSB)修复机制,即同源重组(Homologous recombination, HR)、非同源末端连接(Non-homologous end Joining, NHEJ)机制和单链DNA退火(Single strand annealing, SSA)修复机制。如果无同源供体DNA(Donor DNA)模板,细胞会通过NHEJ修复突变位点,导致靶标DNA发生碱基插入或删除。相反,若存在同源供体DNA或ssDNA(Single-stranded DNA)寡核苷酸,细胞会采用HR或SSA修复机制,准确将外源DNA插入到基因组(图1A)。当仅突变Cas9核酸酶一个功能结构域时,可形成Cas9单切口酶(nickase,通常为D10A或H840A),仅能切割单链DNA。当单切口酶与1对分别位于染色体正负链上、方向相反且距离适当的两个sgRNA(即“paired-gRNA”)共同作用时,可切割靶标DNA,进而导致靶基因碱基缺失。研究表明,该方法能显著降低脱靶风险(图1B)。而当同时突变Cas9核酸酶两个功能结构域后,可使其成为缺陷型核酸酶(Catalytically inactive CRISPR-associated nuclease 9, short for dead Cas9 or dCas9),将仅具备与DNA结合的能力。当dCas9与其他转录调控蛋白融合,如VP16(alpha TIF)或KRAB(Krueppel-associated box),可激活或抑制靶基因的表达[13];dCas9与绿色荧光蛋白(Green fluorescent protein, GFP)融合,则可应用于示踪特异靶位点在基因组上的位置(图1C)。CRISPR/Cas9技术已成功应用于多个物种,并展现出广阔的应用前景。但如何改善和提高基因组编辑效率,同时最大限度降低脱靶风险成为亟待解决的问题,本文围绕这一核心问题进行了综述,并重点介绍了sgRNA的设计要点。

图1 基于CRISPR/Cas9系统介导的基因组定点编辑技术
1 CRISPR/Cas9系统介导的基因组编辑效率与脱靶效应
CRISPR/Cas9系统介导的基因组编辑效率与多种因素有关。Hruscha等[14]利用该技术在斑马鱼中进行基因打靶,发现其基因组编辑效率高达86.0%。如果提供同源供体DNA,通过HR修复机制可将外源性的HA标签插入到特定靶点,插入效率为3.5%~15.6%。Doench等[15]系统性研究了CRISPR/Cas9系统介导的基因组编辑效率,对1841条sgRNA进行比较,发现不同sgRNA的活性有差别。例如,若sgRNA的3'末端第20位碱基为鸟嘌呤核苷酸,则基因组编辑效率高;如果该位置为胞嘧啶核苷酸,则基因组编辑效率低;如果第18和19位碱基为胸腺嘧啶核苷酸,其基因组编辑效率则相对较低。另外,如果sgRNA第16位碱基为胞嘧啶核苷酸,其基因组编辑效率则相对较高;如果该位置为鸟嘌呤核苷酸,其基因组编辑效率则低;如果sgRNA第3位碱基为腺嘌呤核苷酸,其基因组编辑效率比该位置为胞嘧啶核苷酸时高。这一研究为设计高效的sgRNA提供了强大的实验数据支持。利用GFP报告基因载体系统,Zhang等[16]分别通过定性和定量的方法系统地比较了PAM对基因组编辑效率的影响,发现编辑效率依次是NGG>NGA>NAG,(N= A,T, C或G)。Farboud等[17]发现并非所有sgRNA有活性;研究还发现选择3'末端为GG的sgRNA,可显著提高基因组编辑效率。除了sgRNA外,选择特定的小分子化合物也能提高基因组编辑效率。Yu等[18]利用高通量药物筛选的策略,发现使用L755507(β3肾上腺素受体激动剂)或Brefeldin A(蛋白转运抑制剂),可将大片段同源重组效率和基因插入效率分别提高3倍和2倍;而使用两种胸苷类似物(Thymidine analogs),即叠氮胸苷(Azidothymidine, AZT)和曲氟尿苷(Trifluridine, TFT)则能降低同源重组效率。
多项研究表明,CRISPR/Cas9技术存在一定的脱靶效应。Fu等[19]利用GFP报告载体系统和细胞流式技术系统评估了脱靶效应。研究发现,Cas9核酸酶对脱靶位点1~2个错配碱基的耐受能力与其配对位置有关。他们还发现含5个错配碱基的脱靶位点能被Cas9核酸酶切割。Hsu等[20]在HEK 293T(Human embryonic kidney 293T cells)和HEK 293FT细胞系中,对超过700个sgRNA和对应的脱靶位点进行了验证。结果发现,Cas9核酸酶对错配碱基的耐受能力,不仅与错配碱基数量有关,还与错配碱基位置有关,并可将sgRNA划分为靠近PAM的核心区和远离PAM的非核心区,这与Fu等[19]的研究报道一致。此外,研究还发现,Cas9核酸酶切割靶标基因组不受甲基化影响。通过调节Cas9核酸酶和sgRNA浓度可降低脱靶风险,但浓度降低后,相应基因组编辑能力会减弱[20]。Pattanayak等[21]利用高通量测序方法,对sgRNA的特异性进行了评估,发现sgRNA的特异性主要与临近PAM的8~12个序列有关,即种子序列(Seed sequence)。与之相矛盾的是,Zhang等[22]发现脱靶效应与靶标结合位点无关,而与sgRNA和Cas9核酸酶的类型有关。Ran等[23]利用Cas9单切口酶和“paired-gRNAs”进行基因组编辑,结果表明此方法能显著降低细胞系中的脱靶效应(约50~1500倍)。Lin等[24]研究发现,脱靶位点即便存在一个凸起(bulge)的碱基,依然会发生脱靶。Wang等[25]利用整合酶缺陷的慢病毒载体(Integrase-defective lentiviral vectors,IDLVs)对CRISPR/Cas9技术的脱靶效应进行全基因组无偏向性分析,同样发现Cas9核酸酶可切割含单个凸起碱基的脱靶位点。而令人意外地是,发现某些脱靶位点竟含有多达13个错配的碱基。由此可见,CRISPR/Cas9技术存在严重的脱靶风险。为了提高sgRNA特异性,Fu等[26]采用长度短于20nt的sgRNA进行基因打靶研究,发现使用17~18nt的“truncated sgRNA”不影响其活性,但可显著降低脱靶风险。
2 sgRNA设计与脱靶效应评估软件
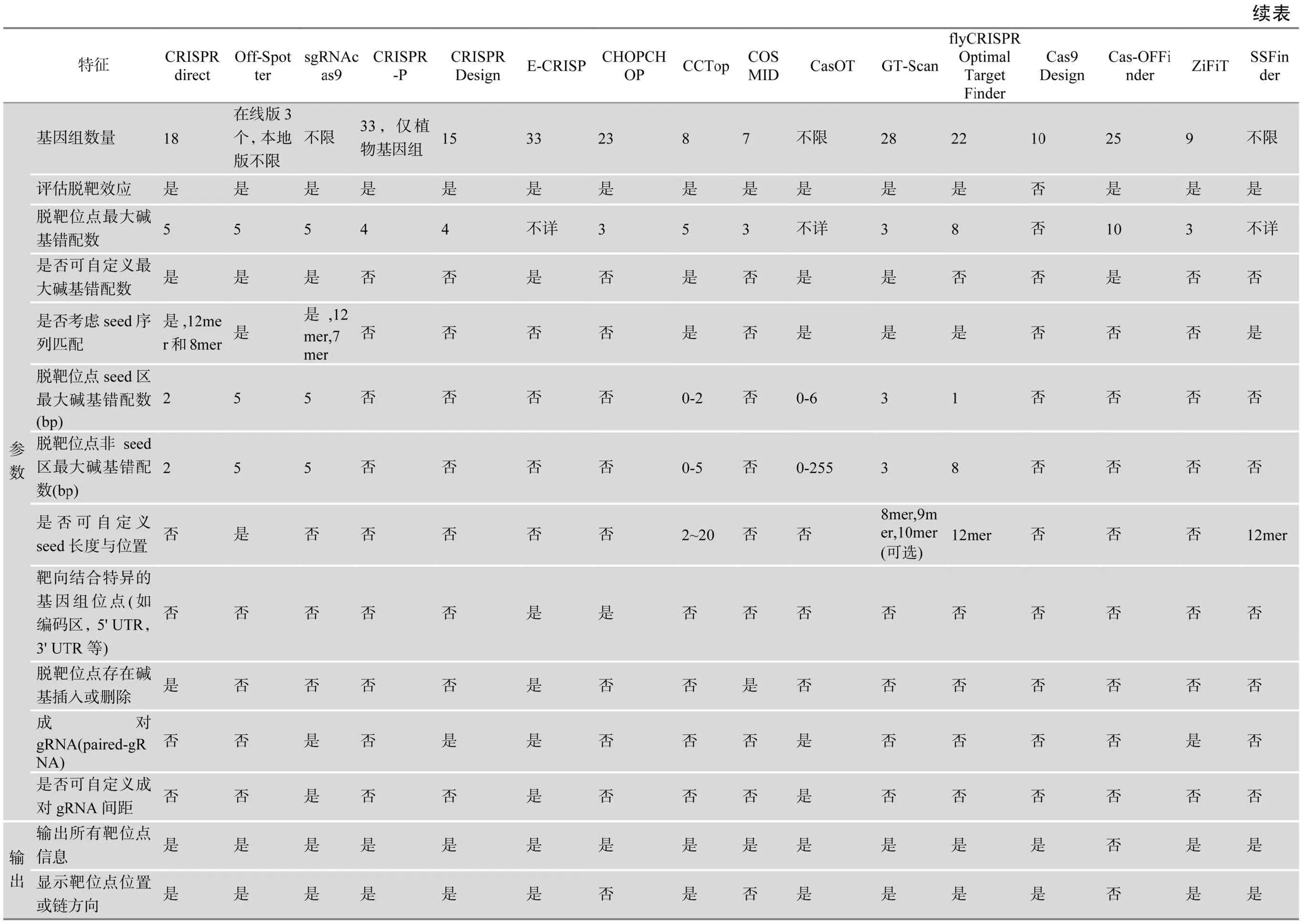
自2013年起,包括本实验室开发的sgRNAcas9软件在内,共有20多款在线和/或单机版sgRNA设计和脱靶效应评估软件陆续公布。按照操作平台、输入、参数设置、结果输出和其他共5项内容,通过制定38个评测指标,本文对其中16款软件的操作性能进行比较分析(附表1),这些软件的特点介绍如下。
2.1在线sgRNA设计和脱靶效应评估软件
2.1.1CRISPR Design
该软件[20]由美国麻省理工学院Broad研究所的张锋实验室开发(http://crispr.mit.edu/),有两种设计模式。单序列(Single sequence)模式仅可针对大小为23~500nt的序列设计sgRNA,且限定sgRNA的长度为20 bp;批量模式(Batch mode)允许上传FASTA格式的多序列文件,可同时针对多条序列设计sgRNA。对于脱靶效应评估,该软件仅包含人、小鼠和大鼠等15个物种基因组供脱靶效应评估,其考察脱靶位点碱基错配数,允许≤4个碱基错配;PAM类型除了“NGG”外,还有“NAG”,并进一步考察脱靶位点是否位于其他基因外显子等。软件将依据特定公式对每个脱靶位点评分,再采用脱靶位点结合反向似然法(Inverse likelihood of off target binding),计算每条sgRNA的总得分。最后,按照分数高低对sgRNA进行排序,并用红、绿和黄3种颜色标示sgRNA特异性高低。其中绿色表示特异性高,总得分大于50的sgRNA;黄色表示特异性为中等水平的sgRNA;而红色表示该sgRNA可能存在较高脱靶风险,应避免使用。
2.1.2ZiFiT
该软件[27]由锌指联盟(Zinc Finger Consortium)开发,最初用于设计ZFN(http://zifit.partners.org/ZiFiT/)。最新版ZiFiT(V4.2)可设计并评估sgRNA的脱靶效应,针对不同类型的Cas9核酸酶有3种设计模式:(1)设计针对野生型Cas9核酸酶的sgRNA(CRISPR/ Cas Nucleases);(2)设计针对Cas9单切口酶的“paired-gRNA”(CRISPR/Cas nickase);(3)设计针对CRISPR RFNs(RNA-guided Fok I nucleases)的“paired-gRNA”。对于第一种模式,可设置靶标结合位点的长度(Length of target site),可选长度范围为2~20nt。由于U6和T7是RNA聚合酶Ⅲ型启动子,它们的转录起始依赖碱基“G”。因此,可限定sgRNA的5'端碱基组成以构建sgRNA表达载体。对于设计针对野生型Cas9核酸酶的sgRNA,仅需输入FASTA格式序列,设置完参数后,点击“鉴定靶标结合位点(Identify target sites)”按钮,即会输出所有sgRNA,再点击“鉴定潜在的脱靶位点(Identify potential offtargets)”按钮,软件会对每条sgRNA进行全基因组水平脱靶分析。该软件目前仅含人、小鼠和大鼠等9个物种基因组供分析,脱靶位点最大允许3个碱基错配。而对于后2种设计模式,软件仅设计“paired-gRNA”,并直接输出用于构建特定表达载体的引物。对于设计针对“RNA-guided Fok Ⅰ Nucleases”和Cas9 单切口酶的“paired-gRNA”,两者差异在于是否限定“paired-gRNA”之间的距离。对“RNA-guided Fok Ⅰ nucleases ”而言,研究表明,当“paired-gRNA”间距为16 bp时,基因组编辑效率最高。因此,该软件主要设计距离接近16 bp的“paired-gRNA”;而针对Cas9单切口酶,并未限定“paired-gRNA”之间的距离。
2.1.3Cas9 Design
该软件[28]由北京大学工学院研究人员开发,仅用于对小鼠、大鼠和斑马鱼等10种模式动物设计sgRNA(http://cas9.cbi.pku.edu.cn/)。sgRNA长度可选范围为20~30 nt。此外,该软件还考察全长sgRNA二级结构对转录效率的影响,允许设置除20nt长度的靶标识别序列之外,与Cas9核酸酶结合的“gRNAscaffod”和转录终止“TTTT”序列。该软件操作界面简洁,输入FASTA序列和设置完参数后,点击“设计(Design)”按钮即可运行。对于每条sgRNA,软件会评估其是否为基因组上唯一存在的序列。其次,检测靶标位点是否含SNPs;该软件主要调用“Vienna RNAFold”程序,对sgRNA的二级结构进行预测,并计算有几个碱基位于茎上,以此来评估sgRNA的转录效率。
2.1.4E-CRISP
该软件[29]可设计并评估sgRNA的脱靶效应(http://www.e-crisp.org/E-CRISP/index.html),分为从头设计(De-novo)和脱靶效应评估(Evaluation)两种模式,含人、小鼠和猪等多达33个物种的基因组。对于sgRNA从头设计模式,可输入FASTA格式靶标序列(Enter target sequence),也可仅输入基因名称(Gene symbol)。该软件除了设计单个sgRNA,也可设计“paired-gRNA”。在起始应用(Start application)选项,有3种模式可供选择:(1)严谨型,限定PAM类型仅为NGG,sgRNA的5'端碱基仅为G,脱靶位点允许多个碱基错配,并排除位于内含子、CpG 岛(CpG islands)与UTRs(Untranslated regions)的脱靶位点;(2)中等型,PAM类型为NAG/NGG,不限sgRNA的5'端碱基组成,脱靶位点允许多个碱基错配,排除位于内含子和CpG岛的脱靶位点;(3)松散型,PAM类型为NAG/NGG,不限sgRNA的5'端碱基组成,脱靶位点完全匹配,包含内含子。在设置设计目的(Design purpose)选项,可依据特定的实验目的,确保sgRNA与目标基因特定位点结合,比如基因敲低或敲除实验(Knock-down/out)、N端标记(N-terminal tagging)、C端标记(C-terminal tagging)和“CRISPR double nicking”,这几种方法可限定sgRNA靶标的位置。该软件的另一特点是,可设置sgRNA的长度(7~30 bp),sgRNA的A、T、C和G不同碱基比例、PAM类型和靶标位点在基因组上的位置等。另外,对于设计“paired-gRNA”,两个sgRNA之间的距离可选范围为-8~25 bp。设置脱靶效应分析(Off-target analysis)选项,可选择使用哪种版本的基因组比对工具(bowtie程序),还可设置参考数据库,如基因组数据库、转录组数据库或不包含内含子的基因组序列,以及选择是否分析脱靶结合位点的二级结构等。对于结果输出,在基因注释排除(Gene annotation filtering)选项,可依据靶标位点在基因组上的位置,设置是否排除该sgRNA序列,如不显示位于非编码区、外显子区或CpG岛的sgRNA等。在输出(Outputs)选项,还可设置每个外显子最大设计几条sgRNA,是否输出为GFF(General feature format)格式的文件,是否生成基因组定位信息模式图等。设置完参数后,点击“开始搜寻sgRNA(Start sgRNA search)”按钮,程序即可运行,结果将以网页的形式输出。对于结果输出,软件会显示目标序列中共有多少条sgRNA、符合参数条件的有多少条、排除了多少条等信息。所有结果均以表格形式呈现,并依据评分高低对sgRNA排序。
2.1.5Cas-OFFinder
该软件[30]仅可评估sgRNA的脱靶效应(http:// www.rgenome.net/cas-offinder/),由韩国首尔国立大学(Seoul National University)Jin-Soo Kim教授实验室开发。除了在线软件外,还提供单机版。该在线软件含人、小鼠和大鼠等25个物种基因组,适用范围较广。可针对不同物种来源的Cas9核酸酶,评估sgRNA的脱靶效应。例如,来自化脓性链球菌(Streptococcus pyogenes)的“SpCas9”核酸酶,其PAM类型为“5'-NGG-3'”或“5'-NRG-3' (R = A或G)”;来自嗜热链球菌(Streptococcus thermophilus)的“StCas9”核酸酶,其PAM类型为“5'-NNAGAAW-3' (W = A或T)”;来自脑膜炎奈瑟氏菌(Neisseria meningitidis)的“Nm-Cas9”核酸酶,其PAM类型为“5'-NNNNGMTT-3' (M = A或 C)”;来自金黄色葡萄球菌(Staphylococcus aureus)的“SaCas9”,其PAM类型为“5'-NNGRRT-3' (R=A或G)”。对不同的Cas9核酸酶,除了PAM类型不同外,对应的sgRNA长度也不同,如针对“SpCas9”核酸酶,要求sgRNA长度为20 bp,针对“StCas9”核酸酶,要求sgRNA长度为18 bp,而针对“NmCas9”核酸酶,要求sgRNA长度为24 bp。另外,还允许使用简并碱基设置PAM类型,如R代表A或G,Y代表C或T,且脱靶位点允许碱基错配范围是0~10 bp。该软件运行速度较快,结果直接在页面输出,除了显示脱靶位点外,还标示染色体位置和碱基错配数等。
2.1.6CRISPR-P
该软件[31]由华中农业大学作物遗传改良国家重点实验室陈玲玲课题组开发(http://cbi.hzau.edu.cn/ crispr/),包含拟南芥、水稻和玉米等33个植物基因组。有3种sgRNA设计方式,如直接输入位点标签(Locus tag)、基因组位置(Position)或基因序列(Sequence)。软件允许输入的序列范围为23~5000 bp。设置完参数后,点击“提交(Submit)”按钮,将输出碱基为“G(N)20GG” 或 “A(N)20GG”的靶标位点。针对构建由 U6或U3启动子驱动的表达载体,可限定sgRNA的5'碱基为G或A。对于脱靶效应评估,该软件调用“BLASTn”程序进行全基因组比对分析,且脱靶位点最大允许4个碱基错配。结果输出中还会标示sgRNA在染色体上的位置,并依据特定公式对sgRNA进行评分并排序。其脱靶评分公式与上述“CRISPR Design”软件相同,但仅针对植物进行sgRNA设计。最大特色是搜寻sgRNA靶标位点中是否含限制性内切酶识别序列,以方便采用酶切法检测基因组切割效率。
2.1.7CHOPCHOP
该软件[32]可同时用于CRISPR/Cas9或TALEN系统介导的基因组定点编辑技术(https://chopchop. rc.fas.harvard.edu/index.php)。对于靶标基因选择,可输入基因名称、基因组位置或碱基序列,其包含有果蝇、人和小鼠等23个物种基因组。如果切换到高级设置选项(Toggle advanced options),可限定sgRNA与基因组特定区域结合(Target specific regionof gene),如基因编码区、外显子、选择性剪切位点、5' UTR和3' UTR等。在限制靶标(Restrict targeting)选项,可设置仅搜寻外显子及相邻的侧翼序列,或选择设计针对外显子的sgRNA;在限定靶标结合位点限制性内切酶(Restriction enzymes)类型选项,可设置限制性内切酶的来源与结合位点数目,如限定仅针对来自NEB或Sigma等公司的限制性内切酶等。对于脱靶效应评估,可切换到确定脱靶位点在基因组的方法(Method for determining off-targets in the genome)选项,其有3种脱靶效应评估规则供选择:(1)与脱靶位点结合的20 bp序列中含大于2个错配碱基;(2)仅与脱靶位点结合的15 bp种子序列完全匹配;(3)与脱靶结合位点序列完全匹配。对于限定sgRNA 的5'碱基组成(5'requirements for gRNA)选项,也有3种类型的碱基组合:(1)GN或NG;(2)GG;(3)不限。而对于PAM类型(PAM motif)选项,同样有3种不同的碱基组合:(1)NGG;(2)NNAGAA;(3)NNNNGANN。选择“CRISPR/Cas9”模式,设置完参数后,点击“寻找靶标结合位点( Find Target Sites!)”按钮,软件即可运行。结果输出会将sgRNA标示到目标基因组的染色体上,并以不同颜色标示其位置,如外显子和内含子等。该软件除了考察靶标位点是否含特定限制性内切酶识别序列外,还有一个特色是每条sgRNA均会设计数条用于检测基因组编辑效率的PCR扩增引物。而PCR扩增产物的大小、引物长度、溶解温度(Tm)和距离靶标结合位点的最小距离等均可自定义。
2.1.8GT-Scan
该软件[33]可用于设计特异性的sgRNA,其包含人、小鼠和斑马鱼等 28个物种基因组供脱靶评估(http://gt-scan.braembl.org.au/gt-scan/submit)。在基本参数设置选项中,可输入或上传FASTA格式的文件,最大支持4 kb的序列,并且有5种sgRNA设计规则供选择(Enter the target rule):(1)sgRNA和PAM一起长度为23nt,其中种子序列含8个碱基,脱靶位点的PAM类型为“NGG”或“NAG”;(2)sgRNA和PAM一起长度为23nt,其中种子序列含9个碱基,脱靶位点的PAM类型为“NGG”或“NAG”;(3)sgRNA和PAM一起长度为23nt,其中种子序列含10个碱基,脱靶位点的PAM类型为“NGG”或“NAG”(默认参数);(4)sgRNA和PAM一起长度为23 nt,如果构建T7或U6启动子驱动的sgRNA表达载体,其5'碱基为G,且种子序列含10个碱基,脱靶位点的PAM类型为“NGG”或“NAG”;(5)sgRNA和PAM一起长度为20nt,其中种子序列仅含5个碱基,脱靶位点的PAM类型仅为“NGG”。在高级参数(Advanced parameters)选项,可通过脱靶位点过滤规则(Enter the off-target filter)选项设置PAM的类型,并可用简并碱基,设置不同类型的PAM,因此适用于针对多种类型的Cas9核酸酶设计sgRNA。在限定特异性错配(Select the high-specificity mismatch limit)选项,可设置脱靶位点含0~3 bp的碱基错配。设置完参数后,点击“扫描(Scan)”按钮,即可快速设计sgRNA并进行脱靶预测。
2.1.9flyCRISPR Optimal Target Finder
该软件[34]适用于为不同品系果蝇、蜜蜂和线虫等22个物种设计特异性的sgRNA(http://tools.flycrispr.molbio.wisc.edu/targetFinder/)。有2种设计模式,其一是可针对序列设计sgRNA并评估脱靶效应,其次是预测已知sgRNA的脱靶效应。允许输入的sgRNA范围为16~20 nt,此外,可限定sgRNA的5'碱基组成,如仅设计5'碱基为G或GG的sgRNA等。设置完参数后,点击“寻找CRISPR靶标(Find CRISPR Targets)”即可运行。如果仅评估已知sgRNA的脱靶效应,可点击“跳到下一步(Skip to next step)”按钮,将进入新的设计页面,在CRISPR靶标(CRISPR targets)选项框中输入sgRNA,在设置脱靶效应严谨性(Stringency)选项,有2种模式供选择:(1)高严谨性,限定种子序列完全匹配,或种子序列含1个碱基错配,非种子序列含少于2个碱基错配;(2)中等严谨性,限定种子序列完全匹配,或种子序列含1个碱基错配,非种子序列含少于5个碱基错配。或种子序列含2个碱基错配,非种子序列含少于2个碱基错配。而PAM可设置为“NGG”,或“NAG”和“NGG”。对于脱靶预测,该软件调用“TagScan Genome-wide Tag Scanner”程序,特点是分析12 bp种子序列的特异性。
2.1.10CRISPRdirect
该软件[35]可设计特异性的sgRNA,其提供有人、小鼠、大鼠和猪等18个物种的基因组(http://crispr. dbcls.jp/)。对于靶基因选择较为灵活,不仅支持直接输入基因登录号或基因组位点,还可输入FASTA格式序列或上传文件。PAM类型默认为NGG,但可通过简并碱基设置其它类型。对于评估脱靶效应,综合考虑了全长和种子序列的特异性,主要有3种规则:(1)20 bp全长序列和PAM(20mer+PAM);(2)12 bp种子序列和PAM(12mer+PAM);(3)8 bp种子序列和PAM(8mer+PAM)。软件最大允许脱靶位点含2个碱基错配,还考察脱靶位点是否有 “gap”。设置完参数后,点击“设计(Design)”按钮即可运行。结果输出中除了计算sgRNA的GC含量外,还会将含有转录终止序列(“TTTT”)的sgRNA标出。
2.1.11COSMID
此软件[36]仅能评估sgRNA的特异性,提供有人、小鼠和大鼠等7个物种的基因组供分析(https://crispr. bme.gatech.edu/crispr/)。该软件设计的sgRNA范围为10~55nt。PAM类型可定义为“NGG”、“NAG”或“NRG”。最大特点是评估脱靶位点中是否有碱基插入和缺失。可对脱靶位点接受的“indels”和错配碱基数(Allowed indels and mismatch)进行设置,分别为:(1)不考察碱基插入与缺失(No indels),脱靶位点碱基数可自定义,且最大允许3个碱基错配;(2)单碱基缺失(1-base Del),脱靶位点碱基数可选,且最大允许2个碱基错配;(3)单碱基插入(1-base Ins),脱靶位点碱基数可选,且最大允许2个碱基错配。软件还可针对不同测序平台设计PCR扩增引物,如“Illumina_250”,“Illumina_250_paired”和“SMRT”等测序技术,用于高通量测序验证脱靶。
2.1.12Off-Spotter
此软件[37]能设计和评估sgRNA的脱靶效应,除了在线软件外,还提供单机版(https://cm.jefferson. edu/Off-Spotter/)。在线软件仅提供人,小鼠和酵母3个物种的基因组供分析。允许输入的序列长度不大于500nt;而对于脱靶分析,可同时对20条sgRNA进行脱靶评估。有4种类型的PAM,如NGG、NAG、NNNNACA 和NNGRRT (R代表A或G)。在设置脱靶位点的最大碱基错配数(Maximum number of mismatches)选项,可选范围为0~5 bp。而在选择种子序列位置(Select the positions that you want fixed ("seed"))选项,有3种类型供选择:(1)全部选择;(2)不选择;(3)选择默认,即仅包含sgRNA的3'端的5个碱基,此外还可自定义种子序列的长度,这也是此软件的特色之一。其次,软件会判断脱靶位点是否位于基因组特定位置,如未剪切的mRNA前体(pre-mRNA)、5'UTR、CDS或3'UTR、长链非编码RNA(lincRNA)等。参数设置完成后,点击“提交(submit)”按钮即可运行软件,输出的结果可读性强。
2.1.13CCTop
此软件[38]可用于设计和评估sgRNA的特异性,仅包含拟南芥、人和斑马鱼等8个物种的基因组供分析(http://crispr.cos.uni-heidelberg.de/index.html),且最大可为500 bp的序列设计长度为20nt的sgRNA。对于设计sgRNA,可选择不同PAM类型(PAM type(targeting)),如NGG或NRG,其中R代表 A 或G,还可限制sgRNA的5'和3'端碱基,在限定靶标5'碱基(Target site 5'limitation)选项,可选的类型有NN、NG和GG。而在限定靶标3'碱基(Target site 3'limitation)选项,可选的类型有:NN和GG。对于评估sgRNA的脱靶效应,也可设置不同PAM类型(PAM type(evaluation)),如NGG或NRG。此外,可设置sgRNA的核心结合位点的长度(core length),即种子序列可选范围是2~20 nt。对于核心结合位点最大错配碱基数(max. core mismatches),可选范围是0~2 bp,而对于全长结合位点的脱靶位点,允许的碱基错配数(max. total mismatches)最大为5 bp。设置完参数后,点击“提交(submit)”按钮即可运行软件。输出的结果中会标示sgRNA在染色体上的相对位置。
2.2单机版软件
2.2.1CasOT
该软件[39]可用于设计和评估sgRNA的脱靶效应,由北京大学生命科学学院张博教授和高歌教授实验室联合开发(http://eendb.zfgenetics.org/casot/index. php)。此软件用Perl语言编写,使用前,需首先在电脑上安装Perl(http://www.perl.org/get.html)程序,通过命令行调用。该软件可用于评估已知单个sgRNA或“paired-gRNA”的脱靶效应,sgRNA的长度范围为18~30nt。或用于设计sgRNA,允许输入的序列长度不大于1 kb。有4种PAM类型供选择:(1)NGG (默认);(2)NGG 和 NAG;(3)NGG、NAG 和NNGG;(4)不限。还可限定sgRNA的5'碱基组成是否为G。对于预测脱靶效应,其将sgRNA划分为种子区和非种子区。对于脱靶位点的种子区,即临近PAM的12个核心序列,最大允许含6个碱基错配。而对于脱靶位点的非种子区,错配碱基可任意设置。对于不同物种FASTA格式的基因组和基因注释文件,可从ensembl在线网站的ftp数据库下载(http://www.ensembl.org/info/data/ftp/index.html)。
2.2.2sgRNAcas9
该软件[40]由本实验室与上海科技大学的黄行许教授实验室联合开发,能用于设计和评估sgRNA的脱靶效应(http://www.biootools.com/)。此软件包有8个功能不同的小程序,最新版本为V3.0.5,均由Perl语言编写,可方便在不同操作平台如Windows和linux等操作系统中使用,运行环境仅需提前安装Perl。使用前,需从ensembl的ftp数据库下载目标物种的基因组和基因注释文件。大致操作流程如下:(1)通过命令行调用“combine_genome.pl”程序,将下载的不同染色体序列合并为FASTA格式的单一基因组文件;(2)用“format_genome.pl”程序处理基因组文件;(3)调用核心程序“sgRNAcas9.pl”设计sgRNA并进行脱靶效应评估。可设置的参数有:①sgRNA的长度,限定范围为17~20nt;②sgRNA 的GC%含量,可限定最大值和最小值,默认为20%~80%;③对于靶标在DNA链上的位置,可限定sgRNA仅定位于正义链、反义链或双链;④既可设计单个sgRNA,还可设计“paired-gRNA”。如果设计“paired-gRNA”,可限定两个sgRNA之间距离最大值和最小值,默认值为-2~32 bp。靶标位点PAM类型为NGG,而对于脱靶评估,PAM类型为NGG和NAG。sgRNA与脱靶位点的碱基错配数可设置的范围为0~5 bp,还自动排除含“TTTT”终止序列的sgRNA。该软件对于脱靶效应评估,除了统计sgRNA全长序列碱基错配位点情况,还考察临近PAM的12 bp种子序列的特异性;(4) 利用从ensembl的ftp数据库下载的基因注释文件,可用“ot2gtf_v2.pl”和“pot2gtf_v2.pl”程序,进一步确定脱靶位点是否位于其它基因内;(5)利用“sgRPrimer.pl”程序,可批量设计用于构建sgRNA表达载体的引物;(6)利用“extract_targetSeq.pl”程序,可批量提取靶标位点或预测的脱靶位点两侧一定长度的基因组序列,以用于设计PCR扩增引物。
2.2.3SSFinder
该软件[41]可设计sgRNA并进行脱靶分析(https: //code.google.com/p/ssfinder/)。此软件用Python语言编写,运行环境需安装有Python(V2.2及以上版本)。PAM类型仅考虑“NGG”,脱靶位点分析考虑了12个碱基的种子序列特异性。设计的sgRNA有4种类型:(1) G/CN7S11G/C;(2) G/CN7S11A/T;(3) A/TN7S11A/T和(4) A/TN7S11G/C(N代表4种任意碱基,S代表种子序列),即限定5'碱基类型。
3 基因组编辑效率与脱靶检测方法及软件
3.1生物学实验方法
目前,用于检测CRISPR/Cas9技术基因组编辑效率和脱靶的实验方法较多。下面阐述几种主要方法的特点:
(1)限制性核酸内切酶法。该方法的特点是选择靶标位点含限制性内切酶识别位点的sgRNA。当靶标位点被切割后,相应的限制性内切酶识别序列会被破坏,由此可用对应的限制性内切酶,检测DNA靶标是否被切割[42]。或者在提供的同源供体DNA中,插入限制性核酸内切酶识别碱基序列,使其通过同源重组插入到靶标位点。再通过限制性内切酶进行酶切,以此方法来判断靶标位点是否发生同源重组[42]。
(2)Surveyor酶切法(Surveyor endonuclease I cleavage assays)。Surveyor酶是一种S1核酸内切酶,能特异性识别并切割异源双链中错配的碱基,电泳分离酶切产物,可根据条带大小判断是否有基因组切割。该方法最初用于检测ZFN和TALEN技术介导的基因组编辑,可检测靶标结合位点是否有碱基替换或“Indels”,目前有用于CRISPR/Cas9技术介导的基因组编辑的报道[23]。
(3)T7E1酶切法(T7 endonuclease I cleavage assays)。T7E1内切酶能识别并切割不完美匹配的DNA、十字形DNA结构、Holliday结构或DNA分叉点及异源二聚体DNA。T7E1酶切法是目前检测CRISPR/ Cas9技术介导的基因组编辑效率的常用方法之一[2~4]。与Surveyor酶切法类似,该酶识别错配的杂合双链,能根据电泳条带亮度强弱计算“Indels”频率。
(4)高分辨率熔解曲线法(High-resolution melting,HRM)。该方法的基本原理是依据DNA双链分子不同的溶解曲线来区分不同基因型的样品,且分辨率能达到单碱基水平。Bassett等[43]利用该方法,检测了利用CRISPR/Cas9技术基因敲除的果蝇。研究表明,该方法能较好区分嵌合体和杂合突变体果蝇,也适用于检测基因组编辑效率高低[43,44]。
(5)单分子实时(SMRT)测序法(Single molecule real time (SMRT™) DNA sequencing)。此为第三代高通量测序技术,由Pacific Biosciences公司开发。Hendel等[45]利用该方法,分别检测了ZFNs、TALENs和CRISPR/Cas9系统介导的基因组编辑效率,发现该方法可同时评估NHEJ和HR介导的基因突变或修复效率。
(6)中性聚丙烯酰胺凝胶电泳法。该技术的实验原理是,在中性聚丙烯酰胺凝胶中,不同构象等长的DNA单链电泳迁移率会发生变化,由此来判断基因是否发生突变。Zhu等[46]采用该方法,分析了利用CRISPR/Cas9技术制备的果蝇模型的基因型。研究发现,该技术能准确判定不同突变类型果蝇的基因型。
(7)SSA (Single-strand annealing)报告载体活性检测法。该方法的核心是使用SSA报告基因载体,该载体中Luciferase报告基因被终止密码子提前终止,这种截短的Luciferase蛋白没有活性。如将sgRNA的靶标位点置于终止密码子之后。若靶标位点发生基因组编辑,可通过同源重组形成有活性的Luciferase,再通过检测Luciferase活性高低,即可评估CRISPR/Cas9技术的基因组编辑效率[47]。
(8)Sanger测序法。该方法为检测CRISPR/Cas9技术基因组编辑效率常用方法之一[5]。可直接将PCR产物进行测序或连接到TA克隆载体中,再挑选单克隆菌落测序,该方法不仅能确定碱基缺失或插入类型,还可计算切割效率大小,但缺点是灵敏度较低。
对于基因组编辑效率和特异性评估,除了上述检测方法外,最近新开发有几种高通量和无偏评估方法:①整合酶缺陷型慢病毒载体(IDLVs)技术,其原理是细胞通过NHEJ机制对DNA修复的过程中,线性双链IDLV基因组能够优先整合到断裂的DNA双链处。该方法最初用于评估ZFN技术的基因组编辑效率[48]。2015年,Wang等[25]将此方法用于评估CRISPR/Cas9和TALENs技术的基因组编辑效率。研究表明,在靶标结合位点60 bp范围内,可发现成簇的IDLV整合位点。该技术仅能检测到频率低至1%的脱靶突变,灵敏度较低,但却是从全基因组水平无偏检测脱靶效应。②GUIDE-seq(Genome-wide unbiased identifications of DSBs evaluated by sequencing)技术,该方法的原理是让细胞吸收特定的双链寡核苷酸(Double stranded oligodeoxynucleotides, dsODN)并整合进基因组断裂位点。然后抽提基因组DNA,随机打断后修复末端,连接用于高通量测序的接头。然后对dsODN的特异序列进行PCR扩增,富集包含dsODN的片段并进行高通量测序,最后利用软件分析Cas9核酸酶的切割位点,由此评估脱靶效应[49]。研究显示,该方法的灵敏度较高,能检测到频率低至0.1%的脱靶突变[39]。③Digenomeseq(Cas9 nuclease-digested whole genome sequencing)技术,其原理是利用全基因组测序法,寻找CRISPR/ Cas9技术介导的基因组打靶和脱靶位点。大致流程是用Cas9核酸酶在试管中消化细胞的基因组DNA,然后通过高通量方法进行全基因组测序。这种体外消化可使得打靶和脱靶位点产生独特的序列模式,通过软件即可计算基因组编辑效率。研究表明,此方法灵敏度同样较高,可检测插入或缺失频率为0.1%的脱靶突变[50]。
3.2基因组编辑效率评估软件
针对上述检测方法,开发相关软件用于基因组切割效率定量分析,如CRISPR-GA(CRISPR Genome Analyzer, http://54.80.152.219/)[51]。该软件针对Illumina Miseq高通量测序平台,分析通过高通量测序技术获得的数据,以此评估碱基敲除和插入效率。其分析流程是:Miseq reads质量控制、定位、检测碱基插入/缺失(Indel calling)、计算HR 和NHEJ和结果输出。该软件操作简单,仅需输入上下游测序数据和靶标位点序列即可。其次是TIDE在线软件[52],由R语言编写(http://tide.nki.nl/)。该软件可针对Sanger测序法,分析色谱图(Chromatogram)来计算碱基插入和缺失频率,可用于计算基因组编辑效率和脱靶。
4 问题与展望
利用CRISPR/Cas9技术关键的两个问题是:如何提高CRISPR/Cas9技术基因组编辑效率?如何最大限度降低脱靶风险?综上所述,影响基因组编辑效率和特异性的因素很多。目前业界对CRISPR/Cas9系统介导的基因组编辑效率和脱靶效应研究尚不够深入,观点还不统一。这反映在不同sgRNA设计与脱靶预测软件中,对sgRNA的活性和特异性评估标准不一致。比如“CRISPR Design”软件对脱靶位点最大仅考虑4个碱基错配,未考虑种子序列的特异性。而sgRNAcas9软件允许脱靶位点最大含 5个碱基错配,还考察了长度为12 bp的种子序列。CRISPRdirect和COSMID软件将脱靶位点是否存在碱基插入与缺失纳入评估范围,Off-Spotter软件允许自定义种子序列位置和长度。E-CRISP软件还评估sgRNA与脱靶位点的二级结构及基因组位置等。目前很多软件并未对预测的脱靶位点进行验证,因而无法比较不同软件评估脱靶效应算法灵敏度高低。
sgRNA和PAM类型影响基因组编辑效率和特异性,而不同sgRNA活性和特异性有差异。因此,选择sgRNA需慎重。选择高效且特异的sgRNA需要注意的因素有:(1)sgRNA的长度为17~20nt;(2)可选3'末端含GG的sgRNA,避免使用含“TTTT”转录终止序列的sgRNA,GC%含量为40%~60%;(3)如果构建U6或T7启动子驱动的sgRNA表达载体,为提高转录效率,可限定sgRNA的5'末端为G或GG;(4)如需造成基因移码突变,尽量选择在功能域或编码区内部设计sgRNA;(5)检查sgRNA靶标位点基因组序列是否存在SNPs;(6)针对Cas9单切口酶设计“paired-gRNA”,需限定两个sgRNA之间的距离,建议为-2~32nt;(7)分析脱靶位点可限定最大允许5个碱基错配,并同时评估种子序列的特异性。另外还可评估是否存在碱基插入或缺失。总之,应选择合适的软件设计sgRNA并进行脱靶分析。一个基因建议选择2~3个sgRNA进行实验验证,再选择活性高的sgRNA开展下一步的功能实验。
在对上述资料分类整理的基础上,本实验室建立了一个CRISPR-Cas9技术资源信息网(http://www.biootools.com/cn/),以方便广大科研工作者使用。与其他基因组编辑技术相比,CRISPR/Cas9技术的特点是操作简单且功能强大。因此,该技术不仅在模式动物基因功能研究领域得到广泛应用,也在农业经济动物(如猪)的遗传育种领域得到开发,并展现出广阔的应用前景。Hai等[6]率先利用CRISPR/ Cas9技术,结合受精卵显微注射法获得了vWF基因遗传修饰猪。Sato等[7]利用该技术成功敲除猪胚胎成纤维细胞的α-1, 3-半乳糖基转移酶基因。Whitworth等[8]利用该技术,结合体细胞核移植技术或受精卵显微注射方法,获得了CD163 和CD1D基因敲除猪。Zhou等[9]利用此技术,同样结合体细胞核移植技术,成功构建了酪氨酸酶基因和PARK2与PINK1双基因敲除小型猪,并且分别建立了人类白化病和帕金森综合征猪模型。尽管CRISPR/Cas9技术目前存在编辑效率低且具有一定的脱靶风险,但随着研究的深入,这些问题势必会得到解决,即可推进功能基因组学和分子育种领域的发展。
附录:附表见文章电子版:www.chinagene.cn。
[1] Brouns SJJ, Jore MM, Lundgren M, Westra ER, Slijkhuis RJH, Snijders APL, Dickman MJ, Makarova KS, Koonin EV, van der Oost J. Small CRISPR RNAs guide antiviral defense in prokaryotes. Science, 2008, 321(5891): 960-964.
[2] Niu YY, Shen B, Cui YQ, Chen YC, Wang JY, Wang L,Kang Y, Zhao XY, Si W, Li W, Xiang AP, Zhou JK, Guo XJ, Bi Y, Si CY, Hu B, Dong GY, Wang H, Zhou ZM, Li TQ, Tan T, Pu XQ, Wang F, Ji SH, Zhou Q, Huang XX, Ji WZ, Sha JH. Generation of gene-modified cynomolgus monkey via Cas9/RNA-mediated gene targeting in one-cell embryos. Cell, 2014, 156(4): 836-843.
[3] Zhou JK, Shen B, Zhang WS, Wang JY, Yang J, Chen L,Zhang N, Zhu K, Xu J, Hu B, Leng QB, Huang XX. One-step generation of different immunodeficient mice with multiple gene modifications by CRISPR/Cas9 mediated genome engineering. Int J Biochem Cell Biol, 2014,46: 49-55.
[4] Ma YW, Zhang X, Shen B, Lu YD, Chen W, Ma J, Bai L,Huang XX, Zhang LF. Generating rats with conditional alleles using CRISPR/Cas9. Cell Res, 2014, 24(1): 122-125.
[5] Chang NN, Sun CH, Gao L, Zhu D, Xu XF, Zhu XJ, Xiong JW, Xi JJ. Genome editing with RNA-guided Cas9 nuclease in zebrafish embryos. Cell Res, 2013, 23(4): 465-472.
[6] Hai T, Teng F, Guo RF, Li W, Zhou Q. One-step generation of knockout pigs by zygote injection of CRISPR/Cas system. Cell Res, 2014, 24(3): 372-375.
[7] Sato M, Miyoshi K, Nagao Y, Nishi Y, Ohtsuka M, Nakamura S, Sakurai T, Watanabe S. The combinational use of CRISPR/Cas9-based gene editing and targeted toxin technology enables efficientbiallelic knockout of the α-1,3-galactosyltransferase gene in porcine embryonic fibroblasts. Xenotransplantation, 2014, 21(3): 291-300.
[8] Whitworth KM, Lee K, Benne JA, Beaton BP, Spate LD,Murphy SL, Samuel MS, Mao JD, O'Gorman C, Walters EM, Murphy CN, Driver J, Mileham A, McLaren D, Wells KD, Prather RS. Use of the CRISPR/Cas9 system to produce genetically engineered pigs from in vitro-derived oocytes and embryos. Biol Reprod, 2014, 91(3): 78.
[9] Zhou XQ, Xin JG, Fan NN, Zou QJ, Huang J, Ouyang Z,Zhao Y, Zhao BT, Liu ZM, Lai SS, Yi XL, Guo L, Esteban MA, Zeng YZ, Yang HQ, Lai LX. Generation of CRISPR/Cas9-mediated gene-targeted pigs via somatic cell nuclear transfer. Cell Mol Life Sci,2015, 72(6): 1175-1184.
[10] Jiang WZ, Zhou HB, Bi HH, Fromm M, Yang B, Weeks DP. Demonstration of CRISPR/Cas9/sgRNA-mediated targeted gene modification in Arabidopsis, tobacco, sorghum and rice. Nucleic Acids Res, 2013, 41(20): e188.
[11] Nishimasu H, Ran FA, Hsu PD, Konermann S, Shehata SI,Dohmae N, Ishitani R, Zhang F, Nureki O. Crystal structure of Cas9 in complex with guide RNA and target DNA. Cell, 2014, 156(5): 935-949.
[12] Barrangou R. RNA events. Cas9 targeting and the CRISPR revolution. Science, 2014, 344(6185): 707-708.
[13] Konermann S, Brigham MD, Trevino AE, Joung J, Abudayyeh OO, Barcena C, Hsu PD, Habib N, Gootenberg JS, Nishimasu H, Nureki O, Zhang F. Genome-scale transcriptional activation by an engineered CRISPR-Cas9 complex. Nature, 2015, 517(7536): 583-588.
[14] Hruscha A, Krawitz P, Rechenberg A, Heinrich V, Hecht J,Haass C, Schmid B. Efficient CRISPR/Cas9 genome editing with low off-target effects in zebrafish. Development,2013, 140(24): 4982-4987.
[15] Doench JG, Hartenian E, Graham DB, Tothova Z, Hegde M, Smith I, Sullender M, Ebert BL, Xavier RJ, Root DE. Rational design of highly active sgRNAs for CRISPRCas9-mediated gene inactivation. Nat Biotechnol, 2014,32(12): 1262-1267.
[16] Zhang YL, Ge XL, Yang FY, Zhang LP, Zheng JY, Tan XF, Jin ZB, Qu J, Gu F. Comparison of non-canonical PAMs for CRISPR/Cas9-mediated DNA cleavage in human cells. Sci Rep, 2014, 4: 5405.
[17] Farboud B, Meyer BJ. Dramatic Enhancement of Genome Editing by CRISPR/Cas9 Through Improved Guide RNA Design. Genetics, 2015, 199(4): 959-971.
[18] Yu C, Liu YX, Ma TH, Liu K, Xu SH, Zhang Y, Liu HL,La Russa M, Xie M, Ding S, Qi LS. Small Molecules Enhance CRISPR Genome Editing in Pluripotent Stem Cells. Cell Stem Cell, 2015, 16(2): 142-147.
[19] Fu YF, Foden JA, Khayter C, Maeder ML, Reyon D,Joung JK, Sander JD. High-frequency off-target mutagenesis induced by CRISPR-Cas nucleases in human cells. Nat Biotechnol, 2013, 31(9): 822-826.
[20] Hsu PD, Scott DA, Weinstein JA, Ran FA, Konermann S,Agarwala V, Li YQ, Fine EJ, Wu XB, Shalem O, Cradick TJ, Marraffini LA, Bao G, Zhang F. DNA targeting specificity of RNA-guided Cas9 nucleases. Nat Biotechnol,2013, 31(9): 827-832.
[21] Pattanayak V, Lin S, Guilinger JP, Ma EB, Doudna JA,Liu DR. High-throughput profiling of off-target DNA cleavage reveals RNA-programmed Cas9 nuclease specificity. Nat Biotechnol, 2013, 31(9): 839-843.
[22] Zhang JH, Pandey M, Kahler JF, Loshakov A, Harris B,Dagur PK, Mo YY, Simonds WF. Improving the specificity and efficacy of CRISPR/CAS9 and gRNA through target specific DNA reporter. J Biotechnol, 2014, 189: 1-8.
[23] Ran FA, Hsu PD, Lin CY, Gootenberg JS, Konermann S,Trevino AE, Scott DA, Inoue A, Matoba S, Zhang Y,Zhang F. Double nicking by RNA-guided CRISPR Cas9 for enhanced genome editing specificity. Cell, 2013,154(6): 1380-1389.
[24] Lin YN, Cradick TJ, Brown MT, Deshmukh H, Ranjan P,Sarode N, Wile BM, Vertino PM, Stewart FJ, Bao G. CRISPR/Cas9 systems have off-target activity with insertions or deletions between target DNA and guide RNA sequences. Nucleic Acids Res, 2014, 42(11): 7473-7485.
[25] Wang XL, Wang YB, Wu XW, Wang JH, Wang YJ, Qiu ZJ, Chang T, Huang H, Lin RJ, Yee JK. Unbiased detection of off-target cleavage by CRISPR-Cas9 and TALENs using integrase-defective lentiviralvectors. Nat Biotechnol,2015, 33(2): 175-178.
[26] Fu YF, Sander JD, Reyon D, Cascio VM, Joung JK. Improving CRISPR-Cas nuclease specificity using truncated guide RNAs. Nat Biotechnol, 2014, 32(3): 279-284.
[27] Sander JD, Zaback P, Joung JK, Voytas DF, Dobbs D. Zinc Finger Targeter (ZiFiT): an engineered zinc finger/target site design tool. Nucleic Acids Res, 2007,35(Web Server issue): W599-W605.
[28] Ma M, Ye AY, Zheng WG, Kong L. A guide RNA sequence design platform for the CRISPR/Cas9 system for model organism genomes. Biomed Res Int, 2013, 2013:270805.
[29] Heigwer F, Kerr G, Boutros M. E-CRISP: fast CRISPR target site identification. Nat Methods, 2014, 11(2): 122-123.
[30] Bae S, Park J, Kim JS. Cas-OFFinder: a fast and versatile algorithm that searches for potential off-target sites of Cas9 RNA-guided endonucleases. Bioinformatics, 2014,30(10): 1473-1475.
[31] Lei Y, Lu L, Liu HY, Li S, Xing F, Chen LL. CRISPR-P: aweb tool for synthetic single-guide RNA design of CRISPR-system in plants. Mol Plant, 2014, 7(9): 1494-1496.
[32] Montague TG, Cruz JM, Gagnon JA, Church GM, Valen E. CHOPCHOP: a CRISPR/Cas9 and TALEN web tool for genome editing. Nucleic Acids Res, 2014, 42(Web Server issue): W401-W407.
[33] O'Brien A, Bailey TL. GT-Scan: identifying unique genomic targets. Bioinformatics, 2014, 30(18): 2673-2675.
[34] Gratz SJ, Ukken FP, Rubinstein CD, Thiede G, Donohue LK, Cummings AM, O'Connor-Giles KM. Highly specific and efficient CRISPR/Cas9-catalyzed homology-directed repair in Drosophila. Genetics, 2014, 196(4): 961-971.
[35] Naito Y, Hino K, Bono H, Ui-Tei K. CRISPRdirect: software for designing CRISPR/Cas guide RNA with reduced off-target sites. Bioinformatics, 2015, 31(7): 1120-1123.
[36] Cradick TJ, Qiu P, Lee CM, Fine EJ, Bao G. COSMID: a web-based tool for identifying and validating CRISPR/Cas off-target sites. Mol Ther Nucleic Acids, 2014, 3(12): e214.
[37] Pliatsika V, Rigoutsos I. "Off-Spotter": very fast and exhaustive enumeration of genomic lookalikes for designing CRISPR/Cas guideRNAs. Biol Direct, 2015, 10(1): 4.
[38] Stemmer M, Thumberger T, Del Sol Keyer M, Wittbrodt J,Mateo JL. CCTop: an intuitive, flexible and reliable CRISPR/Cas9 target prediction tool. PLoS One, 2015,10(4): e0124633.
[39] Xiao A, Cheng ZC, Kong L, Zhu ZY, Lin S, Gao G, Zhang B. CasOT: a genome-wide Cas9/gRNA off-target searching tool. Bioinformatics, 2014, 30(8): 1180-1182.
[40] Xie SS, Shen B, Zhang CB, Huang XX, Zhang YL. sgRNAcas9: a software package for designing CRISPR sgRNA and evaluating potential off-target cleavage sites. PLoS One, 2014, 9(6): e100448.
[41] Upadhyay SK, Sharma S. SSFinder: high throughput CRISPR-Cas target sites prediction tool. Biomed Res Int,2014, 2014: 742482.
[42] Yang H, Wang HY, Shivalila CS, Cheng AW, Shi LY,Jaenisch R. One-step generation of mice carrying reporter and conditional alleles by CRISPR/Cas-mediated genome engineering. Cell, 2013, 154(6): 1370-1379.
[43] Bassett AR, Tibbit C, Ponting CP, Liu JL. Highly efficient targeted mutagenesis of Drosophila with the CRISPR/Cas9 system. Cell Rep, 2013, 4(1): 220-228.
[44] Ansai S, Kinoshita M. Targeted mutagenesis using CRISPR/Cas system in medaka. Biol Open, 2014, 3(5):362-371.
[45] Hendel A, Kildebeck EJ, Fine EJ, Clark JT, Punjya N,Sebastiano V, Bao G, Porteus MH. Quantifying genomeediting outcomes at endogenous loci with SMRT sequencing. Cell Rep, 2014, 7(1): 293-305.
[46] Zhu XX, Xu YJ, Yu SS, Lu L, Ding MQ, Cheng J, Song GX, Gao X, Yao LM, Fan DD, Meng S, Zhang XW, Hu SD, Tian Y. An efficient genotyping method for genome-modified animals and human cells generated with CRISPR/Cas9system. Sci Rep, 2014, 4: 6420.
[47] Cradick TJ, Antico CJ, Bao G. High-throughput cellular screening of engineered nuclease activity using the single-strand annealing assay and luciferase reporter. In: Storici F, ed. Gene Correction: Methods in Molecular Biology. Totowa: Humana Press, 2014, 1114: 339-352.
[48] Lombardo A, Genovese P, Beausejour CM, Colleoni S,Lee YL, Kim KA, Ando D, Urnov FD, Galli C, Gregory PD, Holmes MC, Naldini L. Gene editing in human stem cells using zinc finger nucleases and integrase-defective lentiviral vector delivery. Nat Biotechnol, 2007,25(11): 1298-1306.
[49] Tsai SQ, Zheng Z, Nguyen NT, Liebers M, Topkar VV,Thapar V, Wyvekens N, Khayter C, Iafrate AJ, Le LP,Aryee MJ, Joung JK. GUIDE-seq enables genome-wide profiling of off-target cleavage by CRISPR-Cas nucleases. Nat Biotechnol, 2015, 33(2): 187-197.
[50] Kim D, Bae S, Park J, Kim E, Kim S, Yu HR, Hwang J,Kim JI, Kim JS. Digenome-seq: genome-wide profiling of CRISPR-Cas9 off-target effects in human cells. Nat Methods, 2015, 12(3): 237-243.
[51] Güell M, Yang LH, Church GM. Genome editing assessment using CRISPR Genome Analyzer (CRISPR-GA). Bioinformatics, 2014, 30(20): 2968-2970.
[52] Brinkman EK, Chen T, Amendola M, van Steensel B. Easy quantitative assessment of genome editing by sequence trace decomposition. Nucleic Acids Res, 2014, 42(22): e168.
(责任编委: 张博)
sgRNA design for the CRISPR/Cas9 system and evaluation of its off-target effects
Shengsong Xie1, Yi Zhang2, Lisheng Zhang1, Guanglei Li1, Changzhi Zhao1,Pan Ni1, Shuhong Zhao1
1. Key Lab of Agricultural Animal Genetics, Breeding, and Reproduction of Ministry of Education, Huazhong Agricultural University, Wuhan 430070, China;
2. Department of Obstetrics and Gynecology, No.161 Hospital of PLA, Wuhan 430010, China
The third generation of CRISPR/Cas9-mediated genome editing technology has been successfully applied to genome modification of various species including animals, plants and microorganisms. How to improve the efficiency of CRISPR/Cas9 genome editing and reduce its off-target effects has been extensively explored in this field. Using sgRNA (Small guide RNA) with high efficiency and specificity is one of the critical factors for successful genome editing. Several software have been developed for sgRNA design and/or off-target evaluation, which have advantages and disadvantages respectively. In this review, we summarize characters of 16 kinds online and standalone software for sgRNA design and/or off-target evaluation and conduct a comparative analysis of these different kinds of software through developing 38 evaluation indexes. We also summarize 11 experimental approaches for testing genome editing efficiency and off-target effects as well as how to screen highly efficient and specific sgRNA.
CRISPR/Cas9 system; genome editing; sgRNA; off-target effects


2015-03-02;
2015-06-18
中央高校基本科研业务费专项资金资助项目(编号:2662015BQ005),广东省分子与细胞工程育种团队(编号:2011A020102003)和国家自然科学基金项目(编号:31301226)资助
谢胜松,副研究员,研究方向:动物功能基因组与疾病诊治。E-mail: ssxie@mail.hzau.edu.cn
赵书红,教授,博士生导师,研究方向:动物分子生物学与育种。E-mail: shzhao@mail.hzau.edu.cn
10.16288/j.yczz.15-093
网络出版时间: 2015-8-49:37:06
URL: http://www.cnki.net/kcms/detail/11.1913.R.20150804.0937.002.html

